+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11208 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GLIC pentameric ligand-gated ion channel, pH 5 | |||||||||
 マップデータ マップデータ | GLIC, pentameric ligand-gated ion channel, sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GLIC / pentameric ligand-gated ion channel / cryoem / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sodium channel activity / extracellular ligand-gated monoatomic ion channel activity / potassium channel activity / transmembrane transporter complex / regulation of membrane potential / transmembrane signaling receptor activity / neuron projection / signal transduction / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) | |||||||||
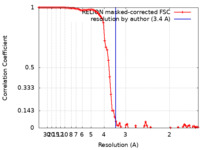
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Rovsnik U / Zhuang Y | |||||||||
| 資金援助 |  スウェーデン, 2件 スウェーデン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Life Sci Alliance / 年: 2021 ジャーナル: Life Sci Alliance / 年: 2021タイトル: Dynamic closed states of a ligand-gated ion channel captured by cryo-EM and simulations. 著者: Urška Rovšnik / Yuxuan Zhuang / Björn O Forsberg / Marta Carroni / Linnea Yvonnesdotter / Rebecca J Howard / Erik Lindahl /   要旨: Ligand-gated ion channels are critical mediators of electrochemical signal transduction across evolution. Biophysical and pharmacological characterization of these receptor proteins relies on high- ...Ligand-gated ion channels are critical mediators of electrochemical signal transduction across evolution. Biophysical and pharmacological characterization of these receptor proteins relies on high-quality structures in multiple, subtly distinct functional states. However, structural data in this family remain limited, particularly for resting and intermediate states on the activation pathway. Here, we report cryo-electron microscopy (cryo-EM) structures of the proton-activated ligand-gated ion channel (GLIC) under three pH conditions. Decreased pH was associated with improved resolution and side chain rearrangements at the subunit/domain interface, particularly involving functionally important residues in the β1-β2 and M2-M3 loops. Molecular dynamics simulations substantiated flexibility in the closed-channel extracellular domains relative to the transmembrane ones and supported electrostatic remodeling around E35 and E243 in proton-induced gating. Exploration of secondary cryo-EM classes further indicated a low-pH population with an expanded pore. These results allow us to define distinct protonation and activation steps in pH-stimulated conformational cycling in GLIC, including interfacial rearrangements largely conserved in the pentameric channel family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11208.map.gz emd_11208.map.gz | 7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11208-v30.xml emd-11208-v30.xml emd-11208.xml emd-11208.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11208_fsc.xml emd_11208_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11208.png emd_11208.png | 114.7 KB | ||
| マスクデータ |  emd_11208_msk_1.map emd_11208_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11208.cif.gz emd-11208.cif.gz | 5.8 KB | ||
| その他 |  emd_11208_additional_1.map.gz emd_11208_additional_1.map.gz emd_11208_half_map_1.map.gz emd_11208_half_map_1.map.gz emd_11208_half_map_2.map.gz emd_11208_half_map_2.map.gz | 49.4 MB 49.5 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11208 http://ftp.pdbj.org/pub/emdb/structures/EMD-11208 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11208 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11208 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11208_validation.pdf.gz emd_11208_validation.pdf.gz | 648.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11208_full_validation.pdf.gz emd_11208_full_validation.pdf.gz | 647.9 KB | 表示 | |
| XML形式データ |  emd_11208_validation.xml.gz emd_11208_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_11208_validation.cif.gz emd_11208_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11208 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11208 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11208 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11208 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11208.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11208.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GLIC, pentameric ligand-gated ion channel, sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11208_msk_1.map emd_11208_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
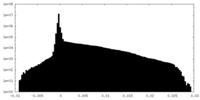
| 密度ヒストグラム |
-追加マップ: GLIC, pentameric ligand-gated ion channel, unsharpened map
| ファイル | emd_11208_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GLIC, pentameric ligand-gated ion channel, unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
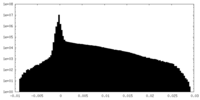
| 密度ヒストグラム |
-ハーフマップ: GLIC, pentameric ligand-gated ion channel, half map 1
| ファイル | emd_11208_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GLIC, pentameric ligand-gated ion channel, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
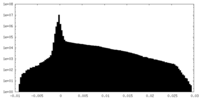
| 密度ヒストグラム |
-ハーフマップ: GLIC, pentameric ligand-gated ion channel, half map 2
| ファイル | emd_11208_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GLIC, pentameric ligand-gated ion channel, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pentameric ion channel
| 全体 | 名称: Pentameric ion channel |
|---|---|
| 要素 |
|
-超分子 #1: Pentameric ion channel
| 超分子 | 名称: Pentameric ion channel / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) |
| 分子量 | 理論値: 180 KDa |
-分子 #1: Proton-gated ion channel
| 分子 | 名称: Proton-gated ion channel / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 |
| 分子量 | 理論値: 36.29175 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AQDMVSPPPP IADEPLTVNT GIYLIECYSL DDKAETFKVN AFLSLSWKDR RLAFDPVRSG VRVKTYEPEA IWIPEIRFVN VENARDADV VDISVSPDGT VQYLERFSAR VLSPLDFRRY PFDSQTLHIY LIVRSVDTRN IVLAVDLEKV GKNDDVFLTG W DIESFTAV ...文字列: AQDMVSPPPP IADEPLTVNT GIYLIECYSL DDKAETFKVN AFLSLSWKDR RLAFDPVRSG VRVKTYEPEA IWIPEIRFVN VENARDADV VDISVSPDGT VQYLERFSAR VLSPLDFRRY PFDSQTLHIY LIVRSVDTRN IVLAVDLEKV GKNDDVFLTG W DIESFTAV VKPANFALED RLESKLDYQL RISRQYFSYI PNIILPMLFI LFISWTAFWS TSYEANVTLV VSTLIAHIAF NI LVETNLP KTPYMTYTGA IIFMIYLFYF VAVIEVTVQH YLKVESQPAR AASITRASRI AFPVVFLLAN IILAFLFFGF UniProtKB: Proton-gated ion channel |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 1.5 s force -1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 6000 / 平均露光時間: 6.0 sec. / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 0.0028 µm 最小 デフォーカス(補正後): 0.0009000000000000001 µm 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 3.8000000000000003 µm 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X