+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1077 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Visualization of ribosome-recycling factor on the Escherichia coli 70S ribosome: functional implications. | |||||||||
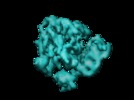
 マップデータ マップデータ | Complex of e.coli 70S ribosome and ribosome recycling factor (RRF). | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Ribosome recycling factor / Ribosome recycling factor domain / RRF superfamily / Ribosome recycling factor / translational termination / cytoplasm / Ribosome-recycling factor 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Agrawal RK | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2004 ジャーナル: Proc Natl Acad Sci U S A / 年: 2004タイトル: Visualization of ribosome-recycling factor on the Escherichia coli 70S ribosome: functional implications. 著者: Rajendra K Agrawal / Manjuli R Sharma / Michael C Kiel / Go Hirokawa / Timothy M Booth / Christian M T Spahn / Robert A Grassucci / Akira Kaji / Joachim Frank /  要旨: After the termination step of protein synthesis, a deacylated tRNA and mRNA remain associated with the ribosome. The ribosome-recycling factor (RRF), together with elongation factor G (EF-G), ...After the termination step of protein synthesis, a deacylated tRNA and mRNA remain associated with the ribosome. The ribosome-recycling factor (RRF), together with elongation factor G (EF-G), disassembles this posttermination complex into mRNA, tRNA, and the ribosome. We have obtained a three-dimensional cryo-electron microscopic map of a complex of the Escherichia coli 70S ribosome and RRF. We find that RRF interacts mainly with the segments of the large ribosomal subunit's (50S) rRNA helices that are involved in the formation of two central intersubunit bridges, B2a and B3. The binding of RRF induces considerable conformational changes in some of the functional domains of the ribosome. As compared to its binding position derived previously by hydroxyl radical probing study, we find that RRF binds further inside the intersubunit space of the ribosome such that the tip of its domain I is shifted (by approximately 13 A) toward protein L5 within the central protuberance of the 50S subunit, and domain II is oriented more toward the small ribosomal subunit (30S). Overlapping binding sites of RRF, EF-G, and the P-site tRNA suggest that the binding of EF-G would trigger the removal of deacylated tRNA from the P site by moving RRF toward the ribosomal E site, and subsequent removal of mRNA may be induced by a shift in the position of 16S rRNA helix 44, which harbors part of the mRNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1077.map.gz emd_1077.map.gz | 7.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1077-v30.xml emd-1077-v30.xml emd-1077.xml emd-1077.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1077.gif 1077.gif | 16.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1077 http://ftp.pdbj.org/pub/emdb/structures/EMD-1077 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1077 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1077 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1077_validation.pdf.gz emd_1077_validation.pdf.gz | 329.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1077_full_validation.pdf.gz emd_1077_full_validation.pdf.gz | 328.9 KB | 表示 | |
| XML形式データ |  emd_1077_validation.xml.gz emd_1077_validation.xml.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1077 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1077 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1077 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1077 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1077.map.gz / 形式: CCP4 / 大きさ: 8.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1077.map.gz / 形式: CCP4 / 大きさ: 8.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Complex of e.coli 70S ribosome and ribosome recycling factor (RRF). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
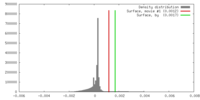
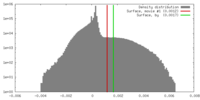
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 70S-RRF complex of e.coli
| 全体 | 名称: 70S-RRF complex of e.coli |
|---|---|
| 要素 |
|
-超分子 #1000: 70S-RRF complex of e.coli
| 超分子 | 名称: 70S-RRF complex of e.coli / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 2.5 MDa / 理論値: 2.5 MDa / 手法: sedimentation |
-超分子 #1: 70S
| 超分子 | 名称: 70S / タイプ: complex / ID: 1 / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 分子量 | 実験値: 2.48 MDa / 理論値: 2.48 MDa |
-分子 #1: RRF
| 分子 | 名称: RRF / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 20 KDa / 理論値: 20 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: 52 mM Tris-HCL (pH7.5), 25 mM KCL, 5.5 mM NH4Cl, 11 mM Mg(OAc)2 0.3 mM Beta-ME |
|---|---|
| グリッド | 詳細: quantifoil grids with holy carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 30 % / チャンバー内温度: 93 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: two side blotting plunger / 手法: Blot for 2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 93 K |
| 日付 | 2002年6月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 14 µm / 実像数: 76 / 平均電子線量: 20 e/Å2 / Od range: 1.2 / ビット/ピクセル: 12 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 49696 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.4 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Cryo transfer / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: defocus groups |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER 詳細: Final map was calculated from 23 CTF corrected defocus groups 使用した粒子像数: 51217 |
| 最終 角度割当 | 詳細: SPIDER: theta 15 degrees, phi 15 degrees |
-原子モデル構築 1
| ソフトウェア | 名称: manual |
|---|---|
| 詳細 | Protocol: Rigid Body. the RRF was fitted as single rigid body |
| 精密化 | プロトコル: RIGID BODY FIT 当てはまり具合の基準: cross correlation coefficient |
| 得られたモデル |  PDB-1t1m:  PDB-1t1o: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)