+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0264 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
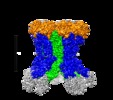
| タイトル | Type VI membrane complex | |||||||||
 マップデータ マップデータ | MolRes Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Membrane complex / tether / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Rapisarda C / Fronzes R | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2019 ジャーナル: EMBO J / 年: 2019タイトル: and high-resolution cryo-EM structure of a bacterial type VI secretion system membrane complex. 著者: Chiara Rapisarda / Yassine Cherrak / Romain Kooger / Victoria Schmidt / Riccardo Pellarin / Laureen Logger / Eric Cascales / Martin Pilhofer / Eric Durand / Rémi Fronzes /   要旨: Bacteria have evolved macromolecular machineries that secrete effectors and toxins to survive and thrive in diverse environments. The type VI secretion system (T6SS) is a contractile machine that is ...Bacteria have evolved macromolecular machineries that secrete effectors and toxins to survive and thrive in diverse environments. The type VI secretion system (T6SS) is a contractile machine that is related to phages. It is composed of a phage tail-like structure inserted in the bacterial cell envelope by a membrane complex (MC) comprising the TssJ, TssL and TssM proteins. We previously reported the low-resolution negative-stain electron microscopy structure of the enteroaggregative MC and proposed a rotational 5-fold symmetry with a TssJ:TssL:TssM stoichiometry of 2:2:2. Here, cryo-electron tomography analyses of the T6SS MC confirm the 5-fold symmetry and identify the regions of the structure that insert into the bacterial membranes. A high-resolution model obtained by single-particle cryo-electron microscopy highlights new features: five additional copies of TssJ, yielding a TssJ:TssL:TssM stoichiometry of 3:2:2, an 11-residue loop in TssM, protruding inside the lumen of the MC and constituting a functionally important periplasmic gate, and hinge regions. Based on these data, we propose an updated model on MC structure and dynamics during T6SS assembly and function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0264.map.gz emd_0264.map.gz | 372.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0264-v30.xml emd-0264-v30.xml emd-0264.xml emd-0264.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0264.png emd_0264.png | 164.1 KB | ||
| Filedesc metadata |  emd-0264.cif.gz emd-0264.cif.gz | 6.3 KB | ||
| その他 |  emd_0264_additional.map.gz emd_0264_additional.map.gz emd_0264_half_map_1.map.gz emd_0264_half_map_1.map.gz emd_0264_half_map_2.map.gz emd_0264_half_map_2.map.gz | 377.2 MB 380.6 MB 381.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0264 http://ftp.pdbj.org/pub/emdb/structures/EMD-0264 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0264 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0264 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0264_validation.pdf.gz emd_0264_validation.pdf.gz | 345.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0264_full_validation.pdf.gz emd_0264_full_validation.pdf.gz | 344.9 KB | 表示 | |
| XML形式データ |  emd_0264_validation.xml.gz emd_0264_validation.xml.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0264 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0264 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0264 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0264 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0264.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0264.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MolRes Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Refinement map
| ファイル | emd_0264_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
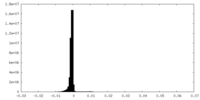
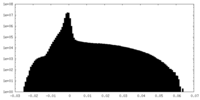
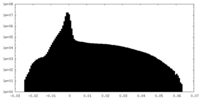
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_0264_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
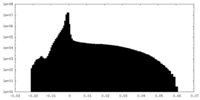
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_0264_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
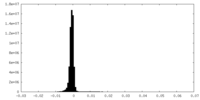
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Membrane complex of the type VI secretion system (TssM and TssJ)
| 全体 | 名称: Membrane complex of the type VI secretion system (TssM and TssJ) |
|---|---|
| 要素 |
|
-超分子 #1: Membrane complex of the type VI secretion system (TssM and TssJ)
| 超分子 | 名称: Membrane complex of the type VI secretion system (TssM and TssJ) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: ImcF-like family protein
| 分子 | 名称: ImcF-like family protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 127.255367 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNKLACLSGR FGRPGIVFIG VAALWWLITR YGAFLGAETR RDQILLLILL SLGLLFVCYL PVMKKYVQEL TYRRRARKEQ RLPDDEERL AQTPPRYVTV QDIRHTLRRQ YGRFWGRKIR ILLITGTASE VELLTPGLTE QFWQEEQGTL LLWGGEPSQP E NADWLAAL ...文字列: MNKLACLSGR FGRPGIVFIG VAALWWLITR YGAFLGAETR RDQILLLILL SLGLLFVCYL PVMKKYVQEL TYRRRARKEQ RLPDDEERL AQTPPRYVTV QDIRHTLRRQ YGRFWGRKIR ILLITGTASE VELLTPGLTE QFWQEEQGTL LLWGGEPSQP E NADWLAAL RRLRYRPADG IVWVTSGLSE TLSAPLTEDA LDRVSRAVSS CCERLGWRLP LYVWSLQESP DERGRITQPV GC LLPAECS SDKLKAQLQA MLPGLVAQGI QQICCAPRYY FLLSLAERFR RNIDAVVEPL SVLLRPYRQL LLAGIVFSPA TVG GERSVR HRWRMDNRWE ALPETVQQLP VRLQPSRTGH NWRRSLAVMA AILMMAQGTG MVVSFLANRS LVAEVQEQIR PAQN QQLSP AERLQALLNL QKSLARLQYR EEHGAPWYLR AGMNQNADLL VVVMPLYAQN AHLLLRDAAA AHLEQQLRTF IRLPP DSPQ RGKMAKAAYD QLRLYLMLAQ PQHMEPAWFS RTLMREWPQR DGVSAVFWQA NGPTLLAYYA SGIITHPQWK LTADEE LVS QSRTLLLRHL GTQNSDAMLY QKMLARVAHQ FADMRLTDMT GDTDVSRLFF TDEVVPGMFT RQAWEEAVLP SIDTVIN ER REEMDWVLTD GRQKAPSPVS PEALRQRLTT RYFADFGNAW LNFLNSLHLR KAQMLSDVTE QLTLMADVRQ SPLVALMN T LAVQGRTGQP REAVTDSLVK SARNLLSQEK QPVAVPESRL HGPLATTFGP VLALMDNQNN SADMLNLQTY LTRVTQVRL RLQQIAGSSD PQAMMQMLAQ TVLQGKSVDL TDTRDYGSLT AAGLGQEWYG FGQTVFVRPM EQAWQQVLTP AAESLNARWR TAVVDGWNN AFSGRYPFKN VSSDASLPLL AKYLNTDTGR IARFLQNNLS GVLHREGSRW VPDTINTRGL TFNPAFLKAI N TLSEIADV AFTTGNAGLH FELRPGTAAG VMQTTLITDN QKLIYVNQMP VWKRFTWPAD TEAPGASLSW VSTQAGTRQY AD LPGSWGL IRLLEMARRK AAPGVASGWS LSWQAQDGRM LNYTLRTEAG EGPLVLLKLR NFVLPETVFE LSGTSAFTGN DED AGDTVE ETD UniProtKB: ImcF-like family protein |
-分子 #2: Type VI secretion system protein VasD
| 分子 | 名称: Type VI secretion system protein VasD / タイプ: protein_or_peptide / ID: 2 / コピー数: 15 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 20.313236 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAIIAGKAGY GLIIALFSLS LSGCGLTQRV ADGTVSATKS LFYRQIKTLH LDIRAREAIN TSAAGIPLSV VVRIYQLKDN RSFDSADYQ ALFTGDNEIL AGDIIAQKDV WLQPGGSVAV DMPLDDAAKF TGVAAMFLEP DQKKNTWRVV LGRDELEPDT P RLIEVSGN TLTLLPVKDK WSHPQFEK UniProtKB: Type VI secretion system protein VasD |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: GRAPHENE OXIDE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 120.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 0.003 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.002 µm / 最小 デフォーカス(公称値): 0.0005 µm / 倍率(公称値): 120000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C5 (5回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 36828 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X