+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9116 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM reconstruction of native lens connexin-46/50 at 3.4 angstrom resolution | |||||||||
 マップデータ マップデータ | Cx46/50 D6 symmetrized map, 3.4 angstrom resolution, sharpened | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報gap junction hemi-channel activity / gap junction-mediated intercellular transport / connexin complex / gap junction channel activity / visual perception / cell-cell signaling / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
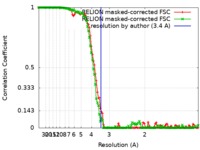
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Myers JB / Reichow SL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Structure of native lens connexin 46/50 intercellular channels by cryo-EM. 著者: Janette B Myers / Bassam G Haddad / Susan E O'Neill / Dror S Chorev / Craig C Yoshioka / Carol V Robinson / Daniel M Zuckerman / Steve L Reichow /   要旨: Gap junctions establish direct pathways for cell-to-cell communication through the assembly of twelve connexin subunits that form intercellular channels connecting neighbouring cells. Co-assembly of ...Gap junctions establish direct pathways for cell-to-cell communication through the assembly of twelve connexin subunits that form intercellular channels connecting neighbouring cells. Co-assembly of different connexin isoforms produces channels with unique properties and enables communication across cell types. Here we used single-particle cryo-electron microscopy to investigate the structural basis of connexin co-assembly in native lens gap junction channels composed of connexin 46 and connexin 50 (Cx46/50). We provide the first comparative analysis to connexin 26 (Cx26), which-together with computational studies-elucidates key energetic features governing gap junction permselectivity. Cx46/50 adopts an open-state conformation that is distinct from the Cx26 crystal structure, yet it appears to be stabilized by a conserved set of hydrophobic anchoring residues. 'Hot spots' of genetic mutations linked to hereditary cataract formation map to the core structural-functional elements identified in Cx46/50, suggesting explanations for many of the disease-causing effects. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9116.map.gz emd_9116.map.gz | 13.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9116-v30.xml emd-9116-v30.xml emd-9116.xml emd-9116.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9116_fsc_1.xml emd_9116_fsc_1.xml emd_9116_fsc_2.xml emd_9116_fsc_2.xml | 11 KB 10.9 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9116.png emd_9116.png | 139.2 KB | ||
| マスクデータ |  emd_9116_msk_1.map emd_9116_msk_1.map emd_9116_msk_2.map emd_9116_msk_2.map | 125 MB 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_9116_additional_1.map.gz emd_9116_additional_1.map.gz emd_9116_additional_2.map.gz emd_9116_additional_2.map.gz emd_9116_additional_3.map.gz emd_9116_additional_3.map.gz | 20.1 MB 95.5 MB 95.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9116 http://ftp.pdbj.org/pub/emdb/structures/EMD-9116 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9116 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9116 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9116_validation.pdf.gz emd_9116_validation.pdf.gz | 408.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9116_full_validation.pdf.gz emd_9116_full_validation.pdf.gz | 407.8 KB | 表示 | |
| XML形式データ |  emd_9116_validation.xml.gz emd_9116_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_9116_validation.cif.gz emd_9116_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9116 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9116 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9116 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9116 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6mhqMC  6mhyMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10212 (タイトル: CryoEM reconstruction of native lens connexin-46/50 at 3.4 angstrom resolution EMPIAR-10212 (タイトル: CryoEM reconstruction of native lens connexin-46/50 at 3.4 angstrom resolutionData size: 774.5 Data #1: Unaligned frame stacks - MP38 dataset 01 [micrographs - multiframe] Data #2: Unaligned frame stacks - MP38 dataset 02 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9116.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9116.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cx46/50 D6 symmetrized map, 3.4 angstrom resolution, sharpened | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.665 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9116_msk_1.map emd_9116_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_9116_msk_2.map emd_9116_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cx46/50 D6 symmetrized map, 3.5 angstrom resolution, sharpened
| ファイル | emd_9116_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cx46/50 D6 symmetrized map, 3.5 angstrom resolution, sharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cx46/50 D6 symmetrized map, 3.5 angstrom resolution, unprocessed
| ファイル | emd_9116_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cx46/50 D6 symmetrized map, 3.5 angstrom resolution, unprocessed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cx46/50 D6 symmetrized map, 3.4 angstrom resolution, unprocessed
| ファイル | emd_9116_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cx46/50 D6 symmetrized map, 3.4 angstrom resolution, unprocessed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Connexin-46 gap junction
| 全体 | 名称: Connexin-46 gap junction |
|---|---|
| 要素 |
|
-超分子 #1: Connexin-46 gap junction
| 超分子 | 名称: Connexin-46 gap junction / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  細胞中の位置: C-terminal truncated version isolated from lens core |
| 分子量 | 理論値: 450 KDa |
-分子 #1: Gap junction alpha-3 protein, connexin-46
| 分子 | 名称: Gap junction alpha-3 protein, connexin-46 / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 37.968516 KDa |
| 配列 | 文字列: MGDWSFLGRL LENAQEHSTV IGKVWLTVLF IFRILVLGAA AEEVWGDEQS DFTCNTQQPG CENVCYDRAF PISHVRFWVL QIIFVSTPT LIYLGHVLHL VRMEEKRKER EEEPPKAAGP AEEHQDPAPV RDDRGKVRIA GALLRTYVFN IIFKTLFEVG F IAGQYFLY ...文字列: MGDWSFLGRL LENAQEHSTV IGKVWLTVLF IFRILVLGAA AEEVWGDEQS DFTCNTQQPG CENVCYDRAF PISHVRFWVL QIIFVSTPT LIYLGHVLHL VRMEEKRKER EEEPPKAAGP AEEHQDPAPV RDDRGKVRIA GALLRTYVFN IIFKTLFEVG F IAGQYFLY GFQLKPLYRC DRWPCPNTVD CFISRPTEKT IFILFMLAVA CVSLLLNVLE IYHLGWKKLK QGMTSPFRPD TP GSRAGSA KPMGGSPLLL PPNSAPPAVT IGFPPYYAPS ASSLGQASAP GYPEPPLPAA LPGTPGTPGT PGTLGGGGGN QGL RAPAQN CANREAEPQT SARKASPPAS TP |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.35 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: 10 sec wait before blotting, 4.0 second blot before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 30 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.25 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)