+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8150 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure and dynamics of single-isoform recombinant neuronal human tubulin | ||||||||||||
 マップデータ マップデータ | None | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | microtubules / tubulin / single isoform / recombinant / dynamic instability / STRUCTURAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報netrin receptor binding / Post-chaperonin tubulin folding pathway / axonemal microtubule / dorsal root ganglion development / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane / Cilium Assembly / Carboxyterminal post-translational modifications of tubulin / organelle transport along microtubule / glial cell differentiation / forebrain morphogenesis ...netrin receptor binding / Post-chaperonin tubulin folding pathway / axonemal microtubule / dorsal root ganglion development / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane / Cilium Assembly / Carboxyterminal post-translational modifications of tubulin / organelle transport along microtubule / glial cell differentiation / forebrain morphogenesis / Sealing of the nuclear envelope (NE) by ESCRT-III / Intraflagellar transport / neuron projection arborization / cytoskeleton-dependent intracellular transport / cerebellar cortex morphogenesis / Formation of tubulin folding intermediates by CCT/TriC / dentate gyrus development / COPI-independent Golgi-to-ER retrograde traffic / pyramidal neuron differentiation / Gap junction assembly / Prefoldin mediated transfer of substrate to CCT/TriC / Kinesins / Assembly and cell surface presentation of NMDA receptors / centrosome cycle / motor behavior / COPI-dependent Golgi-to-ER retrograde traffic / response to L-glutamate / smoothened signaling pathway / intercellular bridge / regulation of synapse organization / startle response / locomotory exploration behavior / Recycling pathway of L1 / microtubule polymerization / microtubule-based process / RHO GTPases activate IQGAPs / response to tumor necrosis factor / Hedgehog 'off' state / COPI-mediated anterograde transport / response to mechanical stimulus / Activation of AMPK downstream of NMDARs / condensed chromosome / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / Loss of Nlp from mitotic centrosomes / Loss of proteins required for interphase microtubule organization from the centrosome / Recruitment of mitotic centrosome proteins and complexes / homeostasis of number of cells within a tissue / Recruitment of NuMA to mitotic centrosomes / Anchoring of the basal body to the plasma membrane / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / MHC class II antigen presentation / Resolution of Sister Chromatid Cohesion / AURKA Activation by TPX2 / cellular response to calcium ion / adult locomotory behavior / filopodium / axon guidance / cell periphery / Translocation of SLC2A4 (GLUT4) to the plasma membrane / RHO GTPases Activate Formins / intracellular protein transport / neuron migration / peptide binding / synapse organization / neuromuscular junction / visual learning / PKR-mediated signaling / recycling endosome / cerebral cortex development / structural constituent of cytoskeleton / mitotic spindle / memory / microtubule cytoskeleton organization / Aggrephagy / HCMV Early Events / cytoplasmic ribonucleoprotein granule / Separation of Sister Chromatids / The role of GTSE1 in G2/M progression after G2 checkpoint / microtubule cytoskeleton / Regulation of PLK1 Activity at G2/M Transition / lamellipodium / mitotic cell cycle / gene expression / growth cone / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / neuron apoptotic process / microtubule / hydrolase activity / protein heterodimerization activity / cell division / axon / GTPase activity / neuronal cell body / dendrite / protein-containing complex binding / GTP binding / structural molecule activity / extracellular exosome / identical protein binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | ||||||||||||
 データ登録者 データ登録者 | Vemu A / Atherton J | ||||||||||||
| 資金援助 |  英国, 英国,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2016 ジャーナル: J Biol Chem / 年: 2016タイトル: Structure and Dynamics of Single-isoform Recombinant Neuronal Human Tubulin. 著者: Annapurna Vemu / Joseph Atherton / Jeffrey O Spector / Agnieszka Szyk / Carolyn A Moores / Antonina Roll-Mecak /   要旨: Microtubules are polymers that cycle stochastically between polymerization and depolymerization, i.e. they exhibit "dynamic instability." This behavior is crucial for cell division, motility, and ...Microtubules are polymers that cycle stochastically between polymerization and depolymerization, i.e. they exhibit "dynamic instability." This behavior is crucial for cell division, motility, and differentiation. Although studies in the last decade have made fundamental breakthroughs in our understanding of how cellular effectors modulate microtubule dynamics, analysis of the relationship between tubulin sequence, structure, and dynamics has been held back by a lack of dynamics measurements with and structural characterization of homogeneous isotypically pure engineered tubulin. Here, we report for the first time the cryo-EM structure and in vitro dynamics parameters of recombinant isotypically pure human tubulin. α1A/βIII is a purely neuronal tubulin isoform. The 4.2-Å structure of post-translationally unmodified human α1A/βIII microtubules shows overall similarity to that of heterogeneous brain microtubules, but it is distinguished by subtle differences at polymerization interfaces, which are hot spots for sequence divergence between tubulin isoforms. In vitro dynamics assays show that, like mosaic brain microtubules, recombinant homogeneous microtubules undergo dynamic instability, but they polymerize slower and have fewer catastrophes. Interestingly, we find that epitaxial growth of α1A/βIII microtubules from heterogeneous brain seeds is inefficient but can be fully rescued by incorporating as little as 5% of brain tubulin into the homogeneous α1A/βIII lattice. Our study establishes a system to examine the structure and dynamics of mammalian microtubules with well defined tubulin species and is a first and necessary step toward uncovering how tubulin genetic and chemical diversity is exploited to modulate intrinsic microtubule dynamics. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8150.map.gz emd_8150.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8150-v30.xml emd-8150-v30.xml emd-8150.xml emd-8150.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8150.png emd_8150.png | 234.8 KB | ||
| Filedesc metadata |  emd-8150.cif.gz emd-8150.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8150 http://ftp.pdbj.org/pub/emdb/structures/EMD-8150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8150 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8150_validation.pdf.gz emd_8150_validation.pdf.gz | 426.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8150_full_validation.pdf.gz emd_8150_full_validation.pdf.gz | 425.8 KB | 表示 | |
| XML形式データ |  emd_8150_validation.xml.gz emd_8150_validation.xml.gz | 4.3 KB | 表示 | |
| CIF形式データ |  emd_8150_validation.cif.gz emd_8150_validation.cif.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8150 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8150 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5jcoMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10071 (タイトル: Structure and Dynamics of Single-isoform Recombinant Neuronal Human Tubulin EMPIAR-10071 (タイトル: Structure and Dynamics of Single-isoform Recombinant Neuronal Human TubulinData size: 487.7 Data #1: Unaligned frame stacks of GMPCPP-bound alpha1a beta3 recombinant tubulin microtubules [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8150.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8150.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.22 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Microtubules
| 全体 | 名称: Microtubules |
|---|---|
| 要素 |
|
-超分子 #1: Microtubules
| 超分子 | 名称: Microtubules / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: 14pf GMPCPP-bound microtubule composed of human beta3 and alpha1a tubulin |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 110 kDa/nm |
-分子 #1: Tubulin beta-3 chain
| 分子 | 名称: Tubulin beta-3 chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 47.809926 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MREIVHIQAG QCGNQIGAKF WEVISDEHGI DPSGNYVGDS DLQLERISVY YNEASSHKYV PRAILVDLEP GTMDSVRSGA FGHLFRPDN FIFGQSGAGN NWAKGHYTEG AELVDSVLDV VRKECENCDC LQGFQLTHSL GGGTGSGMGT LLISKVREEY P DRIMNTFS ...文字列: MREIVHIQAG QCGNQIGAKF WEVISDEHGI DPSGNYVGDS DLQLERISVY YNEASSHKYV PRAILVDLEP GTMDSVRSGA FGHLFRPDN FIFGQSGAGN NWAKGHYTEG AELVDSVLDV VRKECENCDC LQGFQLTHSL GGGTGSGMGT LLISKVREEY P DRIMNTFS VVPSPKVSDT VVEPYNATLS IHQLVENTDE TYCIDNEALY DICFRTLKLA TPTYGDLNHL VSATMSGVTT SL RFPGQLN ADLRKLAVNM VPFPRLHFFM PGFAPLTARG SQQYRALTVP ELTQQMFDAK NMMAACDPRH GRYLTVATVF RGR MSMKEV DEQMLAIQSK NSSYFVEWIP NNVKVAVCDI PPRGLKMSST FIGNSTAIQE LFKRISEQFT AMFRRKAFLH WYTG EGMDE MEFTEAESNM NDLVSEYQQY Q UniProtKB: Tubulin beta-3 chain |
-分子 #2: Tubulin alpha-1A chain
| 分子 | 名称: Tubulin alpha-1A chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 48.649023 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHP EQLITGKEDA ANNYARGHYT IGKEIIDLVL DRIRKLADQC TGLQGFLVFH SFGGGTGSGF TSLLMERLSV D YGKKSKLE ...文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHP EQLITGKEDA ANNYARGHYT IGKEIIDLVL DRIRKLADQC TGLQGFLVFH SFGGGTGSGF TSLLMERLSV D YGKKSKLE FSIYPAPQVS TAVVEPYNSI LTTHTTLEHS DCAFMVDNEA IYDICRRNLD IERPTYTNLN RLIGQIVSSI TA SLRFDGA LNVDLTEFQT NLVPYPRIHF PLATYAPVIS AEKAYHEQLS VAEITNACFE PANQMVKCDP RHGKYMACCL LYR GDVVPK DVNAAIATIK TKRTIQFVDW CPTGFKVGIN YQPPTVVPGG DLAKVQRAVC MLSNTTAIAE AWARLDHKFD LMYA KRAFV HWYVGEGMEE GEFSEAREDM AALEKDYEEV GV UniProtKB: Tubulin alpha-1A chain |
-分子 #3: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER
| 分子 | 名称: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER / タイプ: ligand / ID: 3 / コピー数: 6 / 式: G2P |
|---|---|
| 分子量 | 理論値: 521.208 Da |
| Chemical component information | 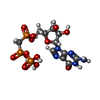 ChemComp-G2P: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 12 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 6 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.9 / 構成要素 - 名称: BRB80 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 検出モード: INTEGRATING / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 8.86 Å 想定した対称性 - らせんパラメータ - ΔΦ: -25.71 ° 想定した対称性 - らせんパラメータ - 軸対称性: C14 (14回回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 4.0 Å / 解像度の算出法: OTHER 詳細: Overall map resolution was 4.2 Angstrom by gold-standard FSCtrue (Chen et al., 2013). The tubulin portion of the map was at higher resolution: at least 4 Angstrom according to the Blocres resolution measure. 使用した粒子像数: 10164 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER / 詳細: Synthetic Kinesin Decorated Microtubule |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー








































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















