+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6310 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A Virus that Infects a Hyperthermophile Encapsidates A-Form DNA | |||||||||
 マップデータ マップデータ | Reconstruction of SIRV2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | archaeal virus / helical symmetry | |||||||||
| 機能・相同性 | Sulfolobus virus, coat protein, C-terminal / Sulfolobus virus coat protein C terminal / helical viral capsid / DNA binding / Major capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Sulfolobus islandicus rod-shaped virus 2 (ウイルス) Sulfolobus islandicus rod-shaped virus 2 (ウイルス) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | DiMaio F / Yu X / Rensen E / Krupovic M / Prangishvili D / Egelman E | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2015 ジャーナル: Science / 年: 2015タイトル: Virology. A virus that infects a hyperthermophile encapsidates A-form DNA. 著者: Frank DiMaio / Xiong Yu / Elena Rensen / Mart Krupovic / David Prangishvili / Edward H Egelman /   要旨: Extremophiles, microorganisms thriving in extreme environmental conditions, must have proteins and nucleic acids that are stable at extremes of temperature and pH. The nonenveloped, rod-shaped virus ...Extremophiles, microorganisms thriving in extreme environmental conditions, must have proteins and nucleic acids that are stable at extremes of temperature and pH. The nonenveloped, rod-shaped virus SIRV2 (Sulfolobus islandicus rod-shaped virus 2) infects the hyperthermophilic acidophile Sulfolobus islandicus, which lives at 80°C and pH 3. We have used cryo-electron microscopy to generate a three-dimensional reconstruction of the SIRV2 virion at ~4 angstrom resolution, which revealed a previously unknown form of virion organization. Although almost half of the capsid protein is unstructured in solution, this unstructured region folds in the virion into a single extended α helix that wraps around the DNA. The DNA is entirely in the A-form, which suggests a common mechanism with bacterial spores for protecting DNA in the most adverse environments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6310.map.gz emd_6310.map.gz | 29.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6310-v30.xml emd-6310-v30.xml emd-6310.xml emd-6310.xml | 8.5 KB 8.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6310.gif 400_6310.gif 80_6310.gif 80_6310.gif | 76.7 KB 5.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6310 http://ftp.pdbj.org/pub/emdb/structures/EMD-6310 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6310 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6310 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6310_validation.pdf.gz emd_6310_validation.pdf.gz | 383.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6310_full_validation.pdf.gz emd_6310_full_validation.pdf.gz | 383.4 KB | 表示 | |
| XML形式データ |  emd_6310_validation.xml.gz emd_6310_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6310 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6310 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6310 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6310 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3j9xMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10060 (タイトル: Subset of image stack used for 3D reconstruction EMPIAR-10060 (タイトル: Subset of image stack used for 3D reconstructionData size: 35.9 Data #1: image segments of SIRV2 [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6310.map.gz / 形式: CCP4 / 大きさ: 31.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6310.map.gz / 形式: CCP4 / 大きさ: 31.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of SIRV2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
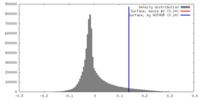
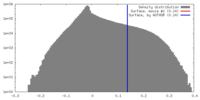
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SIRV2
| 全体 | 名称: SIRV2 |
|---|---|
| 要素 |
|
-超分子 #1000: SIRV2
| 超分子 | 名称: SIRV2 / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Sulfolobus islandicus rod-shaped virus 2
| 超分子 | 名称: Sulfolobus islandicus rod-shaped virus 2 / タイプ: virus / ID: 1 / Name.synonym: SIRV2 / NCBI-ID: 157899 / 生物種: Sulfolobus islandicus rod-shaped virus 2 / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: SIRV2 |
|---|---|
| 宿主 | 生物種:   Sulfolobus islandicus (好気性・好酸性) / 別称: ARCHAEA Sulfolobus islandicus (好気性・好酸性) / 別称: ARCHAEA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 6 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 日付 | 2014年9月19日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 437 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | IHRSR |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 2.9 Å 想定した対称性 - らせんパラメータ - ΔΦ: 24.5 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Spider |
| CTF補正 | 詳細: multiply images by CTF, divide reconstruction by sum of CTF**2 |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)