+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3685 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a pre-catalytic spliceosome (B4 map) | |||||||||
 マップデータ マップデータ | sharpened B4 map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Plaschka C / Lin P-C / Nagai K | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Structure of a pre-catalytic spliceosome. 著者: Clemens Plaschka / Pei-Chun Lin / Kiyoshi Nagai /  要旨: Intron removal requires assembly of the spliceosome on precursor mRNA (pre-mRNA) and extensive remodelling to form the spliceosome's catalytic centre. Here we report the cryo-electron microscopy ...Intron removal requires assembly of the spliceosome on precursor mRNA (pre-mRNA) and extensive remodelling to form the spliceosome's catalytic centre. Here we report the cryo-electron microscopy structure of the yeast Saccharomyces cerevisiae pre-catalytic B complex spliceosome at near-atomic resolution. The mobile U2 small nuclear ribonucleoprotein particle (snRNP) associates with U4/U6.U5 tri-snRNP through the U2/U6 helix II and an interface between U4/U6 di-snRNP and the U2 snRNP SF3b-containing domain, which also transiently contacts the helicase Brr2. The 3' region of the U2 snRNP is flexibly attached to the SF3b-containing domain and protrudes over the concave surface of tri-snRNP, where the U1 snRNP may reside before its release from the pre-mRNA 5' splice site. The U6 ACAGAGA sequence forms a hairpin that weakly tethers the 5' splice site. The B complex proteins Prp38, Snu23 and Spp381 bind the Prp8 N-terminal domain and stabilize U6 ACAGAGA stem-pre-mRNA and Brr2-U4 small nuclear RNA interactions. These results provide important insights into the events leading to active site formation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3685.map.gz emd_3685.map.gz | 394 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3685-v30.xml emd-3685-v30.xml emd-3685.xml emd-3685.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3685.png emd_3685.png | 27.8 KB | ||
| その他 |  emd_3685_additional.map.gz emd_3685_additional.map.gz | 385.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3685 http://ftp.pdbj.org/pub/emdb/structures/EMD-3685 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3685 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3685 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3685_validation.pdf.gz emd_3685_validation.pdf.gz | 234.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3685_full_validation.pdf.gz emd_3685_full_validation.pdf.gz | 233.8 KB | 表示 | |
| XML形式データ |  emd_3685_validation.xml.gz emd_3685_validation.xml.gz | 7.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3685 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3685 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3685 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3685 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3682C  3683C  3684C  3686C  3687C  3688C  5nrlC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10180 (タイトル: Structure of a pre-catalytic spliceosome / Data size: 126.5 EMPIAR-10180 (タイトル: Structure of a pre-catalytic spliceosome / Data size: 126.5 Data #1: Polished parcitles [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3685.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3685.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
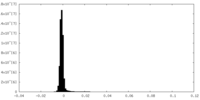
| 注釈 | sharpened B4 map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.43 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpened B4 map
| ファイル | emd_3685_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
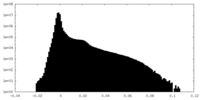
| 注釈 | unsharpened B4 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pre-catalytic B complex Spliceosome
| 全体 | 名称: Pre-catalytic B complex Spliceosome |
|---|---|
| 要素 |
|
-超分子 #1: Pre-catalytic B complex Spliceosome
| 超分子 | 名称: Pre-catalytic B complex Spliceosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 2.5 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.9 構成要素:
詳細: Buffer pH: HEPES, 7.9; EDTA, 8.0 | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK III 詳細: Grids were glow-discharged for 15 s before deposition of 3 microliter sample (~1.5 mg mL-1), and subsequently incubated for 2-3.5 s before blotting and vitrification by plunging into liquid ...詳細: Grids were glow-discharged for 15 s before deposition of 3 microliter sample (~1.5 mg mL-1), and subsequently incubated for 2-3.5 s before blotting and vitrification by plunging into liquid ethane with a Vitrobot Mark III (FEI) operated at 4 degrees Celsius and 100% humidity.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-20 / 実像数: 5115 / 平均露光時間: 16.0 sec. / 平均電子線量: 56.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 5.3 µm / 最小 デフォーカス(補正後): 0.35 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / 温度因子: 105 |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)