+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryogenic electron microscopy 3D map of alpha-catenin ortholog, HMP1 | |||||||||
 マップデータ マップデータ | Cryo-EM 3D map of HMP1 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
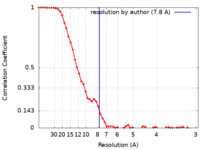
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.8 Å | |||||||||
 データ登録者 データ登録者 | Smith EW / Izard T / Rangarajan ES | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2023 ジャーナル: J Biol Chem / 年: 2023タイトル: The nematode α-catenin ortholog, HMP1, has an extended α-helix when bound to actin filaments. 著者: Erumbi S Rangarajan / Emmanuel W Smith / Tina Izard /  要旨: The regulation of cell-cell junctions during epidermal morphogenesis ensures tissue integrity, a process regulated by α-catenin. This cytoskeletal protein connects the cadherin complex to ...The regulation of cell-cell junctions during epidermal morphogenesis ensures tissue integrity, a process regulated by α-catenin. This cytoskeletal protein connects the cadherin complex to filamentous actin at cell-cell junctions. The cadherin-catenin complex plays key roles in cell physiology, organism development, and disease. While mutagenesis of Caenorhabditis elegans cadherin and catenin shows that these proteins are key for embryonic morphogenesis, we know surprisingly little about their structure and attachment to the cytoskeleton. In contrast to mammalian α-catenin that functions as a dimer or monomer, the α-catenin ortholog from C. elegans, HMP1 for humpback, is a monomer. Our cryogenic electron microscopy (cryoEM) structure of HMP1/α-catenin reveals that the amino- and carboxy-terminal domains of HMP1/α-catenin are disordered and not in contact with the remaining HMP1/α-catenin middle domain. Since the carboxy-terminal HMP1/α-catenin domain is the F-actin-binding domain (FABD), this interdomain constellation suggests that HMP1/α-catenin is constitutively active, which we confirm biochemically. Our perhaps most surprising finding, given the high sequence similarity between the mammalian and nematode proteins, is our cryoEM structure of HMP1/α-catenin bound to F-actin. Unlike the structure of mammalian α-catenin bound to F-actin, binding to F-actin seems to allosterically convert a loop region of the HMP1/α-catenin FABD to extend an HMP1/α-catenin FABD α-helix. We use cryoEM and bundling assays to show for the first time how the FABD of HMP1/α-catenin bundles actin in the absence of force. Collectively, our data advance our understanding of α-catenin regulation of cell-cell contacts and additionally aid our understanding of the evolution of multicellularity in metazoans. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26748.map.gz emd_26748.map.gz | 12.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26748-v30.xml emd-26748-v30.xml emd-26748.xml emd-26748.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26748_fsc.xml emd_26748_fsc.xml | 7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26748.png emd_26748.png | 48.5 KB | ||
| その他 |  emd_26748_half_map_1.map.gz emd_26748_half_map_1.map.gz emd_26748_half_map_2.map.gz emd_26748_half_map_2.map.gz | 12 MB 12 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26748 http://ftp.pdbj.org/pub/emdb/structures/EMD-26748 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26748 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26748 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26748_validation.pdf.gz emd_26748_validation.pdf.gz | 870.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26748_full_validation.pdf.gz emd_26748_full_validation.pdf.gz | 870.4 KB | 表示 | |
| XML形式データ |  emd_26748_validation.xml.gz emd_26748_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_26748_validation.cif.gz emd_26748_validation.cif.gz | 14.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26748 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26748 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26748 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26748 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26748.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26748.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM 3D map of HMP1 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.48 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map A
| ファイル | emd_26748_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
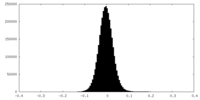
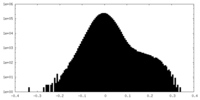
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_26748_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
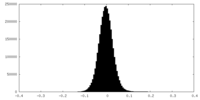
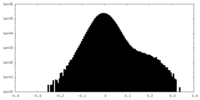
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : alpha-catenin ortholog, HMP1
| 全体 | 名称: alpha-catenin ortholog, HMP1 |
|---|---|
| 要素 |
|
-超分子 #1: alpha-catenin ortholog, HMP1
| 超分子 | 名称: alpha-catenin ortholog, HMP1 / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all / 詳細: Full-length alpha-catenin ortholog, HMP1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 103.99428 KDa |
-分子 #1: alpha-catenin ortholog, HMP1
| 分子 | 名称: alpha-catenin ortholog, HMP1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPANGNSHAY FNIDEVRSKN VLKQITQLIN EVTNITETFP LKPGQTTEGL VATLDAAVAN FLQTGSFAI SKCPIANSDP RAIDLLHEAL GAVQDTGQVM IQTGRDFVRD STSTNKRAIA T NSGRNLLT AVAKFLILAD SIDVKVIVDK VDEVRETAHQ MIEADTKIKV ...文字列: MPANGNSHAY FNIDEVRSKN VLKQITQLIN EVTNITETFP LKPGQTTEGL VATLDAAVAN FLQTGSFAI SKCPIANSDP RAIDLLHEAL GAVQDTGQVM IQTGRDFVRD STSTNKRAIA T NSGRNLLT AVAKFLILAD SIDVKVIVDK VDEVRETAHQ MIEADTKIKV DDLYNLLISQ IE ELDITVR RRAIDLVKPN QRDDLLAARS ALRQTAPLLY TSTRTFVRHP EHEEARRNRD YTA DEMHSA LNALESVLNG QQPKVTFSEY GRIGDLINEI DTFQNRIEID PAHYRRGTDR PDLE GHCER IVSGSASIAD AESTRENRKQ KIVAECNNLR QALQELLTEY EKSTGRRDDN DDIPL GIAE VHKRTKDLRR HLRRAIVDHI SDAFLDTRTP LILLIEAAKE GHEENTRYRS KMFQEH ANE IVSVARLSCQ LSSDVESVSV IQHTAAQLEK LAPQVAQAAI LLCHQPTSKT AQENMET YK NAWFDKVRLL TTALDNITTL DDFLAVSEAH IVEDCERGIK GITANASTPD ENAANCET V DCAAGSIRGR ALRVCDVVDA EMDFLQNSEY TETVKQAVRI LKTQRVDQFA ERASALANR QEAHGLTWDP KTKEEEMNEF INACTLVHDA VKDIRHALLM NRSMNDVDSD VEYEADGVGA ANADANRTI SEQENQQNLM RRLPEEEKKK IQAQIDIFKV TQTRFEREVA KWDETGNDII S LANNMCKI MMSMTEFTRG CGPLKTTMDV IRAAQEISLN GSKLNALARQ IGEESADSQT KK DLLAYLS QITLYCQQLN ICSKVKADVT QVGNELVVSA LDSAMSLIQT ARNLLTAVVQ TVK AAYIAS TKFRRPNANS VRVEWRMAPP KKQPLIRPQK NNAIIRRASE RRPLQPAKVL AEFT RNEIE TGRDSDDEEL DRRHQQRING RL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 240 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 1294 / 平均露光時間: 0.1 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | プロトコル: FLEXIBLE FIT |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X