+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21443 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BurrH bound to DNA Origami Goniometer | ||||||||||||
 マップデータ マップデータ | Final refinement map for BurrH bound to DNA Goniometer tilt DNA | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Paraburkholderia rhizoxinica (バクテリア) Paraburkholderia rhizoxinica (バクテリア) | ||||||||||||
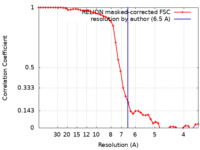
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.5 Å | ||||||||||||
 データ登録者 データ登録者 | Aksel T / Yu Z / Cheng Y / Douglas SM | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Biotechnol / 年: 2021 ジャーナル: Nat Biotechnol / 年: 2021タイトル: Molecular goniometers for single-particle cryo-electron microscopy of DNA-binding proteins. 著者: Tural Aksel / Zanlin Yu / Yifan Cheng / Shawn M Douglas /  要旨: Correct reconstruction of macromolecular structure by cryo-electron microscopy (cryo-EM) relies on accurate determination of the orientation of single-particle images. For small (<100 kDa) DNA-binding proteins, obtaining particle images with sufficiently asymmetric features to correctly guide alignment is challenging. We apply DNA origami to construct molecular goniometers-instruments that precisely orient objects-and use them to dock a DNA-binding protein on a double-helix stage that has user-programmable tilt and rotation angles. We construct goniometers with 14 different stage configurations to orient and visualize the protein just above the cryo-EM grid surface. Each goniometer has a distinct barcode pattern that we use during particle classification to assign angle priors to the bound protein. We use goniometers to obtain a 6.5-Å structure of BurrH, an 82-kDa DNA-binding protein whose helical pseudosymmetry prevents accurate image orientation using traditional cryo-EM. Our approach should be adaptable to other DNA-binding proteins as well as small proteins fused to DNA-binding domains. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Molecular goniometers for single-particle cryo-EM of DNA-binding proteins 著者: Aksel T / Yu Z / Cheng Y / Douglas SM | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21443.map.gz emd_21443.map.gz | 6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21443-v30.xml emd-21443-v30.xml emd-21443.xml emd-21443.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21443_fsc.xml emd_21443_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21443.png emd_21443.png | 62.7 KB | ||
| マスクデータ |  emd_21443_msk_1.map emd_21443_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_21443_half_map_1.map.gz emd_21443_half_map_1.map.gz emd_21443_half_map_2.map.gz emd_21443_half_map_2.map.gz | 6 MB 6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21443 http://ftp.pdbj.org/pub/emdb/structures/EMD-21443 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21443 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21443 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10373 (タイトル: BurrH bound to DNA Origami Goniometer / Data size: 1.3 TB EMPIAR-10373 (タイトル: BurrH bound to DNA Origami Goniometer / Data size: 1.3 TBData #1: DNA Origami Goniometers in complex with BurrH DNA binding protein [micrographs - single frame] Data #2: BurrH particle set used for the final refinement [picked particles - multiframe - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21443.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21443.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final refinement map for BurrH bound to DNA Goniometer tilt DNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
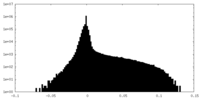
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21443_msk_1.map emd_21443_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
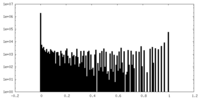
| 密度ヒストグラム |
-ハーフマップ: The first half map for BurrH bound to DNA Goniometer tilt DNA
| ファイル | emd_21443_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The first half map for BurrH bound to DNA Goniometer tilt DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
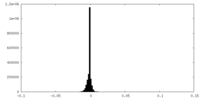
| 密度ヒストグラム |
-ハーフマップ: The second half map for BurrH bound to DNA Goniometer tilt DNA
| ファイル | emd_21443_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The second half map for BurrH bound to DNA Goniometer tilt DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
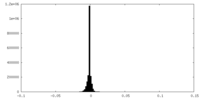
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BurrH bound to DNA
| 全体 | 名称: BurrH bound to DNA |
|---|---|
| 要素 |
|
-超分子 #1: BurrH bound to DNA
| 超分子 | 名称: BurrH bound to DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Paraburkholderia rhizoxinica (バクテリア) Paraburkholderia rhizoxinica (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 81 KDa |
-分子 #1: BurrH
| 分子 | 名称: BurrH / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 配列 | 文字列: MGSTAFVDQD KQMANRLNLS PLERSKIEKQ YGGATTLAFI SNKQNELAQI LSRADILKIA SYDCAAHALQ AVLDCGPMLG KRGFSQSDI VKIAGNIGGA QALQAVLDLE SMLGKRGFSR DDIAKMAGNI GGAQTLQAVL DLESAFRERG FSQADIVKIA G NNGGAQAL ...文字列: MGSTAFVDQD KQMANRLNLS PLERSKIEKQ YGGATTLAFI SNKQNELAQI LSRADILKIA SYDCAAHALQ AVLDCGPMLG KRGFSQSDI VKIAGNIGGA QALQAVLDLE SMLGKRGFSR DDIAKMAGNI GGAQTLQAVL DLESAFRERG FSQADIVKIA G NNGGAQAL YSVLDVEPTL GKRGFSRADI VKIAGNTGGA QALHTVLDLE PALGKRGFSR IDIVKIAANN GGAQALHAVL DL GPTLREC GFSQATIAKI AGNIGGAQAL QMVLDLGPAL GKRGFSQATI AKIAGNIGGA QALQTVLDLE PALCERGFSQ ATI AKMAGN NGGAQALQTV LDLEPALRKR DFRQADIIKI AGNDGGAQAL QAVIEHGPTL RQHGFNLADI VKMAGNIGGA QALQ AVLDL KPVLDEHGFS QPDIVKMAGN IGGAQALQAV LSLGPALRER GFSQPDIVKI AGNTGGAQAL QAVLDLELTL VEHGF SQPD IVRITGNRGG AQALQAVLAL ELTLRERGFS QPDIVKIAGN SGGAQALQAV LDLELTFRER GFSQADIVKI AGNDGG TQA LHAVLDLERM LGERGFSRAD IVNVAGNNGG AQALKAVLEH EATLNERGFS RADIVKIAGN GGGAQALKAV LEHEATL DE RGFSRADIVR IAGNGGGAQA LKAVLEHGPT LNERGFNLTD IVEMAANSGG AQALKAVLEH GPTLRQRGLS LIDIVEIA S NGGAQALKAV LKYGPVLMQA GRSNEEIVHV AARRGGAGRI RKMVAPLLER QWGSGHHHHH H |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | DNA Origami goniometer complexed with BurrH. Total concentration comprises the concentrations of both. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 56.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 20.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 22000 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: RMSD |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X