+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12341 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Akirin2 bound human proteasome | |||||||||
 マップデータ マップデータ | denoised map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | proteasome / nuclear import / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of endopeptidase activity / purine ribonucleoside triphosphate binding / positive regulation of innate immune response / regulation of endopeptidase activity / proteasome core complex / embryo development ending in birth or egg hatching / Regulation of ornithine decarboxylase (ODC) / Cross-presentation of soluble exogenous antigens (endosomes) / Somitogenesis / immune system process ...positive regulation of endopeptidase activity / purine ribonucleoside triphosphate binding / positive regulation of innate immune response / regulation of endopeptidase activity / proteasome core complex / embryo development ending in birth or egg hatching / Regulation of ornithine decarboxylase (ODC) / Cross-presentation of soluble exogenous antigens (endosomes) / Somitogenesis / immune system process / myofibril / NF-kappaB binding / proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / negative regulation of inflammatory response to antigenic stimulus / : / transcription repressor complex / sarcomere / proteasome complex / Regulation of activated PAK-2p34 by proteasome mediated degradation / ciliary basal body / proteolysis involved in protein catabolic process / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D / AUF1 (hnRNP D0) binds and destabilizes mRNA / TNFR2 non-canonical NF-kB pathway / Assembly of the pre-replicative complex / Vpu mediated degradation of CD4 / Degradation of DVL / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Dectin-1 mediated noncanonical NF-kB signaling / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / Hh mutants are degraded by ERAD / lipopolysaccharide binding / Degradation of AXIN / Degradation of GLI1 by the proteasome / Activation of NF-kappaB in B cells / Hedgehog ligand biogenesis / Defective CFTR causes cystic fibrosis / transcription coregulator activity / Negative regulation of NOTCH4 signaling / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / G2/M Checkpoints / Vif-mediated degradation of APOBEC3G / Autodegradation of the E3 ubiquitin ligase COP1 / Hedgehog 'on' state / Regulation of RUNX3 expression and activity / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / P-body / MAPK6/MAPK4 signaling / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / response to virus / Degradation of beta-catenin by the destruction complex / ABC-family proteins mediated transport / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / response to organic cyclic compound / CDK-mediated phosphorylation and removal of Cdc6 / CLEC7A (Dectin-1) signaling / SCF(Skp2)-mediated degradation of p27/p21 / Regulation of expression of SLITs and ROBOs / nuclear matrix / FCERI mediated NF-kB activation / Regulation of PTEN stability and activity / Interleukin-1 signaling / Orc1 removal from chromatin / Regulation of RAS by GAPs / Separation of Sister Chromatids / Regulation of RUNX2 expression and activity / The role of GTSE1 in G2/M progression after G2 checkpoint / UCH proteinases / positive regulation of interleukin-6 production / KEAP1-NFE2L2 pathway / Antigen processing: Ubiquitination & Proteasome degradation / Downstream TCR signaling / RUNX1 regulates transcription of genes involved in differentiation of HSCs / Neddylation / peptidase activity / positive regulation of NF-kappaB transcription factor activity / ER-Phagosome pathway / regulation of inflammatory response / postsynapse / proteasome-mediated ubiquitin-dependent protein catabolic process / secretory granule lumen / endopeptidase activity / response to oxidative stress / ficolin-1-rich granule lumen / response to lipopolysaccharide / nuclear body / ribosome / Ub-specific processing proteases / cadherin binding / negative regulation of gene expression 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Singh K / Brunner H / Grishkovskaya I / de Almeida M / Hinterndorfer M / Zuber J / Haselbach D | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: AKIRIN2 controls the nuclear import of proteasomes in vertebrates. 著者: Melanie de Almeida / Matthias Hinterndorfer / Hanna Brunner / Irina Grishkovskaya / Kashish Singh / Alexander Schleiffer / Julian Jude / Sumit Deswal / Robert Kalis / Milica Vunjak / Thomas ...著者: Melanie de Almeida / Matthias Hinterndorfer / Hanna Brunner / Irina Grishkovskaya / Kashish Singh / Alexander Schleiffer / Julian Jude / Sumit Deswal / Robert Kalis / Milica Vunjak / Thomas Lendl / Richard Imre / Elisabeth Roitinger / Tobias Neumann / Susanne Kandolf / Michael Schutzbier / Karl Mechtler / Gijs A Versteeg / David Haselbach / Johannes Zuber /     要旨: Protein expression and turnover are controlled through a complex interplay of transcriptional, post-transcriptional and post-translational mechanisms to enable spatial and temporal regulation of ...Protein expression and turnover are controlled through a complex interplay of transcriptional, post-transcriptional and post-translational mechanisms to enable spatial and temporal regulation of cellular processes. To systematically elucidate such gene regulatory networks, we developed a CRISPR screening assay based on time-controlled Cas9 mutagenesis, intracellular immunostaining and fluorescence-activated cell sorting that enables the identification of regulatory factors independent of their effects on cellular fitness. We pioneered this approach by systematically probing the regulation of the transcription factor MYC, a master regulator of cell growth. Our screens uncover a highly conserved protein, AKIRIN2, that is essentially required for nuclear protein degradation. We found that AKIRIN2 forms homodimers that directly bind to fully assembled 20S proteasomes to mediate their nuclear import. During mitosis, proteasomes are excluded from condensing chromatin and re-imported into newly formed daughter nuclei in a highly dynamic, AKIRIN2-dependent process. Cells undergoing mitosis in the absence of AKIRIN2 become devoid of nuclear proteasomes, rapidly causing accumulation of MYC and other nuclear proteins. Collectively, our study reveals a dedicated pathway controlling the nuclear import of proteasomes in vertebrates and establishes a scalable approach to decipher regulators in essential cellular processes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12341.map.gz emd_12341.map.gz | 150.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12341-v30.xml emd-12341-v30.xml emd-12341.xml emd-12341.xml | 35 KB 35 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_12341.png emd_12341.png | 72.5 KB | ||
| Filedesc metadata |  emd-12341.cif.gz emd-12341.cif.gz | 9.1 KB | ||
| その他 |  emd_12341_additional_1.map.gz emd_12341_additional_1.map.gz | 141.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12341 http://ftp.pdbj.org/pub/emdb/structures/EMD-12341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12341 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12341_validation.pdf.gz emd_12341_validation.pdf.gz | 359 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12341_full_validation.pdf.gz emd_12341_full_validation.pdf.gz | 358.5 KB | 表示 | |
| XML形式データ |  emd_12341_validation.xml.gz emd_12341_validation.xml.gz | 6.9 KB | 表示 | |
| CIF形式データ |  emd_12341_validation.cif.gz emd_12341_validation.cif.gz | 8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12341 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12341 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12341 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12341 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7nhtMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10752 (タイトル: Single particle cryo EM Data of a human Akirin2 proteasome complex EMPIAR-10752 (タイトル: Single particle cryo EM Data of a human Akirin2 proteasome complexData size: 2.0 TB Data #1: Unaligned Multiframe micrographs of Akirin2 bound to the human proteasome [micrographs - multiframe] Data #2: Motion corrected files dose weighted and summed as well as without dose weigthening [micrographs - single frame] Data #3: Extracted particles of an Akirin2 bound proteasome [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12341.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12341.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | denoised map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
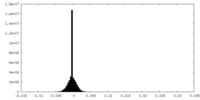
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpend map
| ファイル | emd_12341_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpend map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
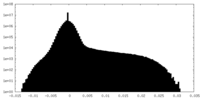
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Akirin2 bound to 20S proteasome
+超分子 #1: Akirin2 bound to 20S proteasome
+超分子 #2: 20S proteasome
+超分子 #3: Akirin2
+分子 #1: Proteasome subunit alpha type-2
+分子 #2: Proteasome subunit alpha type-4
+分子 #3: Proteasome subunit alpha type-7
+分子 #4: Proteasome subunit alpha type-5
+分子 #5: Proteasome subunit alpha type-1
+分子 #6: Proteasome subunit alpha type-3
+分子 #7: Proteasome subunit alpha type-6
+分子 #8: Proteasome subunit beta type-7
+分子 #9: Proteasome subunit beta type-3
+分子 #10: Proteasome subunit beta type-2
+分子 #11: Proteasome subunit beta type-5
+分子 #12: Proteasome subunit beta type-1
+分子 #13: Proteasome subunit beta type-4
+分子 #14: Proteasome subunit beta type-6
+分子 #15: Akirin-2
+分子 #16: POTASSIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R3.5/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 4595 / 平均露光時間: 1.0 sec. / 平均電子線量: 33.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X