+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1aze | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | NMR STRUCTURE OF THE COMPLEX BETWEEN THE C32S-Y7V MUTANT OF THE NSH3 DOMAIN OF GRB2 WITH A PEPTIDE FROM SOS, 10 STRUCTURES | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | COMPLEX (ADAPTOR PROTEIN/PEPTIDE) / COMPLEX (ADAPTOR PROTEIN-PEPTIDE) / SH3 DOMAIN / GUANINE-NUCLEOTIDE RELEASING FACTOR / COMPLEX (ADAPTOR PROTEIN-PEPTIDE) complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / NRAGE signals death through JNK / G alpha (12/13) signalling events / rhabdomere microvillus membrane / guanyl-nucleotide exchange factor activity => GO:0005085 / guanyl-nucleotide exchange factor activity => GO:0005085 / photoreceptor cell fate determination / SOS-mediated signalling / Signaling by SCF-KIT ...: / : / NRAGE signals death through JNK / G alpha (12/13) signalling events / rhabdomere microvillus membrane / guanyl-nucleotide exchange factor activity => GO:0005085 / guanyl-nucleotide exchange factor activity => GO:0005085 / photoreceptor cell fate determination / SOS-mediated signalling / Signaling by SCF-KIT / Regulation of KIT signaling / Signalling to RAS / DAP12 signaling / SHC-related events triggered by IGF1R / Role of LAT2/NTAL/LAB on calcium mobilization / FCERI mediated Ca+2 mobilization / SHC-mediated cascade:FGFR1 / FRS-mediated FGFR1 signaling / SHC-mediated cascade:FGFR2 / FRS-mediated FGFR2 signaling / SHC-mediated cascade:FGFR3 / FRS-mediated FGFR3 signaling / FRS-mediated FGFR4 signaling / SHC-mediated cascade:FGFR4 / Signal attenuation / Insulin receptor signalling cascade / FCERI mediated MAPK activation / SHC1 events in ERBB4 signaling / GRB2 events in EGFR signaling / SHC1 events in EGFR signaling / GRB2 events in ERBB2 signaling / EGFR Transactivation by Gastrin / torso signaling pathway / RAF/MAP kinase cascade / tracheal outgrowth, open tracheal system / R7 cell fate commitment / sevenless signaling pathway / epithelial cell migration, open tracheal system / imaginal disc-derived leg morphogenesis / guanyl-nucleotide exchange factor adaptor activity / Grb2-EGFR complex / branching involved in labyrinthine layer morphogenesis / STAT5 Activation / COP9 signalosome / neurotrophin TRKA receptor binding / Activated NTRK2 signals through PI3K / MET receptor recycling / transmembrane receptor protein tyrosine kinase adaptor activity / Signaling by cytosolic FGFR1 fusion mutants / Interleukin-15 signaling / positive regulation of Ras protein signal transduction / MET activates PTPN11 / axon midline choice point recognition / vesicle membrane / MET activates RAP1 and RAC1 / Costimulation by the CD28 family / Signaling by LTK / CD28 dependent Vav1 pathway / MET activates PI3K/AKT signaling / Signal regulatory protein family interactions / natural killer cell mediated cytotoxicity / Regulation of KIT signaling / epidermal growth factor receptor binding / negative regulation of G1/S transition of mitotic cell cycle / small GTPase-mediated signal transduction / positive regulation of actin filament polymerization / PI-3K cascade:FGFR3 / STAT5 activation downstream of FLT3 ITD mutants / PI-3K cascade:FGFR2 / PI-3K cascade:FGFR4 / endodermal cell differentiation / PI-3K cascade:FGFR1 / positive regulation of Rac protein signal transduction / extrinsic component of cytoplasmic side of plasma membrane / GRB2:SOS provides linkage to MAPK signaling for Integrins / regulation of MAPK cascade / RHOU GTPase cycle / PI3K events in ERBB2 signaling / SOS-mediated signalling / positive regulation of cell size / RET signaling / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / Interleukin-3, Interleukin-5 and GM-CSF signaling / insulin receptor substrate binding / PI3K Cascade / SHC1 events in ERBB4 signaling / Signalling to RAS / RHO GTPases Activate WASPs and WAVEs / signal transduction in response to DNA damage / fibroblast growth factor receptor signaling pathway / SHC-related events triggered by IGF1R / GAB1 signalosome / Activated NTRK2 signals through FRS2 and FRS3 / Role of LAT2/NTAL/LAB on calcium mobilization / Interleukin receptor SHC signaling / Signal attenuation / SHC-mediated cascade:FGFR3 / vascular endothelial growth factor receptor signaling pathway / MET activates RAS signaling 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / SIMULATED ANNEALING ENERGY MINIMIZATION | ||||||
 データ登録者 データ登録者 | Vidal, M. / Gincel, E. / Goudreau, N. / Cornille, F. / Parker, F. / Duchesne, M. / Tocque, B. / Garbay, C. / Roques, B.P. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1999 ジャーナル: J.Mol.Biol. / 年: 1999タイトル: Molecular and cellular analysis of Grb2 SH3 domain mutants: interaction with Sos and dynamin. 著者: Vidal, M. / Goudreau, N. / Cornille, F. / Cussac, D. / Gincel, E. / Garbay, C. #1:  ジャーナル: Nat.Struct.Biol. / 年: 1994 ジャーナル: Nat.Struct.Biol. / 年: 1994タイトル: NMR Structure of the N-Terminal SH3 Domain of Grb2 and its Complex with a Proline-Rich Peptide from SOS 著者: Goudreau, N. / Cornille, F. / Duchesne, M. / Parker, F. / Tocque, B. / Garbay, C. / Roques, B.P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1aze.cif.gz 1aze.cif.gz | 215.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1aze.ent.gz pdb1aze.ent.gz | 177.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1aze.json.gz 1aze.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/az/1aze https://data.pdbj.org/pub/pdb/validation_reports/az/1aze ftp://data.pdbj.org/pub/pdb/validation_reports/az/1aze ftp://data.pdbj.org/pub/pdb/validation_reports/az/1aze | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 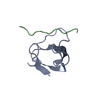
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6477.309 Da / 分子数: 1 / 断片: N-TERMINAL SH3 DOMAIN, RESIDUES 1 - 55 / 変異: Y7V, C32S / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GRB2 / 器官: FRUIT / 参照: UniProt: P62993 Homo sapiens (ヒト) / 遺伝子: GRB2 / 器官: FRUIT / 参照: UniProt: P62993 |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 1173.432 Da / 分子数: 1 / 断片: BINDING SITE IN H-SOS, PEPTIDE VPPPVPPRRR / 由来タイプ: 組換発現 由来: (組換発現)  参照: UniProt: P26675 |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: H2O, PHOSPHATE BUFFER 20MM, NACL 100MM |
|---|---|
| 試料状態 | イオン強度: NACL 100mM / pH: 6 / 圧: 1 atm / 温度: 293 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AMX600 / 製造業者: Bruker / モデル: AMX600 / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: SIMULATED ANNEALING ENERGY MINIMIZATION / ソフトェア番号: 1 | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: BEST ENERGY, LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 10 | ||||||||||||
| 精密化 | *PLUS | ||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj
























