+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9851 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | membrane structure | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | alpha/beta translocator / membrane protein complex / Protein import / mitochondria / TRANSLOCASE | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein transmembrane transport / protein targeting to mitochondrion / porin activity / protein insertion into mitochondrial outer membrane / pore complex / protein transmembrane transporter activity / monoatomic ion transport ...mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein transmembrane transport / protein targeting to mitochondrion / porin activity / protein insertion into mitochondrial outer membrane / pore complex / protein transmembrane transporter activity / monoatomic ion transport / mitochondrial intermembrane space / mitochondrial outer membrane / mitochondrion / cytosol 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.81 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Araiso Y / Tsutsumi A | ||||||||||||||||||
| 資金援助 |  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structure of the mitochondrial import gate reveals distinct preprotein paths. 著者: Yuhei Araiso / Akihisa Tsutsumi / Jian Qiu / Kenichiro Imai / Takuya Shiota / Jiyao Song / Caroline Lindau / Lena-Sophie Wenz / Haruka Sakaue / Kaori Yunoki / Shin Kawano / Junko Suzuki / ...著者: Yuhei Araiso / Akihisa Tsutsumi / Jian Qiu / Kenichiro Imai / Takuya Shiota / Jiyao Song / Caroline Lindau / Lena-Sophie Wenz / Haruka Sakaue / Kaori Yunoki / Shin Kawano / Junko Suzuki / Marilena Wischnewski / Conny Schütze / Hirotaka Ariyama / Toshio Ando / Thomas Becker / Trevor Lithgow / Nils Wiedemann / Nikolaus Pfanner / Masahide Kikkawa / Toshiya Endo /      要旨: The translocase of the outer mitochondrial membrane (TOM) is the main entry gate for proteins. Here we use cryo-electron microscopy to report the structure of the yeast TOM core complex at 3.8-Å ...The translocase of the outer mitochondrial membrane (TOM) is the main entry gate for proteins. Here we use cryo-electron microscopy to report the structure of the yeast TOM core complex at 3.8-Å resolution. The structure reveals the high-resolution architecture of the translocator consisting of two Tom40 β-barrel channels and α-helical transmembrane subunits, providing insight into critical features that are conserved in all eukaryotes. Each Tom40 β-barrel is surrounded by small TOM subunits, and tethered by two Tom22 subunits and one phospholipid. The N-terminal extension of Tom40 forms a helix inside the channel; mutational analysis reveals its dual role in early and late steps in the biogenesis of intermembrane-space proteins in cooperation with Tom5. Each Tom40 channel possesses two precursor exit sites. Tom22, Tom40 and Tom7 guide presequence-containing preproteins to the exit in the middle of the dimer, whereas Tom5 and the Tom40 N extension guide preproteins lacking a presequence to the exit at the periphery of the dimer. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9851.map.gz emd_9851.map.gz | 16.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9851-v30.xml emd-9851-v30.xml emd-9851.xml emd-9851.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9851_fsc.xml emd_9851_fsc.xml | 8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9851.png emd_9851.png | 114.7 KB | ||
| マスクデータ |  emd_9851_msk_1.map emd_9851_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-9851.cif.gz emd-9851.cif.gz | 6.5 KB | ||
| その他 |  emd_9851_half_map_1.map.gz emd_9851_half_map_1.map.gz emd_9851_half_map_2.map.gz emd_9851_half_map_2.map.gz | 19.7 MB 19.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9851 http://ftp.pdbj.org/pub/emdb/structures/EMD-9851 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9851 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9851 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9851_validation.pdf.gz emd_9851_validation.pdf.gz | 731.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9851_full_validation.pdf.gz emd_9851_full_validation.pdf.gz | 731.2 KB | 表示 | |
| XML形式データ |  emd_9851_validation.xml.gz emd_9851_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_9851_validation.cif.gz emd_9851_validation.cif.gz | 17.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9851 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9851 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9851 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9851 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6jnfMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10332 (タイトル: Cryo-EM structure of the translocator of the outer mitochondrial membrane EMPIAR-10332 (タイトル: Cryo-EM structure of the translocator of the outer mitochondrial membraneData size: 1.9 TB Data #1: Unaligned multi-frame micrographs of yeast TOM complex [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9851.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9851.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2915 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
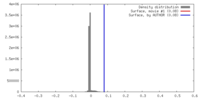
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9851_msk_1.map emd_9851_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
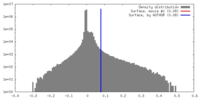
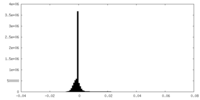
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_9851_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
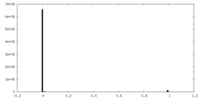
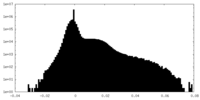
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_9851_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
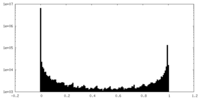
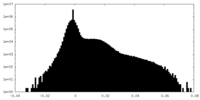
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TOM complex
| 全体 | 名称: TOM complex |
|---|---|
| 要素 |
|
-超分子 #1: TOM complex
| 超分子 | 名称: TOM complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Mitochondrial import receptor subunit TOM40
| 分子 | 名称: Mitochondrial import receptor subunit TOM40 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.071141 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAPTPLAEA SQIPTIPALS PLTAKQSKGN FFSSNPISSF VVDTYKQLHS HRQSLELVNP GTVENLNKEV SRDVFLSQYF FTGLRADLN KAFSMNPAFQ TSHTFSIGSQ ALPKYAFSAL FANDNLFAQG NIDNDLSVSG RLNYGWDKKN ISKVNLQISD G QPTMCQLE ...文字列: MSAPTPLAEA SQIPTIPALS PLTAKQSKGN FFSSNPISSF VVDTYKQLHS HRQSLELVNP GTVENLNKEV SRDVFLSQYF FTGLRADLN KAFSMNPAFQ TSHTFSIGSQ ALPKYAFSAL FANDNLFAQG NIDNDLSVSG RLNYGWDKKN ISKVNLQISD G QPTMCQLE QDYQASDFSV NVKTLNPSFS EKGEFTGVAV ASFLQSVTPQ LALGLETLYS RTDGSAPGDA GVSYLTRYVS KK QDWIFSG QLQANGALIA SLWRKVAQNV EAGIETTLQA GMVPITDPLM GTPIGIQPTV EGSTTIGAKY EYRQSVYRGT LDS NGKVAC FLERKVLPTL SVLFCGEIDH FKNDTKIGCG LQFETAGNQE LLMLQQGLDA DGNPLQALPQ L UniProtKB: Mitochondrial import receptor subunit TOM40 |
-分子 #2: Mitochondrial import receptor subunit TOM7
| 分子 | 名称: Mitochondrial import receptor subunit TOM7 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 6.876955 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSFLPSFILS DESKERISKI LTLTHNVAHY GWIPFVLYLG WAHTSNRPNF LNLLSPLPSV UniProtKB: Mitochondrial import receptor subunit TOM7 |
-分子 #3: Mitochondrial import receptor subunit TOM22
| 分子 | 名称: Mitochondrial import receptor subunit TOM22 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 18.481139 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVELTEIKDD VVQLDEPQFS RNQAIVEEKA SATNNDVVDD EDDSDSDFED EFDENETLLD RIVALKDIVP PGKRQTISNF FGFTSSFVR NAFTKSGNLA WTLTTTALLL GVPLSLSILA EQQLIEMEKT FDLQSDANNI LAQGEKDAAA TANGSPGHHH H HHHHHH UniProtKB: Mitochondrial import receptor subunit TOM22 |
-分子 #4: Mitochondrial import receptor subunit TOM5
| 分子 | 名称: Mitochondrial import receptor subunit TOM5 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 5.993924 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFGLPQQEVS EEEKRAHQEQ TEKTLKQAAY VAAFLWVSPM IWHLVKKQWK UniProtKB: Mitochondrial import receptor subunit TOM5 |
-分子 #5: Mitochondrial import receptor subunit TOM6
| 分子 | 名称: Mitochondrial import receptor subunit TOM6 / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 6.41046 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDGMFAMPGA AAGAASPQQP KSRFQAFKES PLYTIALNGA FFVAGVAFIQ SPLMDMLAPQ L UniProtKB: Mitochondrial import receptor subunit TOM6 |
-分子 #6: (2R)-3-{[(S)-(2-aminoethoxy)(hydroxy)phosphoryl]oxy}-2-(tetradeca...
| 分子 | 名称: (2R)-3-{[(S)-(2-aminoethoxy)(hydroxy)phosphoryl]oxy}-2-(tetradecanoyloxy)propyl tetradecanoate タイプ: ligand / ID: 6 / コピー数: 1 / 式: 46E |
|---|---|
| 分子量 | 理論値: 635.853 Da |
| Chemical component information |  ChemComp-46E: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)