登録情報 データベース : EMDB / ID : EMD-9785タイトル Cryo-EM structure of Xanthomonos oryzae transcription elongation complex with NusA and the bacteriophage protein P7 複合体 : Xoo transcription elongation complex with P7 and NusA (P7-NusA-TEC)タンパク質・ペプチド : x 6種DNA : x 2種RNA : x 1種リガンド : x 2種 / / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
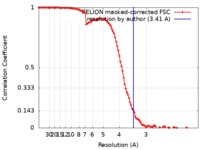
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Xanthomonas oryzae pv. oryzae (バクテリア) / Xanthomonas oryzae pv. oryzae (strain PXO99A) (バクテリア) / Xanthomonas oryzae pv. oryzae PXO99A (バクテリア) / / synthetic construct (人工物) 手法 / / 解像度 : 3.41 Å You LL / Zhang Y 資金援助 Organization Grant number 国 National Natural Science Foundation of China 31822001
ジャーナル : Nat Commun / 年 : 2019タイトル : Structural basis for transcription antitermination at bacterial intrinsic terminator.著者 : Linlin You / Jing Shi / Liqiang Shen / Lingting Li / Chengli Fang / Chengzhi Yu / Wenbo Cheng / Yu Feng / Yu Zhang / 要旨 : Bacteriophages typically hijack the host bacterial transcriptional machinery to regulate their own gene expression and that of the host bacteria. The structural basis for bacteriophage protein- ... Bacteriophages typically hijack the host bacterial transcriptional machinery to regulate their own gene expression and that of the host bacteria. The structural basis for bacteriophage protein-mediated transcription regulation-in particular transcription antitermination-is largely unknown. Here we report the 3.4 Å and 4.0 Å cryo-EM structures of two bacterial transcription elongation complexes (P7-NusA-TEC and P7-TEC) comprising the bacteriophage protein P7, a master host-transcription regulator encoded by bacteriophage Xp10 of the rice pathogen Xanthomonas oryzae pv. Oryzae (Xoo) and discuss the mechanisms by which P7 modulates the host bacterial RNAP. The structures together with biochemical evidence demonstrate that P7 prevents transcription termination by plugging up the RNAP RNA-exit channel and impeding RNA-hairpin formation at the intrinsic terminator. Moreover, P7 inhibits transcription initiation by restraining RNAP-clamp motions. Our study reveals the structural basis for transcription antitermination by phage proteins and provides insights into bacterial transcription regulation. 履歴 登録 2019年1月22日 - ヘッダ(付随情報) 公開 2019年7月17日 - マップ公開 2019年7月17日 - 更新 2024年3月27日 - 現状 2024年3月27日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Xanthomonas oryzae pv. oryzae (バクテリア) /
Xanthomonas oryzae pv. oryzae (バクテリア) /  Xanthomonas oryzae pv. oryzae (strain PXO99A) (バクテリア) /
Xanthomonas oryzae pv. oryzae (strain PXO99A) (バクテリア) /  Xanthomonas oryzae pv. oryzae PXO99A (バクテリア) /
Xanthomonas oryzae pv. oryzae PXO99A (バクテリア) /  Xanthomonas virus Xp10 (ウイルス) / synthetic construct (人工物)
Xanthomonas virus Xp10 (ウイルス) / synthetic construct (人工物) データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2019
ジャーナル: Nat Commun / 年: 2019
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_9785.map.gz
emd_9785.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-9785-v30.xml
emd-9785-v30.xml emd-9785.xml
emd-9785.xml EMDBヘッダ
EMDBヘッダ emd_9785_fsc.xml
emd_9785_fsc.xml FSCデータファイル
FSCデータファイル emd_9785.png
emd_9785.png emd-9785.cif.gz
emd-9785.cif.gz emd_9785_half_map_1.map.gz
emd_9785_half_map_1.map.gz emd_9785_half_map_2.map.gz
emd_9785_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-9785
http://ftp.pdbj.org/pub/emdb/structures/EMD-9785 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9785
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9785 emd_9785_validation.pdf.gz
emd_9785_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_9785_full_validation.pdf.gz
emd_9785_full_validation.pdf.gz emd_9785_validation.xml.gz
emd_9785_validation.xml.gz emd_9785_validation.cif.gz
emd_9785_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9785
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9785 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9785
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9785 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_9785.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_9785.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)