+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9614 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM reconstruction of Mycobacterium Mfd Oligomer (Dodecamer) | |||||||||
 マップデータ マップデータ | 1.5 sigma | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) | |||||||||
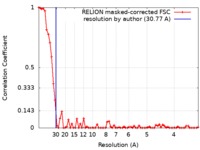
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 30.77 Å | |||||||||
 データ登録者 データ登録者 | Vinayak B / Putta S / Rao DN / Nagaraja V / Natesh R | |||||||||
| 資金援助 |  インド, 1件 インド, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural basis for nucleotide-mediated remodelling mechanism of Mycobacterium Mfd 著者: Putta S / Prabha S / Bhat V / Fox GC / Walsh MA / Rao DN / Nagaraja V / Natesh R | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9614.map.gz emd_9614.map.gz | 19.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9614-v30.xml emd-9614-v30.xml emd-9614.xml emd-9614.xml | 13.5 KB 13.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9614_fsc.xml emd_9614_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9614.png emd_9614.png | 42.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9614 http://ftp.pdbj.org/pub/emdb/structures/EMD-9614 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9614 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9614 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9614_validation.pdf.gz emd_9614_validation.pdf.gz | 78.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9614_full_validation.pdf.gz emd_9614_full_validation.pdf.gz | 77.7 KB | 表示 | |
| XML形式データ |  emd_9614_validation.xml.gz emd_9614_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9614 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9614 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9614 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9614 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9614.map.gz / 形式: CCP4 / 大きさ: 26.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9614.map.gz / 形式: CCP4 / 大きさ: 26.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 1.5 sigma | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterium tuberculosis Mfd Oligomer (Dodecamer)
| 全体 | 名称: Mycobacterium tuberculosis Mfd Oligomer (Dodecamer) |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacterium tuberculosis Mfd Oligomer (Dodecamer)
| 超分子 | 名称: Mycobacterium tuberculosis Mfd Oligomer (Dodecamer) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Oligomeric MtbMfd fraction from Size Exclusion Chromatography. |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) / 株: H37Rv Mycobacterium tuberculosis H37Rv (結核菌) / 株: H37Rv |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 1.596 MDa |
-分子 #1: Mycobactetrium tuberculosis Mfd Oligomer (Dodecamer)
| 分子 | 名称: Mycobactetrium tuberculosis Mfd Oligomer (Dodecamer) タイプ: protein_or_peptide / ID: 1 / 詳細: MtbMfd oligomer peak from SEC / 光学異性体: LEVO / EC番号: ec: 3.4.6.- |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MTAPGPACS DTPIAGLVEL ALSAPTFQQL MQRAGGRPDE LTLIAPASAR LLVASALARQ GPLLVVTATG R EADDLAAE LRGVFGDAVA LLPSWETLPH ERLSPGVDTV GTRLMALRRL AHPDDAQLGP PLGVVVTSVR SL LQPMTPQ ...文字列: MGSSHHHHHH SSGLVPRGSH MTAPGPACS DTPIAGLVEL ALSAPTFQQL MQRAGGRPDE LTLIAPASAR LLVASALARQ GPLLVVTATG R EADDLAAE LRGVFGDAVA LLPSWETLPH ERLSPGVDTV GTRLMALRRL AHPDDAQLGP PLGVVVTSVR SL LQPMTPQ LGMMEPLTLT VGDESPFDGV VARLVELAYT RVDMVGRRGE FAVRGGILDI FAPTAEHPVR VEF WGDEIT EMRMFSVADQ RSIPEIDIHT LVAFACRELL LSEDVRARAA QLAARHPAAE STVTGSASDM LAKL AEGIA VDGMEAVLPV LWSDGHALLT DQLPDGTPVL VCDPEKVRTR AADLIRTGRE FLEASWSVAA LGTAE NQAP VDVEQLGGSG FVELDQVRAA AARTGHPWWT LSQLSDESAI ELDVRAAPSA RGHQRDIDEI FAMLRA HIA TGGYAALVAP GTGTAHRVVE RLSESDTPAG MLDPGQAPKP GVVGVLQGPL RDGVIIPGAN LVVITET DL TGSRVSAAEG KRLAAKRRNI VDPLALTAGD LVVHDQHGIG RFVEMVERTV GGARREYLVL EYASAKRG G GAKNTDKLYV PMDSLDQLSR YVGGQAPALS RLGGSDWANT KTKARRAVRE IAGELVSLYA KRQASPGHA FSPDTPWQAE LEDAFGFTET VDQLTAIEEV KADMEKPIPM DRVICGDVGY GKTEIAVRAA FKAVQDGKQV AVLVPTTLL ADQHLQTFGE RMSGFPVTIK GLSRFTDAAE SRAVIDGLAD GSVDIVIGTH RLLQTGVRWK D LGLVVVDE EQRFGVEHKE HIKSLRTHVD VLTMSATPIP RTLEMSLAGI REMSTILTPP EERYPVLTYV GP HDDKQIA AALRRELLRD GQAFYVHNRV SSIDAAAARV RELVPEARVV VAHGQMPEDL LETTVQRFWN REH DILVCT TIVETGLDIS NANTLIVERA DTFGLSQLHQ LRGRVGRSRE RGYAYFLYPP QVPLTETAYD RLAT IAQNN ELGAGMAVAL KDLEIRGAGN VLGIEQSGHV AGVGFDLYVR LVGEALETYR DAYRAAADGQ TVRTA EEPK DVRIDLPVDA HLPPDYIASD RLRLEGYRRL AAASSDREVA AVVDELTDRY GALPEPARRL AAVARL RLL CRGSGITDVT AASAATVRLS PLTLPDSAQV RLKRMYPGAH YRATTATVQV PIPRAGGLGA PRIRDVE LV QMVADLITAL AGKPRQHIGI TNPSPPGEDG RGRNTTIKER QP |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20mM Tris pH8.0, 500mM NaCl, 10mM beta mercaptoethanol |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate 詳細: Negatively stained EM specimen were prepared by stained with 2% uranyl acetate for one minute and air dried. |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS 詳細: negative stain specimen was prepared by fishing method without Glow Discharge |
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1200EXII |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ORIUS SC600 (2.7k x 2.7k) 撮影したグリッド数: 1 / 実像数: 100 / 平均露光時間: 1.0 sec. / 平均電子線量: 10.0 e/Å2 / 詳細: Images were collected on CCD. |
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 倍率(補正後): 52974 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 3.5 mm / 倍率(公称値): 40000 |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 詳細 | Initial manually moved into the EM map and then Rigid body fitted using 43 symmetry 12 mer as single rigid body into the EM map using Chimera. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)