+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8091 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of GluA2/3 AMPA receptor heterotetramer (model II) | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AMPA glutamate receptor / Signaling protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Trafficking of AMPA receptors / Synaptic adhesion-like molecules / parallel fiber to Purkinje cell synapse / protein heterotetramerization / spine synapse / dendritic spine neck / dendritic spine head / Activation of AMPA receptors / perisynaptic space / AMPA glutamate receptor activity ...Trafficking of AMPA receptors / Synaptic adhesion-like molecules / parallel fiber to Purkinje cell synapse / protein heterotetramerization / spine synapse / dendritic spine neck / dendritic spine head / Activation of AMPA receptors / perisynaptic space / AMPA glutamate receptor activity / ligand-gated monoatomic cation channel activity / Trafficking of GluR2-containing AMPA receptors / response to lithium ion / extracellularly glutamate-gated ion channel activity / immunoglobulin binding / AMPA glutamate receptor complex / kainate selective glutamate receptor activity / ionotropic glutamate receptor complex / cellular response to glycine / asymmetric synapse / regulation of receptor recycling / Unblocking of NMDA receptors, glutamate binding and activation / glutamate receptor binding / positive regulation of synaptic transmission / extracellular ligand-gated monoatomic ion channel activity / glutamate-gated receptor activity / synaptic cleft / response to fungicide / glutamate-gated calcium ion channel activity / presynaptic active zone membrane / regulation of synaptic transmission, glutamatergic / ionotropic glutamate receptor binding / somatodendritic compartment / dendrite membrane / cellular response to brain-derived neurotrophic factor stimulus / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / cytoskeletal protein binding / dendrite cytoplasm / ionotropic glutamate receptor signaling pathway / SNARE binding / dendritic shaft / synaptic transmission, glutamatergic / synaptic membrane / long-term synaptic potentiation / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / PDZ domain binding / protein tetramerization / postsynaptic density membrane / establishment of protein localization / modulation of chemical synaptic transmission / Schaffer collateral - CA1 synapse / terminal bouton / receptor internalization / cerebral cortex development / synaptic vesicle membrane / synaptic vesicle / presynapse / signaling receptor activity / presynaptic membrane / amyloid-beta binding / growth cone / scaffold protein binding / chemical synaptic transmission / perikaryon / postsynaptic membrane / protein homotetramerization / dendritic spine / postsynaptic density / neuron projection / axon / neuronal cell body / glutamatergic synapse / dendrite / synapse / protein-containing complex binding / protein kinase binding / cell surface / endoplasmic reticulum / protein-containing complex / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 10.31 Å | |||||||||
 データ登録者 データ登録者 | Herguedas B / Garcia-Nafria J | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2016 ジャーナル: Science / 年: 2016タイトル: Structure and organization of heteromeric AMPA-type glutamate receptors. 著者: Beatriz Herguedas / Javier García-Nafría / Ondrej Cais / Rafael Fernández-Leiro / James Krieger / Hinze Ho / Ingo H Greger /  要旨: AMPA-type glutamate receptors (AMPARs), which are central mediators of rapid neurotransmission and synaptic plasticity, predominantly exist as heteromers of the subunits GluA1 to GluA4. Here we ...AMPA-type glutamate receptors (AMPARs), which are central mediators of rapid neurotransmission and synaptic plasticity, predominantly exist as heteromers of the subunits GluA1 to GluA4. Here we report the first AMPAR heteromer structures, which deviate substantially from existing GluA2 homomer structures. Crystal structures of the GluA2/3 and GluA2/4 N-terminal domains reveal a novel compact conformation with an alternating arrangement of the four subunits around a central axis. This organization is confirmed by cysteine cross-linking in full-length receptors, and it permitted us to determine the structure of an intact GluA2/3 receptor by cryogenic electron microscopy. Two models in the ligand-free state, at resolutions of 8.25 and 10.3 angstroms, exhibit substantial vertical compression and close associations between domain layers, reminiscent of N-methyl-D-aspartate receptors. Model 1 resembles a resting state and model 2 a desensitized state, thus providing snapshots of gating transitions in the nominal absence of ligand. Our data reveal organizational features of heteromeric AMPARs and provide a framework to decipher AMPAR architecture and signaling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8091.map.gz emd_8091.map.gz | 2.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8091-v30.xml emd-8091-v30.xml emd-8091.xml emd-8091.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8091_fsc.xml emd_8091_fsc.xml | 5.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8091.png emd_8091.png | 94 KB | ||
| マスクデータ |  emd_8091_msk_1.map emd_8091_msk_1.map emd_8091_msk_2.map emd_8091_msk_2.map | 16.8 MB 16.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-8091.cif.gz emd-8091.cif.gz | 7.6 KB | ||
| その他 |  emd_8091_half_map_1.map.gz emd_8091_half_map_1.map.gz emd_8091_half_map_2.map.gz emd_8091_half_map_2.map.gz | 12.8 MB 12.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8091 http://ftp.pdbj.org/pub/emdb/structures/EMD-8091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8091 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8091_validation.pdf.gz emd_8091_validation.pdf.gz | 395.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8091_full_validation.pdf.gz emd_8091_full_validation.pdf.gz | 394.9 KB | 表示 | |
| XML形式データ |  emd_8091_validation.xml.gz emd_8091_validation.xml.gz | 11.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8091 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8091 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8091.map.gz / 形式: CCP4 / 大きさ: 16.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8091.map.gz / 形式: CCP4 / 大きさ: 16.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.76 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_8091_msk_1.map emd_8091_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
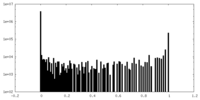
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_8091_msk_2.map emd_8091_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
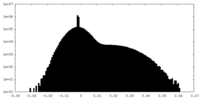
| 密度ヒストグラム |
-ハーフマップ: None
| ファイル | emd_8091_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: None
| ファイル | emd_8091_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AMPA GluA2/3 heterotetramer
| 全体 | 名称: AMPA GluA2/3 heterotetramer |
|---|---|
| 要素 |
|
-超分子 #1: AMPA GluA2/3 heterotetramer
| 超分子 | 名称: AMPA GluA2/3 heterotetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Full-length GluA2/3 heterotetramer containing the A2_N292C and A3_265C mutations |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Glutamate receptor 2
| 分子 | 名称: Glutamate receptor 2 / タイプ: protein_or_peptide / ID: 1 詳細: The sequence corresponds to mature rat GluA2 (residues 22-883, isoform Flip, edited at R/G and Q/R sites) with a Myc tag after the first residue and the N292C mutation コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 97.663188 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VEQKLISEED LSSNSIQIGG LFPRGADQEY SAFRVGMVQF STSEFRLTPH IDNLEVANSF AVTNAFCSQF SRGVYAIFGF YDKKSVNTI TSFCGTLHVS FITPSFPTDG THPFVIQMRP DLKGALLSLI EYYQWDKFAY LYDSDRGLST LQAVLDSAAE K KWQVTAIN ...文字列: VEQKLISEED LSSNSIQIGG LFPRGADQEY SAFRVGMVQF STSEFRLTPH IDNLEVANSF AVTNAFCSQF SRGVYAIFGF YDKKSVNTI TSFCGTLHVS FITPSFPTDG THPFVIQMRP DLKGALLSLI EYYQWDKFAY LYDSDRGLST LQAVLDSAAE K KWQVTAIN VGNINNDKKD ETYRSLFQDL ELKKERRVIL DCERDKVNDI VDQVITIGKH VKGYHYIIAN LGFTDGDLLK IQ FGGANVS GFQIVDYDDS LVSKFIERWS TLEEKEYPGA HTATIKYTSA LTYDAVQVMT EAFRCLRKQR IEISRRGNAG DCL ANPAVP WGQGVEIERA LKQVQVEGLS GNIKFDQNGK RINYTINIME LKTNGPRKIG YWSEVDKMVV TLTELPSGND TSGL ENKTV VVTTILESPY VMMKKNHEML EGNERYEGYC VDLAAEIAKH CGFKYKLTIV GDGKYGARDA DTKIWNGMVG ELVYG KADI AIAPLTITLV REEVIDFSKP FMSLGISIMI KKPQKSKPGV FSFLDPLAYE IWMCIVFAYI GVSVVLFLVS RFSPYE WHT EEFEDGRETQ SSESTNEFGI FNSLWFSLGA FMQQGCDISP RSLSGRIVGG VWWFFTLIII SSYTANLAAF LTVERMV SP IESAEDLSKQ TEIAYGTLDS GSTKEFFRRS KIAVFDKMWT YMRSAEPSVF VRTTAEGVAR VRKSKGKYAY LLESTMNE Y IEQRKPCDTM KVGGNLDSKG YGIATPKGSS LGTPVNLAVL KLSEQGVLDK LKNKWWYDKG ECGAKDSGSK EKTSALSLS NVAGVFYILV GGLGLAMLVA LIEFCYKSRA EAKRMKVAKN PQNINPSSSQ NSQNFATYKE GYNVYGIESV KI UniProtKB: Glutamate receptor 2 |
-分子 #2: Glutamate receptor 3
| 分子 | 名称: Glutamate receptor 3 / タイプ: protein_or_peptide / ID: 2 詳細: The sequence corresponds to the mature rat GluA3 subunit (residues 23-888, Flip isoform) with a Flag tag after the first residue and mutated at R439G and R265C コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 99.075664 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GDYKDDDDKF PNTISIGGLF MRNTVQEHSA FRFAVQLYNT NQNTTEKPFH LNYHVDHLDS SNSFSVTNAF CSQFSRGVYA IFGFYDQMS MNTLTSFCGA LHTSFVTPSF PTDADVQFVI QMRPALKGAI LSLLSYYKWE KFVYLYDTER GFSVLQAIME A AVQNNWQV ...文字列: GDYKDDDDKF PNTISIGGLF MRNTVQEHSA FRFAVQLYNT NQNTTEKPFH LNYHVDHLDS SNSFSVTNAF CSQFSRGVYA IFGFYDQMS MNTLTSFCGA LHTSFVTPSF PTDADVQFVI QMRPALKGAI LSLLSYYKWE KFVYLYDTER GFSVLQAIME A AVQNNWQV TARSVGNIKD VQEFRRIIEE MDRRQEKRYL IDCEVERINT ILEQVVILGK HSRGYHYMLA NLGFTDILLE RV MHGGANI TGFQIVNNEN PMVQQFIQRW VRLDECEFPE AKNAPLKYTS ALTHDAILVI AEAFRYLRRQ RVDVSRRGSA GDC LANPAV PWSQGIDIER ALKMVQVQGM TGNIQFDTYG RRTNYTIDVY EMKVSGSRKA GYWNEYERFV PFSDQQISND SSSS ENRTI VVTTILESPY VMYKKNHEQL EGNERYEGYC VDLAYEIAKH VGIKYKLSIV GDGKYGARDP ETKIWNGMVG ELVYG RADI AVAPLTITLV REEVIDFSKP FMSLGISIMI KKPQKSKPGV FSFLDPLAYE IWMCIVFAYI GVSVVLFLVS RFSPYE WHL EDNNEEPRDP QSPPDPPNEF GIFNSLWFSL GAFMQQGCDI SPRSLSGRIV GGVWWFFTLI IISSYTANLA AFLTVER MV SPIESAEDLA KQTEIAYGTL DSGSTKEFFR RSKIAVYEKM WSYMKSAEPS VFTKTTADGV ARVRKSKGKF AFLLESTM N EYIEQRKPCD TMKVGGNLDS KGYGVATPKG SALGTPVNLA VLKLSEQGIL DKLKNKWWYD KGECGAKDSG SKDKTSALS LSNVAGVFYI LVGGLGLAMM VALIEFCYKS RAESKRMKLT KNTQNFKPAP ATNTQNYATY REGYNVYGTE SVKI UniProtKB: Glutamate receptor 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 25 mM Tris pH 7.4, 0.25 % DDM, 150 mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Incubated for 1 minute, blotted for 3 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 2 / 実像数: 980 / 平均露光時間: 25.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 28409 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 詳細 | For GluA2 chains (A,C), 2 copies of GluA2 NTD (3HSY) and two copies of GluA2 LBD (1FTO) were fitted. For GluA3 chains (B,D), 2 copies of GluA3NTD (3O21) and two copies of GluA2 LBD (3UA8) were fitted.For the TMD region, the 4 chains of 3KG2 TMD (residues 509-544 594-629 784-817) were fitted as a rigid body. After fitting the sequence was corrected including mutations and side chains were removed. | ||||||||||||
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||||||||
| 得られたモデル |  PDB-5idf: |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)