+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7zub | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the indirubin-bound Hsp90-XAP2-AHR complex | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | GENE REGULATION / complex / nuclear receptor / chemical pollutants / detoxification / cancer | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GAF domain binding / cytosolic aryl hydrocarbon receptor complex / regulation of B cell proliferation / cellular response to 2,3,7,8-tetrachlorodibenzodioxine / HSP90-CDC37 chaperone complex / regulation of adaptive immune response / nuclear aryl hydrocarbon receptor complex / negative regulation of proteasomal protein catabolic process / Aryl hydrocarbon receptor signalling / aryl hydrocarbon receptor complex ...GAF domain binding / cytosolic aryl hydrocarbon receptor complex / regulation of B cell proliferation / cellular response to 2,3,7,8-tetrachlorodibenzodioxine / HSP90-CDC37 chaperone complex / regulation of adaptive immune response / nuclear aryl hydrocarbon receptor complex / negative regulation of proteasomal protein catabolic process / Aryl hydrocarbon receptor signalling / aryl hydrocarbon receptor complex / cellular response to molecule of bacterial origin / negative regulation of T cell mediated immune response to tumor cell / histone methyltransferase binding / dynein axonemal particle / receptor ligand inhibitor activity / Xenobiotics / protein kinase regulator activity / ATP-dependent protein binding / positive regulation of protein localization to cell surface / Phase I - Functionalization of compounds / protein folding chaperone complex / : / Respiratory syncytial virus genome replication / telomerase holoenzyme complex assembly / positive regulation of transforming growth factor beta receptor signaling pathway / blood vessel development / Uptake and function of diphtheria toxin / TPR domain binding / dendritic growth cone / E-box binding / Assembly and release of respiratory syncytial virus (RSV) virions / aryl hydrocarbon receptor binding / TFIID-class transcription factor complex binding / protein phosphatase activator activity / Sema3A PAK dependent Axon repulsion / The NLRP3 inflammasome / regulation of protein ubiquitination / HSF1-dependent transactivation / Endogenous sterols / response to unfolded protein / HSF1 activation / Attenuation phase / chaperone-mediated protein complex assembly / RHOBTB2 GTPase cycle / axonal growth cone / telomere maintenance via telomerase / Purinergic signaling in leishmaniasis infection / cis-regulatory region sequence-specific DNA binding / : / supramolecular fiber organization / DNA polymerase binding / heat shock protein binding / protein folding chaperone / peptide binding / xenobiotic metabolic process / negative regulation of proteasomal ubiquitin-dependent protein catabolic process / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / cellular response to interleukin-4 / ESR-mediated signaling / cellular response to forskolin / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / TBP-class protein binding / cellular response to cAMP / placenta development / nitric-oxide synthase regulator activity / protein maturation / peptidyl-prolyl cis-trans isomerase activity / positive regulation of cell differentiation / circadian regulation of gene expression / ATP-dependent protein folding chaperone / Hsp90 protein binding / PPARA activates gene expression / DDX58/IFIH1-mediated induction of interferon-alpha/beta / Regulation of actin dynamics for phagocytic cup formation / negative regulation of inflammatory response / response to toxic substance / histone deacetylase binding / tau protein binding / transcription coactivator binding / kinase binding / nuclear receptor activity / Chaperone Mediated Autophagy / disordered domain specific binding / sequence-specific double-stranded DNA binding / positive regulation of nitric oxide biosynthetic process / MHC class II protein complex binding / The role of GTSE1 in G2/M progression after G2 checkpoint / melanosome / unfolded protein binding / protein folding / double-stranded RNA binding / regulation of protein localization / cellular response to heat / regulation of gene expression / secretory granule lumen / transcription regulator complex / Estrogen-dependent gene expression / ficolin-1-rich granule lumen / Potential therapeutics for SARS / RNA polymerase II-specific DNA-binding transcription factor binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.85 Å | ||||||||||||
 データ登録者 データ登録者 | Gruszczyk, J. / Savva, C.G. / Lai-Kee-Him, J. / Bous, J. / Ancelin, A. / Kwong, H.S. / Grandvuillemin, L. / Bourguet, W. | ||||||||||||
| 資金援助 | European Union,  フランス, 3件 フランス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Cryo-EM structure of the agonist-bound Hsp90-XAP2-AHR cytosolic complex. 著者: Jakub Gruszczyk / Loïc Grandvuillemin / Josephine Lai-Kee-Him / Matteo Paloni / Christos G Savva / Pierre Germain / Marina Grimaldi / Abdelhay Boulahtouf / Hok-Sau Kwong / Julien Bous / ...著者: Jakub Gruszczyk / Loïc Grandvuillemin / Josephine Lai-Kee-Him / Matteo Paloni / Christos G Savva / Pierre Germain / Marina Grimaldi / Abdelhay Boulahtouf / Hok-Sau Kwong / Julien Bous / Aurélie Ancelin / Cherine Bechara / Alessandro Barducci / Patrick Balaguer / William Bourguet /    要旨: The aryl hydrocarbon receptor (AHR) is a ligand-dependent transcription factor that mediates a broad spectrum of (patho)physiological processes in response to numerous substances including ...The aryl hydrocarbon receptor (AHR) is a ligand-dependent transcription factor that mediates a broad spectrum of (patho)physiological processes in response to numerous substances including pollutants, natural products and metabolites. However, the scarcity of structural data precludes understanding of how AHR is activated by such diverse compounds. Our 2.85 Å structure of the human indirubin-bound AHR complex with the chaperone Hsp90 and the co-chaperone XAP2, reported herein, reveals a closed conformation Hsp90 dimer with AHR threaded through its lumen and XAP2 serving as a brace. Importantly, we disclose the long-awaited structure of the AHR PAS-B domain revealing a unique organisation of the ligand-binding pocket and the structural determinants of ligand-binding specificity and promiscuity of the receptor. By providing structural details of the molecular initiating event leading to AHR activation, our study rationalises almost forty years of biochemical data and provides a framework for future mechanistic studies and structure-guided drug design. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7zub.cif.gz 7zub.cif.gz | 674 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7zub.ent.gz pdb7zub.ent.gz | 555.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7zub.json.gz 7zub.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zu/7zub https://data.pdbj.org/pub/pdb/validation_reports/zu/7zub ftp://data.pdbj.org/pub/pdb/validation_reports/zu/7zub ftp://data.pdbj.org/pub/pdb/validation_reports/zu/7zub | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  14971MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 3種, 4分子 ABCD
| #1: タンパク質 | 分子量: 84213.141 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: HSP90AB1, HSP90B, HSPC2, HSPCB Homo sapiens (ヒト) / 遺伝子: HSP90AB1, HSP90B, HSPC2, HSPCB発現宿主:  参照: UniProt: P08238 #2: タンパク質 | | 分子量: 37691.047 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: AIP, XAP2 Homo sapiens (ヒト) / 遺伝子: AIP, XAP2発現宿主:  参照: UniProt: O00170 #3: タンパク質 | | 分子量: 49767.879 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: AHR, BHLHE76 Homo sapiens (ヒト) / 遺伝子: AHR, BHLHE76発現宿主:  参照: UniProt: P35869 |
|---|
-非ポリマー , 4種, 7分子 

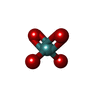
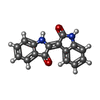

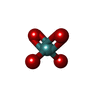

| #4: 化合物 | | #5: 化合物 | #6: 化合物 | #7: 化合物 | ChemComp-JY6 / ( | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Hsp90-XAP2-AHR complex / タイプ: COMPLEX / Entity ID: #1-#3 / 由来: RECOMBINANT | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.256 MDa / 実験値: YES | |||||||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:  | |||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7 | |||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 0.2 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: C-flat-1.2/1.3 | |||||||||||||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 295 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 800 nm / Cs: 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 3 sec. / 電子線照射量: 1.1 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 9300 |
| 電子光学装置 | エネルギーフィルター名称: GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.20_4459: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING ONLY | ||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 11546649 | ||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.85 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 678724 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj





































