+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7908 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
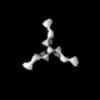
| タイトル | VIC170 Fab in complex with Ebola virus GP | |||||||||
 マップデータ マップデータ | VIC170 antibody Fab in complex with Ebola virus GP | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 22.0 Å | |||||||||
 データ登録者 データ登録者 | Turner H / Murin CD / Pallesen J / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2018 ジャーナル: Cell / 年: 2018タイトル: Systematic Analysis of Monoclonal Antibodies against Ebola Virus GP Defines Features that Contribute to Protection. 著者: Erica Ollmann Saphire / Sharon L Schendel / Marnie L Fusco / Karthik Gangavarapu / Bronwyn M Gunn / Anna Z Wec / Peter J Halfmann / Jennifer M Brannan / Andrew S Herbert / Xiangguo Qiu / ...著者: Erica Ollmann Saphire / Sharon L Schendel / Marnie L Fusco / Karthik Gangavarapu / Bronwyn M Gunn / Anna Z Wec / Peter J Halfmann / Jennifer M Brannan / Andrew S Herbert / Xiangguo Qiu / Kshitij Wagh / Shihua He / Elena E Giorgi / James Theiler / Kathleen B J Pommert / Tyler B Krause / Hannah L Turner / Charles D Murin / Jesper Pallesen / Edgar Davidson / Rafi Ahmed / M Javad Aman / Alexander Bukreyev / Dennis R Burton / James E Crowe / Carl W Davis / George Georgiou / Florian Krammer / Christos A Kyratsous / Jonathan R Lai / Cory Nykiforuk / Michael H Pauly / Pramila Rijal / Ayato Takada / Alain R Townsend / Viktor Volchkov / Laura M Walker / Cheng-I Wang / Larry Zeitlin / Benjamin J Doranz / Andrew B Ward / Bette Korber / Gary P Kobinger / Kristian G Andersen / Yoshihiro Kawaoka / Galit Alter / Kartik Chandran / John M Dye / /       要旨: Antibodies are promising post-exposure therapies against emerging viruses, but which antibody features and in vitro assays best forecast protection are unclear. Our international consortium ...Antibodies are promising post-exposure therapies against emerging viruses, but which antibody features and in vitro assays best forecast protection are unclear. Our international consortium systematically evaluated antibodies against Ebola virus (EBOV) using multidisciplinary assays. For each antibody, we evaluated epitopes recognized on the viral surface glycoprotein (GP) and secreted glycoprotein (sGP), readouts of multiple neutralization assays, fraction of virions left un-neutralized, glycan structures, phagocytic and natural killer cell functions elicited, and in vivo protection in a mouse challenge model. Neutralization and induction of multiple immune effector functions (IEFs) correlated most strongly with protection. Neutralization predominantly occurred via epitopes maintained on endosomally cleaved GP, whereas maximal IEF mapped to epitopes farthest from the viral membrane. Unexpectedly, sGP cross-reactivity did not significantly influence in vivo protection. This comprehensive dataset provides a rubric to evaluate novel antibodies and vaccine responses and a roadmap for therapeutic development for EBOV and related viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7908.map.gz emd_7908.map.gz | 967 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7908-v30.xml emd-7908-v30.xml emd-7908.xml emd-7908.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7908.png emd_7908.png | 23.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7908 http://ftp.pdbj.org/pub/emdb/structures/EMD-7908 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7908 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7908 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7908_validation.pdf.gz emd_7908_validation.pdf.gz | 77.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7908_full_validation.pdf.gz emd_7908_full_validation.pdf.gz | 76.8 KB | 表示 | |
| XML形式データ |  emd_7908_validation.xml.gz emd_7908_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7908 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7908 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7908 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7908 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7909C  7910C  7911C  7912C  7914C  7915C  7916C  7917C  7918C  7919C  7920C  7921C  7922C  7923C  7924C  7925C  7926C  7927C  7928C  7929C  7930C  7931C  7932C  7933C  7934C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7908.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7908.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | VIC170 antibody Fab in complex with Ebola virus GP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of VIC170 Fab bound to Ebola virus GP
| 全体 | 名称: Complex of VIC170 Fab bound to Ebola virus GP |
|---|---|
| 要素 |
|
-超分子 #1: Complex of VIC170 Fab bound to Ebola virus GP
| 超分子 | 名称: Complex of VIC170 Fab bound to Ebola virus GP / タイプ: complex / ID: 1 / 親要素: 0 詳細: Fab fragment generated by proteolytic cleavage of IgG antibody with papain. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  組換細胞: S2 / 組換プラスミド: pMT-puro |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: Solution was made from 10x concentration stock. | |||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: Stained using 2% uranyl formate on carbon-coated grids. | |||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | |||||||||
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 61 / 平均露光時間: 0.5 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Common lines model |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 22.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN2 (ver. 2.1) / 使用した粒子像数: 16959 |
| 初期 角度割当 | タイプ: COMMON LINE / ソフトウェア - 名称: EMAN2 |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: EMAN2 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)