+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7884 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BG505 MD64 N332-GT5 SOSIP trimer in complex with BG18-like precursor HMP1 fragment antigen binding and base-binding RM20A3 fragment antigen binding | ||||||||||||
 マップデータ マップデータ | Sharpened map | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||
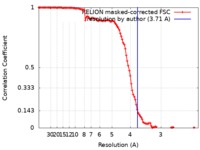
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.71 Å | ||||||||||||
 データ登録者 データ登録者 | Torres JL / Ozorowski G / Steichen JM / Schief WR / Ward AB | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2019 ジャーナル: Science / 年: 2019タイトル: A generalized HIV vaccine design strategy for priming of broadly neutralizing antibody responses. 著者: Jon M Steichen / Ying-Cing Lin / Colin Havenar-Daughton / Simone Pecetta / Gabriel Ozorowski / Jordan R Willis / Laura Toy / Devin Sok / Alessia Liguori / Sven Kratochvil / Jonathan L Torres ...著者: Jon M Steichen / Ying-Cing Lin / Colin Havenar-Daughton / Simone Pecetta / Gabriel Ozorowski / Jordan R Willis / Laura Toy / Devin Sok / Alessia Liguori / Sven Kratochvil / Jonathan L Torres / Oleksandr Kalyuzhniy / Eleonora Melzi / Daniel W Kulp / Sebastian Raemisch / Xiaozhen Hu / Steffen M Bernard / Erik Georgeson / Nicole Phelps / Yumiko Adachi / Michael Kubitz / Elise Landais / Jeffrey Umotoy / Amanda Robinson / Bryan Briney / Ian A Wilson / Dennis R Burton / Andrew B Ward / Shane Crotty / Facundo D Batista / William R Schief /  要旨: Vaccine induction of broadly neutralizing antibodies (bnAbs) to HIV remains a major challenge. Germline-targeting immunogens hold promise for initiating the induction of certain bnAb classes; yet for ...Vaccine induction of broadly neutralizing antibodies (bnAbs) to HIV remains a major challenge. Germline-targeting immunogens hold promise for initiating the induction of certain bnAb classes; yet for most bnAbs, a strong dependence on antibody heavy chain complementarity-determining region 3 (HCDR3) is a major barrier. Exploiting ultradeep human antibody sequencing data, we identified a diverse set of potential antibody precursors for a bnAb with dominant HCDR3 contacts. We then developed HIV envelope trimer-based immunogens that primed responses from rare bnAb-precursor B cells in a mouse model and bound a range of potential bnAb-precursor human naïve B cells in ex vivo screens. Our repertoire-guided germline-targeting approach provides a framework for priming the induction of many HIV bnAbs and could be applied to most HCDR3-dominant antibodies from other pathogens. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7884.map.gz emd_7884.map.gz | 117 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7884-v30.xml emd-7884-v30.xml emd-7884.xml emd-7884.xml | 27.1 KB 27.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7884_fsc.xml emd_7884_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7884.png emd_7884.png | 50 KB | ||
| マスクデータ |  emd_7884_msk_1.map emd_7884_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_7884_half_map_1.map.gz emd_7884_half_map_1.map.gz emd_7884_half_map_2.map.gz emd_7884_half_map_2.map.gz | 98.4 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7884 http://ftp.pdbj.org/pub/emdb/structures/EMD-7884 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7884 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7884 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7884_validation.pdf.gz emd_7884_validation.pdf.gz | 536.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7884_full_validation.pdf.gz emd_7884_full_validation.pdf.gz | 535.7 KB | 表示 | |
| XML形式データ |  emd_7884_validation.xml.gz emd_7884_validation.xml.gz | 16.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7884 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7884 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7884 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7884 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6nf5MC  7875C  7876C  7885C  6dfgC  6dfhC  6nfcC  6oc7C  7886 C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7884.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7884.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
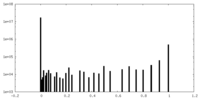
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_7884_msk_1.map emd_7884_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
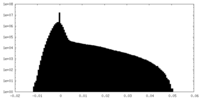
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_7884_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_7884_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BG505 MD64 N332-GT5 SOSIP trimer in complex with BG18-like precur...
| 全体 | 名称: BG505 MD64 N332-GT5 SOSIP trimer in complex with BG18-like precursor HMP1 fragment antigen binding and base-binding RM20A3 fragment antigen binding |
|---|---|
| 要素 |
|
-超分子 #1: BG505 MD64 N332-GT5 SOSIP trimer in complex with BG18-like precur...
| 超分子 | 名称: BG505 MD64 N332-GT5 SOSIP trimer in complex with BG18-like precursor HMP1 fragment antigen binding and base-binding RM20A3 fragment antigen binding タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 720 KDa |
-超分子 #2: SOSIP trimer
| 超分子 | 名称: SOSIP trimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換株: HEK293F Homo sapiens (ヒト) / 組換株: HEK293F |
-超分子 #3: HMP1 fragment antigen binding
| 超分子 | 名称: HMP1 fragment antigen binding / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換株: HEK293F Homo sapiens (ヒト) / 組換株: HEK293F |
-超分子 #4: RM20A3 fragment antigen binding
| 超分子 | 名称: RM20A3 fragment antigen binding / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #4-#5 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換株: HEK293F Homo sapiens (ヒト) / 組換株: HEK293F |
-分子 #1: BG505 MD64 N332-GT5 SOSIP
| 分子 | 名称: BG505 MD64 N332-GT5 SOSIP / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHEDIIS LWDQSLKPCV KLTPLCVTLQ CTNYAPKLRS MMRGEIKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE YRLINCNTSA ...文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHEDIIS LWDQSLKPCV KLTPLCVTLQ CTNYAPKLRS MMRGEIKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE YRLINCNTSA ITQACPKVSF EPIPIHYCAP AGFAILKCKD KKFNGTGPCP SVSTVQCTHG IKPVVSTQLL LNGSLAEEEV IIRSENITNN AKNILVQLNT PVQINCTRPS NNTVKSIRIG PGQAFYYFGD VLGHVRMAHC NISKATWNET LGKVVKQLRK HFGNNTIIRF AQSSGGDLEV TTHSFNCGGE FFYCNTSGLF NSTWISNTSV QGSNSTGSND SLILPCWIKQ IINMWQRIGQ AMYAPPIQGV IRCVSNITGL ILTRDGGSTN STTETFRPGG GDMRDNWRSE LYKYKVVKIE PLGVAPTRCK RRVVGRRRRR RAVGIGAVSL GFLGAAGSTM GAASMTLTVQ ARNLLSGIVQ QQSNLLRAPE PQQHLLKDTH WGIKQLQARV LAVEHYLRDQ QLLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNEQDL LALDGTKHHH HHH |
-分子 #2: BG18-like precursor HMP1 fragment antigen binding light chain
| 分子 | 名称: BG18-like precursor HMP1 fragment antigen binding light chain タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SYELTQPPSV SVSPGQTARI TCSGDALPKQ YAYWYQQKPG QAPVLVIYKD SERPSGIPER FSGSSSGTTV TLTISGVQAE DEADYYCQSA DSSGEVFGGG TKLTVL |
-分子 #3: BG18-like precursor HMP1 fragment antigen binding heavy chain
| 分子 | 名称: BG18-like precursor HMP1 fragment antigen binding heavy chain タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSQTLSL TCTVSGGSIS SGSYYWSWIR QPAGKGLEWI GRIYTSGSTN YNPSLKSRVT ISVDTSKNQF SLKLSSVTAA DTAVYYCARE GFTIFGVVTF SEGYYYGMDV WGKGTTVTVS S |
-分子 #4: RM20A3 fragment antigen binding light chain
| 分子 | 名称: RM20A3 fragment antigen binding light chain / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ALTQPPSVSG SPGQSVTISC TGTSSDIGSY NYVSWYQQHP GKAPKLMIYD VTQRPSGVSD RFSGSKSGNT ASLTISGLQA DDEADYYCSA YAGRQTFYIF GGGTRLTVLG QPKASPTVTL FPPSSEEL |
-分子 #5: RM20A3 fragment antigen binding heavy chain
| 分子 | 名称: RM20A3 fragment antigen binding heavy chain / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVETGGG LVQPGGSLKL SCRASGYTFS SFAMSWVRQA PGKGLEWVSL INDRGGLTFY VDSVKGRFTI SRDNSKNTLS LQMHSLRDGD TAVYYCATGG MSSALQSSKY YFDFWGQGAL VTVSS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.9 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: Detergent (DDM) added immediately prior to grid preparation | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER / 詳細: Gatan Model 950 Advanced Plasma System | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: 6 seconds blotting time. | ||||||||||||
| 詳細 | SEC purified complex |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / 撮影したグリッド数: 1 / 実像数: 1796 / 平均露光時間: 11.75 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)