+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7476 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cdc48-Npl4 complex in the presence of ATP-gamma-S | |||||||||
 マップデータ マップデータ | primary map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ / ERAD / zinc finger / ubiquitin-binding protein / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA transport / protein transport / ubiquitin-dependent protein catabolic process / nuclear membrane / cell division / perinuclear region of cytoplasm / ATP hydrolysis activity / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetomium thermophilum (菌類) / Chaetomium thermophilum (菌類) /  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) | |||||||||
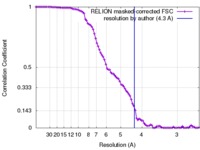
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Kim KH / Bodnar NO | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Structure of the Cdc48 ATPase with its ubiquitin-binding cofactor Ufd1-Npl4. 著者: Nicholas O Bodnar / Kelly H Kim / Zhejian Ji / Thomas E Wales / Vladimir Svetlov / Evgeny Nudler / John R Engen / Thomas Walz / Tom A Rapoport /  要旨: Many polyubiquitinated proteins are extracted from membranes or complexes by the conserved ATPase Cdc48 (in yeast; p97 or VCP in mammals) before proteasomal degradation. Each Cdc48 hexamer contains ...Many polyubiquitinated proteins are extracted from membranes or complexes by the conserved ATPase Cdc48 (in yeast; p97 or VCP in mammals) before proteasomal degradation. Each Cdc48 hexamer contains two stacked ATPase rings (D1 and D2) and six N-terminal (N) domains. Cdc48 binds various cofactors, including the Ufd1-Npl4 heterodimer. Here, we report structures of the Cdc48-Ufd1-Npl4 complex from Chaetomium thermophilum. Npl4 interacts through its UBX-like domain with a Cdc48 N domain, and it uses two Zn-finger domains to anchor the enzymatically inactive Mpr1-Pad1 N-terminal (MPN) domain, homologous to domains found in several isopeptidases, to the top of the D1 ATPase ring. The MPN domain of Npl4 is located above Cdc48's central pore, a position similar to the MPN domain from deubiquitinase Rpn11 in the proteasome. Our results indicate that Npl4 is unique among Cdc48 cofactors and suggest a mechanism for binding and translocation of polyubiquitinated substrates into the ATPase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7476.map.gz emd_7476.map.gz | 5.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7476-v30.xml emd-7476-v30.xml emd-7476.xml emd-7476.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7476_fsc.xml emd_7476_fsc.xml | 9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7476.png emd_7476.png | 119 KB | ||
| Filedesc metadata |  emd-7476.cif.gz emd-7476.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7476 http://ftp.pdbj.org/pub/emdb/structures/EMD-7476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7476 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7476_validation.pdf.gz emd_7476_validation.pdf.gz | 396.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7476_full_validation.pdf.gz emd_7476_full_validation.pdf.gz | 395.9 KB | 表示 | |
| XML形式データ |  emd_7476_validation.xml.gz emd_7476_validation.xml.gz | 10.6 KB | 表示 | |
| CIF形式データ |  emd_7476_validation.cif.gz emd_7476_validation.cif.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7476 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7476 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7476 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7476 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7476.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7476.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cdc48-Npl4/Ufd1 complex
| 全体 | 名称: Cdc48-Npl4/Ufd1 complex |
|---|---|
| 要素 |
|
-超分子 #1: Cdc48-Npl4/Ufd1 complex
| 超分子 | 名称: Cdc48-Npl4/Ufd1 complex / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
-分子 #1: Npl4
| 分子 | 名称: Npl4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 73.880391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLLRMRCPDG MFRLTVEKDD TFGELVRQLV PKLPPTVDPK SITLSNHPSG GDSKRIDEIA RFKIGQVGHG DLIFVRYQTS DTVANGRSV DISSQTAGLS SSANRLNGKP VLPTEDHPID PPPNTSAERI KNPWEVVRQS PLDDRLDKLD GKIPRKRGAM C RHGPKGMC ...文字列: MLLRMRCPDG MFRLTVEKDD TFGELVRQLV PKLPPTVDPK SITLSNHPSG GDSKRIDEIA RFKIGQVGHG DLIFVRYQTS DTVANGRSV DISSQTAGLS SSANRLNGKP VLPTEDHPID PPPNTSAERI KNPWEVVRQS PLDDRLDKLD GKIPRKRGAM C RHGPKGMC DYCTPLDPFN PQYLEEKKIK YMSVHAYMRK INSATNRPEL GSSFIPPLVE PYYRVKRDCP SGHPQWPEGI CT KCQPSAI TLQPQPFRMV DHVEFASPQI IDRFLDAWRR TGVQRLGILY GRYLEYDAVP LGIKAVVEAI YEPPQVDEID GIT LNPWEN EQEVNQVAKY CGLEQVGVIW TDLLDAGKGD GSVVCKRHAD SYFLAAQEIV FAARLQAQHP KPSKWSDTGR FGSN FVTCV VSGNEQGEIS ISAYQMSNDA VEMVRADIIE PSADPTLMLV REEEEDDGSP SKTRYIPEVF YRKINEYGAN VLENA KPAF PVEYLFVTLT HGFPDSPSPL FTDNIFPIEN REYVGEAQEV SAVAKALKVH EADAPMNVSD FHLLCFIHQM SVLSKE EEA LLCRVATLHD LAESFQLRST TGWQTLHMIL QSTGERVPKR PRFSDNVPSS SQDTQEALGG NNLDPGEPLA KRFAAVR LN ENNQSSPKPY APSRWNL UniProtKB: Nuclear protein localization protein 4 |
-分子 #2: Putative cell division control protein
| 分子 | 名称: Putative cell division control protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 90.432516 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAPESKDHKK KVNLTDPSGA EHREENDTAT AILKKKKKPN QLMVTDAVND DNSIIALSNN TMEALQLFRG DTVLVRGKKR RDTVLIVLA DDDLDDGSAR INRVVRHNLR VKHGDMITIH PCPDIKYAKR IAVLPIADTV EGLTGSLFDV FLAPYFREAY R PVRQGDLF ...文字列: MAPESKDHKK KVNLTDPSGA EHREENDTAT AILKKKKKPN QLMVTDAVND DNSIIALSNN TMEALQLFRG DTVLVRGKKR RDTVLIVLA DDDLDDGSAR INRVVRHNLR VKHGDMITIH PCPDIKYAKR IAVLPIADTV EGLTGSLFDV FLAPYFREAY R PVRQGDLF TVRGGMRQVE FKVVEVDPPE YGIVAQDTII HCEGEPIPRE EEENNLNEVG YDDIGGCRKQ LAQIREMVEL PL RHPQLFK SIGIKPPRGV LLYGPPGTGK TLMARAVANE TGAFFFLING PEIMSKMAGE SESNLRKAFE EAEKNSPAII FID EIDSIA PKREKTNGEV ERRVVSQLLT LMDGMKARSN VVVMAATNRP NSIDPALRRF GRFDREVDIG IPDPTGRLEI LQIH TKNMK LADDVDLEQI AAETHGYVGS DLAALCSEAA MQQIREKMDL IDLDEDTIDA EVLDSLGVTM DNFRYALGVS NPSAL REVA VVEVPNVRWE DIGGLEQVKQ ELKEQVQYPV DHPEKFLKFG LSPSRGVLFY GPPGTGKTML AKAVANECAA NFISVK GPE LLSMWFGESE SNIRDIFDKA RAAAPCVVFL DELDSIAKAR GGSIGDAGGA SDRVVNQLLT EMDGMTSKKN VFVIGAT NR PEQLDPALCR PGRLDQLIYV PLPDEAGRLS ILKAQLRKTP VSKDVDLAYI ASKTHGFSGA DLAFITQRAV KLAIKESI A AEIERQKARE AAGEDVNMED DEDPVPELTK RHFEEAMRDA RRSVSDVEIR RYEAFAQQMK NAGPGAFFKF PDSTTDNSA SNAAGNSFGD AGNDDDLYT UniProtKB: Putative cell division control protein |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #4: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 12 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 12 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 9299 / 平均露光時間: 15.0 sec. / 平均電子線量: 1.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6chs: |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)