+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7470 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
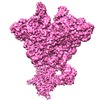
| タイトル | RAG1/2 HFC complex | |||||||||
 マップデータ マップデータ | RAG1/2 HFC complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | V(D)J recombination / RAG1/2 / RSS / Immunity / RECOMBINATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of restriction endodeoxyribonuclease activity / regulation of tolerance induction / positive regulation of mismatch repair / regulation of T cell mediated immune response to tumor cell / negative regulation of apoptotic cell clearance / negative regulation of RNA polymerase II transcription preinitiation complex assembly / DNA geometric change / myeloid dendritic cell activation / mature B cell differentiation involved in immune response / T-helper 1 cell activation ...regulation of restriction endodeoxyribonuclease activity / regulation of tolerance induction / positive regulation of mismatch repair / regulation of T cell mediated immune response to tumor cell / negative regulation of apoptotic cell clearance / negative regulation of RNA polymerase II transcription preinitiation complex assembly / DNA geometric change / myeloid dendritic cell activation / mature B cell differentiation involved in immune response / T-helper 1 cell activation / C-X-C chemokine binding / T-helper 1 cell differentiation / positive regulation of dendritic cell differentiation / DNA recombinase complex / negative regulation of CD4-positive, alpha-beta T cell differentiation / B cell homeostatic proliferation / DN2 thymocyte differentiation / negative regulation of T cell differentiation in thymus / endodeoxyribonuclease complex / positive regulation of toll-like receptor 9 signaling pathway / neutrophil clearance / positive regulation of DNA ligation / double-stranded DNA endonuclease activity / pre-B cell allelic exclusion / positive regulation of interleukin-1 production / positive regulation of organ growth / RAGE receptor binding / Regulation of TLR by endogenous ligand / regulation of behavioral fear response / alphav-beta3 integrin-HMGB1 complex / bubble DNA binding / V(D)J recombination / negative regulation of T cell apoptotic process / Apoptosis induced DNA fragmentation / phosphatidylinositol-3,4-bisphosphate binding / inflammatory response to antigenic stimulus / negative regulation of thymocyte apoptotic process / positive regulation of chemokine (C-X-C motif) ligand 2 production / positive regulation of monocyte chemotaxis / MyD88 deficiency (TLR2/4) / supercoiled DNA binding / phosphatidylinositol-3,5-bisphosphate binding / apoptotic cell clearance / dendritic cell chemotaxis / positive regulation of T cell differentiation / DNA binding, bending / positive regulation of vascular endothelial cell proliferation / IRAK4 deficiency (TLR2/4) / regulation of T cell differentiation / MyD88:MAL(TIRAP) cascade initiated on plasma membrane / organ growth / T cell lineage commitment / B cell lineage commitment / positive regulation of activated T cell proliferation / phosphatidylserine binding / chemoattractant activity / T cell homeostasis / phosphatidylinositol-3,4,5-trisphosphate binding / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process / DNA topological change / TRAF6 mediated NF-kB activation / positive regulation of interleukin-10 production / Advanced glycosylation endproduct receptor signaling / negative regulation of blood vessel endothelial cell migration / negative regulation of type II interferon production / endoplasmic reticulum-Golgi intermediate compartment / T cell differentiation / positive regulation of blood vessel endothelial cell migration / positive regulation of DNA binding / protein autoubiquitination / Pyroptosis / positive regulation of autophagy / heterochromatin formation / DNA polymerase binding / four-way junction DNA binding / condensed chromosome / methylated histone binding / phosphatidylinositol-4,5-bisphosphate binding / positive regulation of interleukin-12 production / activation of innate immune response / transcription repressor complex / phosphatidylinositol binding / B cell differentiation / thymus development / cytokine activity / positive regulation of interleukin-8 production / lipopolysaccharide binding / positive regulation of JNK cascade / TAK1-dependent IKK and NF-kappa-B activation / RING-type E3 ubiquitin transferase / visual learning / autophagy / double-strand break repair via nonhomologous end joining / ubiquitin-protein transferase activity / transcription corepressor activity / positive regulation of interleukin-6 production / positive regulation of tumor necrosis factor production / neuron projection development / ubiquitin protein ligase activity / integrin binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.17 Å | |||||||||
 データ登録者 データ登録者 | Chen X / Kim M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2018 ジャーナル: Mol Cell / 年: 2018タイトル: Cracking the DNA Code for V(D)J Recombination. 著者: Min-Sung Kim / Watchalee Chuenchor / Xuemin Chen / Yanxiang Cui / Xing Zhang / Z Hong Zhou / Martin Gellert / Wei Yang /    要旨: To initiate V(D)J recombination for generating the adaptive immune response of vertebrates, RAG1/2 recombinase cleaves DNA at a pair of recombination signal sequences, the 12- and 23-RSS. We have ...To initiate V(D)J recombination for generating the adaptive immune response of vertebrates, RAG1/2 recombinase cleaves DNA at a pair of recombination signal sequences, the 12- and 23-RSS. We have determined crystal and cryo-EM structures of RAG1/2 with DNA in the pre-reaction and hairpin-forming complexes up to 2.75 Å resolution. Both protein and DNA exhibit structural plasticity and undergo dramatic conformational changes. Coding-flank DNAs extensively rotate, shift, and deform for nicking and hairpin formation. Two intertwined RAG1 subunits crisscross four times between the asymmetric pair of severely bent 12/23-RSS DNAs. Location-sensitive bending of 60° and 150° in 12- and 23-RSS spacers, respectively, must occur for RAG1/2 to capture the nonamers and pair the heptamers for symmetric double-strand breakage. DNA pairing is thus sequence-context dependent and structure specific, which partly explains the "beyond 12/23" restriction. Finally, catalysis in crystallo reveals the process of DNA hairpin formation and its stabilization by interleaved base stacking. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7470.map.gz emd_7470.map.gz | 5.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7470-v30.xml emd-7470-v30.xml emd-7470.xml emd-7470.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7470.png emd_7470.png | 182.5 KB | ||
| Filedesc metadata |  emd-7470.cif.gz emd-7470.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7470 http://ftp.pdbj.org/pub/emdb/structures/EMD-7470 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7470 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7470 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7470_validation.pdf.gz emd_7470_validation.pdf.gz | 362.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7470_full_validation.pdf.gz emd_7470_full_validation.pdf.gz | 362.3 KB | 表示 | |
| XML形式データ |  emd_7470_validation.xml.gz emd_7470_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_7470_validation.cif.gz emd_7470_validation.cif.gz | 7.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7470 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7470 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7470 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7470 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6cg0MC  7480C  5zdzC  5ze0C  5ze1C  5ze2C  6cijC  6cikC  6cilC  6cimC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7470.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7470.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RAG1/2 HFC complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
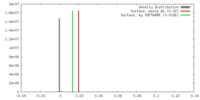
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : RAG1/2 in complex with nicked DNAs
+超分子 #1: RAG1/2 in complex with nicked DNAs
+分子 #1: V(D)J recombination-activating protein 1
+分子 #2: V(D)J recombination-activating protein 2
+分子 #9: High mobility group protein B1
+分子 #3: DNA (46-MER)
+分子 #4: DNA (5'-D(*GP*AP*TP*CP*TP*GP*GP*CP*CP*TP*GP*TP*CP*TP*TP*A)-3')
+分子 #5: DNA (5'-D(P*CP*TP*GP*GP*AP*TP*CP*TP*GP*GP*CP*CP*TP*GP*TP*CP*TP*TP...
+分子 #6: DNA (60-MER)
+分子 #7: DNA (30-MER)
+分子 #8: DNA (41-MER)
+分子 #10: ZINC ION
+分子 #11: CALCIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 57.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.17 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 2.1) / 使用した粒子像数: 139781 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: COMMON LINE |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation Coefficient |
|---|---|
| 得られたモデル |  PDB-6cg0: |
 ムービー
ムービー コントローラー
コントローラー


































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)