+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7018 | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human apo-TRPML3 channel at pH 7.4 | ||||||||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||
 キーワード キーワード | ion channel / TRP channel / lysosomal / TRANSPORT PROTEIN | ||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NAADP-sensitive calcium-release channel activity / inner ear auditory receptor cell differentiation / monoatomic anion channel activity / TRP channels / sodium channel activity / autophagosome membrane / potassium channel activity / locomotory behavior / calcium ion transmembrane transport / calcium channel activity ...NAADP-sensitive calcium-release channel activity / inner ear auditory receptor cell differentiation / monoatomic anion channel activity / TRP channels / sodium channel activity / autophagosome membrane / potassium channel activity / locomotory behavior / calcium ion transmembrane transport / calcium channel activity / late endosome membrane / early endosome membrane / lysosomal membrane / lipid binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.06 Å | ||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Zhou X / Li M | ||||||||||||||||||||||||||||||
| 資金援助 |  中国, 中国,  米国, 9件 米国, 9件
| ||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2017 ジャーナル: Nat Struct Mol Biol / 年: 2017タイトル: Cryo-EM structures of the human endolysosomal TRPML3 channel in three distinct states. 著者: Xiaoyuan Zhou / Minghui Li / Deyuan Su / Qi Jia / Huan Li / Xueming Li / Jian Yang /   要旨: TRPML3 channels are mainly localized to endolysosomes and play a critical role in the endocytic pathway. Their dysfunction causes deafness and pigmentation defects in mice. TRPML3 activity is ...TRPML3 channels are mainly localized to endolysosomes and play a critical role in the endocytic pathway. Their dysfunction causes deafness and pigmentation defects in mice. TRPML3 activity is inhibited by low endolysosomal pH. Here we present cryo-electron microscopy (cryo-EM) structures of human TRPML3 in the closed, agonist-activated, and low-pH-inhibited states, with resolutions of 4.06, 3.62, and 4.65 Å, respectively. The agonist ML-SA1 lodges between S5 and S6 and opens an S6 gate. A polycystin-mucolipin domain (PMD) forms a luminal cap. S1 extends into this cap, forming a 'gating rod' that connects directly to a luminal pore loop, which undergoes dramatic conformational changes in response to low pH. S2 extends intracellularly and interacts with several intracellular regions to form a 'gating knob'. These unique structural features, combined with the results of electrophysiological studies, indicate a new mechanism by which luminal pH and other physiological modulators such as PIP regulate TRPML3 by changing S1 and S2 conformations. | ||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7018.map.gz emd_7018.map.gz | 2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7018-v30.xml emd-7018-v30.xml emd-7018.xml emd-7018.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7018.png emd_7018.png | 89.3 KB | ||
| Filedesc metadata |  emd-7018.cif.gz emd-7018.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7018 http://ftp.pdbj.org/pub/emdb/structures/EMD-7018 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7018 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7018 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7018_validation.pdf.gz emd_7018_validation.pdf.gz | 398 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7018_full_validation.pdf.gz emd_7018_full_validation.pdf.gz | 397.6 KB | 表示 | |
| XML形式データ |  emd_7018_validation.xml.gz emd_7018_validation.xml.gz | 5.5 KB | 表示 | |
| CIF形式データ |  emd_7018_validation.cif.gz emd_7018_validation.cif.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7018 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7018 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7018 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7018 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7018.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7018.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
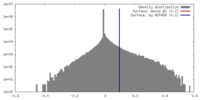
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : human TRPML3 apo channel at pH 7.4
| 全体 | 名称: human TRPML3 apo channel at pH 7.4 |
|---|---|
| 要素 |
|
-超分子 #1: human TRPML3 apo channel at pH 7.4
| 超分子 | 名称: human TRPML3 apo channel at pH 7.4 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Mucolipin-3
| 分子 | 名称: Mucolipin-3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 64.625785 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GGGGSMADPE VVVSSCSSHE EENRCNFNQQ TSPSEELLLE DQMRRKLKFF FMNPCEKFWA RGRKPWKLAI QILKIAMVTI QLVLFGLSN QMVVAFKEEN TIAFKHLFLK GYMDRMDDTY AVYTQSDVYD QLIFAVNQYL QLYNVSVGNH AYENKGTKQS A MAICQHFY ...文字列: GGGGSMADPE VVVSSCSSHE EENRCNFNQQ TSPSEELLLE DQMRRKLKFF FMNPCEKFWA RGRKPWKLAI QILKIAMVTI QLVLFGLSN QMVVAFKEEN TIAFKHLFLK GYMDRMDDTY AVYTQSDVYD QLIFAVNQYL QLYNVSVGNH AYENKGTKQS A MAICQHFY KRGNIYPGND TFDIDPEIET ECFFVEPDEP FHIGTPAENK LNLTLDFHRL LTVELQFKLK AINLQTVRHQ EL PDCYDFT LTITFDNKAH SGRIKISLDN DISIRECKDW HVSGSIQKNT HYMMIFDAFV ILTCLVSLIL CIRSVIRGLQ LQQ EFVNFF LLHYKKEVSV SDQMEFVNGW YIMIIISDIL TIIGSILKME IQAKSLTSYD VCSILLGTST MLVWLGVIRY LGFF AKYNL LILTLQAALP NVIRFCCCAA MIYLGYCFCG WIVLGPYHDK FRSLNMVSEC LFSLINGDDM FATFAKMQQK SYLVW LFSR IYLYSFISLF IYMILSLFIA LITDTYETIK QYQQDGFPET ELRTFISECK DLPNSGKYRL EDDPPVSLFC CCKK UniProtKB: Mucolipin-3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.9 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: The pH of the buffer was adjusted to 7.4 by NaOH. | |||||||||
| グリッド | モデル: Quantifoil holey carbon grid R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV 詳細: waiting for 3 seconds before blotting for 4 seconds(double-sided, blot force 1),then the grid was immediately plunged into liquid ethane cooled by liquid-nitrogen. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - 画像ごとのフレーム数: 1-32 / 平均露光時間: 8.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)