+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6b8h | ||||||
|---|---|---|---|---|---|---|---|
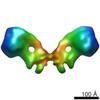
| タイトル | Mosaic model of yeast mitochondrial ATP synthase monomer | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | MEMBRANE PROTEIN / Complex / Proton transporter | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Mitochondrial protein degradation / mitochondrial proton-transporting ATP synthase complex assembly / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / proton-transporting two-sector ATPase complex, proton-transporting domain / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATPase activity, rotational mechanism / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex ...Mitochondrial protein degradation / mitochondrial proton-transporting ATP synthase complex assembly / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / proton-transporting two-sector ATPase complex, proton-transporting domain / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATPase activity, rotational mechanism / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / proton transmembrane transport / ADP binding / mitochondrial intermembrane space / protein-containing complex assembly / mitochondrial inner membrane / lipid binding / ATP hydrolysis activity / mitochondrion / ATP binding / identical protein binding / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||
 データ登録者 データ登録者 | Guo, H. / Bueler, S.A. / Rubinstein, J.L. | ||||||
 引用 引用 |  ジャーナル: Science / 年: 2017 ジャーナル: Science / 年: 2017タイトル: Atomic model for the dimeric F region of mitochondrial ATP synthase. 著者: Hui Guo / Stephanie A Bueler / John L Rubinstein /  要旨: Mitochondrial adenosine triphosphate (ATP) synthase produces the majority of ATP in eukaryotic cells, and its dimerization is necessary to create the inner membrane folds, or cristae, characteristic ...Mitochondrial adenosine triphosphate (ATP) synthase produces the majority of ATP in eukaryotic cells, and its dimerization is necessary to create the inner membrane folds, or cristae, characteristic of mitochondria. Proton translocation through the membrane-embedded F region turns the rotor that drives ATP synthesis in the soluble F region. Although crystal structures of the F region have illustrated how this rotation leads to ATP synthesis, understanding how proton translocation produces the rotation has been impeded by the lack of an experimental atomic model for the F region. Using cryo-electron microscopy, we determined the structure of the dimeric F complex from at a resolution of 3.6 angstroms. The structure clarifies how the protons travel through the complex, how the complex dimerizes, and how the dimers bend the membrane to produce cristae. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6b8h.cif.gz 6b8h.cif.gz | 3.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6b8h.ent.gz pdb6b8h.ent.gz | 2.6 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6b8h.json.gz 6b8h.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6b8h_validation.pdf.gz 6b8h_validation.pdf.gz | 2.5 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6b8h_full_validation.pdf.gz 6b8h_full_validation.pdf.gz | 2.6 MB | 表示 | |
| XML形式データ |  6b8h_validation.xml.gz 6b8h_validation.xml.gz | 220.5 KB | 表示 | |
| CIF形式データ |  6b8h_validation.cif.gz 6b8h_validation.cif.gz | 369.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b8/6b8h https://data.pdbj.org/pub/pdb/validation_reports/b8/6b8h ftp://data.pdbj.org/pub/pdb/validation_reports/b8/6b8h ftp://data.pdbj.org/pub/pdb/validation_reports/b8/6b8h | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP synthase ... , 16種, 58分子 1234567890LMNPQRSTUJAVapbqdres...
| #1: タンパク質 | 分子量: 7762.375 Da / 分子数: 20 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P61829 #2: タンパク質・ペプチド | 分子量: 5825.215 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P00856 #3: タンパク質 | 分子量: 27900.430 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P00854 #4: タンパク質 | 分子量: 23194.498 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P05626 #5: タンパク質 | 分子量: 19709.424 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P30902 #6: タンパク質・ペプチド | 分子量: 4188.154 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c #7: タンパク質 | 分子量: 10584.166 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: Q06405 #9: タンパク質 | 分子量: 6696.771 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P81450 #10: タンパク質 | 分子量: 7546.778 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P81451 #11: タンパク質 | 分子量: 55007.402 Da / 分子数: 6 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P07251 #12: タンパク質 | 分子量: 51181.082 Da / 分子数: 6 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P00830, H+-transporting two-sector ATPase #13: タンパク質 | 分子量: 30657.160 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P38077 #14: タンパク質 | 分子量: 14565.385 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: Q12165 #15: タンパク質 | 分子量: 6642.381 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  #16: タンパク質 | 分子量: 20901.139 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P09457 #17: タンパク質・ペプチド | 分子量: 1805.216 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c |
|---|
-タンパク質 , 1種, 2分子 gu
| #8: タンパク質 | 分子量: 9039.134 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c |
|---|
-非ポリマー , 2種, 20分子 


| #18: 化合物 | ChemComp-ANP / #19: 化合物 | ChemComp-MG / |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Mosaic model of the dimeric F1FO yeast mitochondrial ATP synthase タイプ: COMPLEX / Entity ID: #1-#17 / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.6 MDa / 実験値: YES |
| 由来(天然) | 生物種:  |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 71 e/Å2 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION |
|---|---|
| 3次元再構成 | 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 238848 / クラス平均像の数: 1 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj















