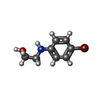
Entry Database : PDB / ID : 6xdsTitle Crystal structure of MBP-TREM2 Ig domain fusion with fragment, 2-((4-bromophenyl)amino)ethan-1-ol Sugar ABC transporter substrate-binding protein,Triggering receptor expressed on myeloid cells 2 Keywords / / / / Function / homology Function Domain/homology Component
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / Biological species Methanosarcina mazei (archaea)Homo sapiens (human)Method / / / Resolution : 1.466 Å Authors Su, H.P. Journal : Protein Expr.Purif. / Year : 2021Title : Development of a robust crystallization platform for immune receptor TREM2 using a crystallization chaperone strategy.Authors : Byrne, N.J. / Lee, A.C. / Kostas, J. / Reid, J.C. / Partridge, A.T. / So, S.S. / Cowan, J.E. / Abeywickrema, P. / Huang, H. / Zebisch, M. / Barker, J.J. / Soisson, S.M. / Brooun, A. / Su, H.P. History Deposition Jun 11, 2020 Deposition site / Processing site Revision 1.0 Feb 17, 2021 Provider / Type Revision 1.1 Nov 20, 2024 Group / Database references / Structure summaryCategory chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature Item / _database_2.pdbx_database_accession / _pdbx_entry_details.has_protein_modification
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information Methanosarcina mazei (archaea)
Methanosarcina mazei (archaea) Homo sapiens (human)
Homo sapiens (human) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  FOURIER SYNTHESIS / Resolution: 1.466 Å
FOURIER SYNTHESIS / Resolution: 1.466 Å  Authors
Authors Citation
Citation Journal: Protein Expr.Purif. / Year: 2021
Journal: Protein Expr.Purif. / Year: 2021 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 6xds.cif.gz
6xds.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb6xds.ent.gz
pdb6xds.ent.gz PDB format
PDB format 6xds.json.gz
6xds.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads 6xds_validation.pdf.gz
6xds_validation.pdf.gz wwPDB validaton report
wwPDB validaton report 6xds_full_validation.pdf.gz
6xds_full_validation.pdf.gz 6xds_validation.xml.gz
6xds_validation.xml.gz 6xds_validation.cif.gz
6xds_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xd/6xds
https://data.pdbj.org/pub/pdb/validation_reports/xd/6xds ftp://data.pdbj.org/pub/pdb/validation_reports/xd/6xds
ftp://data.pdbj.org/pub/pdb/validation_reports/xd/6xds Links
Links Assembly
Assembly
 Components
Components Methanosarcina mazei (archaea), (gene. exp.)
Methanosarcina mazei (archaea), (gene. exp.)  Homo sapiens (human)
Homo sapiens (human) Homo sapiens (human)
Homo sapiens (human) X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  APS
APS  / Beamline: 19-ID / Wavelength: 0.91636 Å
/ Beamline: 19-ID / Wavelength: 0.91636 Å Processing
Processing FOURIER SYNTHESIS / Resolution: 1.466→21.44 Å / Cor.coef. Fo:Fc: 0.957 / Cor.coef. Fo:Fc free: 0.948 / SU R Cruickshank DPI: 0.085 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.09 / SU Rfree Blow DPI: 0.086 / SU Rfree Cruickshank DPI: 0.082
FOURIER SYNTHESIS / Resolution: 1.466→21.44 Å / Cor.coef. Fo:Fc: 0.957 / Cor.coef. Fo:Fc free: 0.948 / SU R Cruickshank DPI: 0.085 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.09 / SU Rfree Blow DPI: 0.086 / SU Rfree Cruickshank DPI: 0.082  Movie
Movie Controller
Controller










 PDBj
PDBj