+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6783 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GMPCPP-microtubule complexed with nucleotide-free KIF5C | |||||||||||||||
 マップデータ マップデータ | KIF5C(nucleotide-free) and Microtubule(GMPCPP) | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Microtubule / KIF5C / Kinesin / STRUCTURAL PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報motile cilium / structural constituent of cytoskeleton / microtubule cytoskeleton organization / neuron migration / mitotic cell cycle / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / microtubule / hydrolase activity / GTPase activity / GTP binding ...motile cilium / structural constituent of cytoskeleton / microtubule cytoskeleton organization / neuron migration / mitotic cell cycle / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / microtubule / hydrolase activity / GTPase activity / GTP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   | |||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 6.43 Å | |||||||||||||||
 データ登録者 データ登録者 | Morikawa M / Shigematsu H / Nitta R | |||||||||||||||
| 資金援助 |  日本, 4件 日本, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2018 ジャーナル: J Cell Biol / 年: 2018タイトル: Kinesin-binding-triggered conformation switching of microtubules contributes to polarized transport. 著者: Tomohiro Shima / Manatsu Morikawa / Junichi Kaneshiro / Taketoshi Kambara / Shinji Kamimura / Toshiki Yagi / Hiroyuki Iwamoto / Sotaro Uemura / Hideki Shigematsu / Mikako Shirouzu / Taro ...著者: Tomohiro Shima / Manatsu Morikawa / Junichi Kaneshiro / Taketoshi Kambara / Shinji Kamimura / Toshiki Yagi / Hiroyuki Iwamoto / Sotaro Uemura / Hideki Shigematsu / Mikako Shirouzu / Taro Ichimura / Tomonobu M Watanabe / Ryo Nitta / Yasushi Okada / Nobutaka Hirokawa /   要旨: Kinesin-1, the founding member of the kinesin superfamily of proteins, is known to use only a subset of microtubules for transport in living cells. This biased use of microtubules is proposed as the ...Kinesin-1, the founding member of the kinesin superfamily of proteins, is known to use only a subset of microtubules for transport in living cells. This biased use of microtubules is proposed as the guidance cue for polarized transport in neurons, but the underlying mechanisms are still poorly understood. Here, we report that kinesin-1 binding changes the microtubule lattice and promotes further kinesin-1 binding. This high-affinity state requires the binding of kinesin-1 in the nucleotide-free state. Microtubules return to the initial low-affinity state by washing out the binding kinesin-1 or by the binding of non-hydrolyzable ATP analogue AMPPNP to kinesin-1. X-ray fiber diffraction, fluorescence speckle microscopy, and second-harmonic generation microscopy, as well as cryo-EM, collectively demonstrated that the binding of nucleotide-free kinesin-1 to GDP microtubules changes the conformation of the GDP microtubule to a conformation resembling the GTP microtubule. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6783.map.gz emd_6783.map.gz | 28.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6783-v30.xml emd-6783-v30.xml emd-6783.xml emd-6783.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6783.png emd_6783.png | 312.3 KB | ||
| Filedesc metadata |  emd-6783.cif.gz emd-6783.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6783 http://ftp.pdbj.org/pub/emdb/structures/EMD-6783 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6783 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6783 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6783.map.gz / 形式: CCP4 / 大きさ: 30.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6783.map.gz / 形式: CCP4 / 大きさ: 30.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
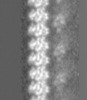
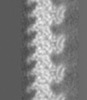
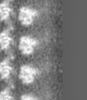
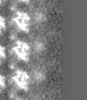
| 注釈 | KIF5C(nucleotide-free) and Microtubule(GMPCPP) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.284 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
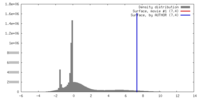
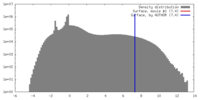
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : GMPCPP-microtubule complexed with nucleotide-free KIF5C
| 全体 | 名称: GMPCPP-microtubule complexed with nucleotide-free KIF5C |
|---|---|
| 要素 |
|
-超分子 #1: GMPCPP-microtubule complexed with nucleotide-free KIF5C
| 超分子 | 名称: GMPCPP-microtubule complexed with nucleotide-free KIF5C タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Tubulin alpha-1A chain
| 分子 | 名称: Tubulin alpha-1A chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.638793 KDa |
| 配列 | 文字列: RECISIHVGQ AGVQIGNACW ELYCLEHGIQ PDGQMPSDKT IGGGDDSFNT FFSETGAGKH VPRAVFVDLE PTVIDEVRTG TYRQLFHPE QLITGKEDAA NNYARGHYTI GKEIIDLVLD RIRKLADQCT GLQGFSVFHS FGGGTGSGFT SLLMERLSVD Y GKKSKLEF ...文字列: RECISIHVGQ AGVQIGNACW ELYCLEHGIQ PDGQMPSDKT IGGGDDSFNT FFSETGAGKH VPRAVFVDLE PTVIDEVRTG TYRQLFHPE QLITGKEDAA NNYARGHYTI GKEIIDLVLD RIRKLADQCT GLQGFSVFHS FGGGTGSGFT SLLMERLSVD Y GKKSKLEF SIYPAPQVST AVVEPYNSIL TTHTTLEHSD CAFMVDNEAI YDICRRNLDI ERPTYTNLNR LIGQIVSSIT AS LRFDGAL NVDLTEFQTN LVPYPRGHFP LATYAPVISA EKAYHEQLSV AEITNACFEP ANQMVKCDPR HGKYMACCLL YRG DVVPKD VNAAIATIKT KRTIQFVDWC PTGFKVGINY EPPTVVPGGD LAKVQRAVCM LSNTTAIAEA WARLDHKFDL MYAK RAFVH WYVGEGMEEG EFSEAREDMA ALEKDYEEVG VDS UniProtKB: Tubulin alpha-1A chain |
-分子 #2: Tubulin beta chain
| 分子 | 名称: Tubulin beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 47.809746 KDa |
| 配列 | 文字列: REIVHIQAGQ CGNQIGAKFW EVISDEHGID PTGSYHGDSD LQLERINVYY NEAAGNKYVP RAILVDLEPG TMDSVRSGPF GQIFRPDNF VFGQSGAGNN WAKGHYTEGA ELVDSVLDVV RKESESCDCL QGFQLTHSLG GGTGSGMGTL LISKIREEYP D RIMNTFSV ...文字列: REIVHIQAGQ CGNQIGAKFW EVISDEHGID PTGSYHGDSD LQLERINVYY NEAAGNKYVP RAILVDLEPG TMDSVRSGPF GQIFRPDNF VFGQSGAGNN WAKGHYTEGA ELVDSVLDVV RKESESCDCL QGFQLTHSLG GGTGSGMGTL LISKIREEYP D RIMNTFSV VPSPKVSDTV VEPYNATLSV HQLVENTDET YCIDNEALYD ICFRTLKLTT PTYGDLNHLV SATMSGVTTC LR FPGQLNA DLRKLAVNMV PFPRLHFFMP GFAPLTSRGS QQYRALTVPE LTQQMFDAKN MMAACDPRHG RYLTVAAVFR GRM SMKEVD EQMLNVQNKN SSYFVEWIPN NVKTAVCDIP PRGLKMSATF IGNSTAIQEL FKRISEQFTA MFRRKAFLHW YTGE GMDEM EFTEAESNMN DLVSEYQQYQ D UniProtKB: Tubulin beta chain |
-分子 #3: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 9 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 18 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER
| 分子 | 名称: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 9 / 式: G2P |
|---|---|
| 分子量 | 理論値: 521.208 Da |
| Chemical component information | 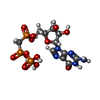 ChemComp-G2P: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 36 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 300 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 8.8632 Å 想定した対称性 - らせんパラメータ - ΔΦ: -25.7541 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 6.43 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIGN (ver. 9) / 使用した粒子像数: 5822 |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-5xxx: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)