+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6470 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle reconstruction of AAV-DJ in complex with ARIXTRA | |||||||||
 マップデータ マップデータ | Reconstruction of adeno-associated virus variant DJ in complex with Arixtra | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | gene therapy | |||||||||
| 生物種 | unidentified (未定義) /   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) | |||||||||
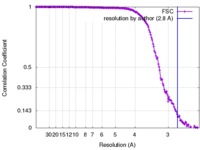
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Spear JM / Noble AJ / Xie Q / Sousa DR / Chapman MS / Stagg SM | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2015 ジャーナル: J Struct Biol / 年: 2015タイトル: The influence of frame alignment with dose compensation on the quality of single particle reconstructions. 著者: John M Spear / Alex J Noble / Qing Xie / Duncan R Sousa / Michael S Chapman / Scott M Stagg /  要旨: As direct electron detection devices in cryo-electron microscopy become ubiquitous, the field is now ripe for new developments in image analysis techniques that take advantage of their increased SNR ...As direct electron detection devices in cryo-electron microscopy become ubiquitous, the field is now ripe for new developments in image analysis techniques that take advantage of their increased SNR coupled with their high-throughput frame collection abilities. In approaching atomic resolution of native-like biomolecules, the accurate extraction of structural locations and orientations of side-chains from frames depends not only on the electron dose that a sample receives but also on the ability to accurately estimate the CTF. Here we use a new 2.8Å resolution structure of a recombinant gene therapy virus, AAV-DJ with Arixtra, imaged on an FEI Titan Krios with a DE-20 direct electron detector to probe new metrics including relative side-chain density and ResLog analysis for optimizing the compensation of electron beam damage and to characterize the factors that are limiting the resolution of the reconstruction. The influence of dose compensation on the accuracy of CTF estimation and particle classifiability are also presented. We show that rigorous dose compensation allows for better particle classifiability and greater recovery of structural information from negatively charged, electron-sensitive side-chains, resulting in a more accurate macromolecular model. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6470.map.gz emd_6470.map.gz | 258.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6470-v30.xml emd-6470-v30.xml emd-6470.xml emd-6470.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6470_fsc.xml emd_6470_fsc.xml | 17.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  400_6470.gif 400_6470.gif 80_6470.gif 80_6470.gif | 90.7 KB 4.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6470 http://ftp.pdbj.org/pub/emdb/structures/EMD-6470 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6470 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6470 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6470_validation.pdf.gz emd_6470_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6470_full_validation.pdf.gz emd_6470_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_6470_validation.xml.gz emd_6470_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6470 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6470 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6470 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6470 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6470.map.gz / 形式: CCP4 / 大きさ: 300.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6470.map.gz / 形式: CCP4 / 大きさ: 300.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of adeno-associated virus variant DJ in complex with Arixtra | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.215 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus
| 全体 | 名称:   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: Adeno-associated virus
| 超分子 | 名称: Adeno-associated virus / タイプ: sample / ID: 1000 / 集合状態: 60 viral subunits form the icosahedral capsid / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 3.75 MDa |
-超分子 #1: Adeno-associated virus
| 超分子 | 名称: Adeno-associated virus / タイプ: virus / ID: 1 / NCBI-ID: 272636 / 生物種: Adeno-associated virus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Sci species serotype: DJ |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| Host system | 生物種:  組換細胞: SF9 / 組換プラスミド: bacmid |
| 分子量 | 実験値: 3.75 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Shell 1 / 直径: 250 Å / T番号(三角分割数): 1 |
-分子 #1: Fondaparinux
| 分子 | 名称: Fondaparinux / タイプ: ligand / ID: 1 / Name.synonym: Arixtra 詳細: Arixtra was added in a 10X molar excess to AAV-DJ monomers 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 実験値: 1.727 KDa |
| Chemical component information | 
ChemComp-PRD_900028: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50mM HEPES, 25mM MgCl2, 25mM NaCl |
| グリッド | 詳細: R2/2 200 mesh copper grids that were rendered hydrophilic in 75/25 % Ar/O. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK IV 詳細: 3ul of 0.6 mg/ml AAV-DJ was applied to grid and then hand blotted. After initial hand blotting, 3ul of 5.7mM Arixtra was added, the grid was mechanically blotted, and then vitrified. 手法: blot force = 1, blot time = 3 seconds, total blots = 1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 平均: 93 K |
| アライメント法 | Legacy - 非点収差: Astigmatism was corrected at 155000 times magnification |
| 日付 | 2015年1月6日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 実像数: 1051 / 平均電子線量: 66 e/Å2 / 詳細: every image is the sum of 45 frames / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 49383 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダー: liquid nitrogen cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー