+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6239 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Atomic structure of a non-enveloped virus reveals pH sensors for a coordinated process of cell entry | |||||||||
 マップデータ マップデータ | Reconstruction of BTV virion | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Non-enveloped virus / cell entry / cryo-EM / pH sensor | |||||||||
| 機能・相同性 | Outer capsid protein VP2, Orbivirus / Orbivirus outer capsid protein VP2 / clathrin-dependent endocytosis of virus by host cell / virion attachment to host cell / structural molecule activity / Outer capsid protein VP2 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Bluetongue virus 1 (ウイルス) Bluetongue virus 1 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Zhang X / Patel A / Celma C / Roy P / Zhou ZH | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2016 ジャーナル: Nat Struct Mol Biol / 年: 2016タイトル: Atomic model of a nonenveloped virus reveals pH sensors for a coordinated process of cell entry. 著者: Xing Zhang / Avnish Patel / Cristina C Celma / Xuekui Yu / Polly Roy / Z Hong Zhou /   要旨: Viruses sense environmental cues such as pH to engage in membrane interactions for cell entry during infection, but how nonenveloped viruses sense pH is largely undefined. Here, we report both high- ...Viruses sense environmental cues such as pH to engage in membrane interactions for cell entry during infection, but how nonenveloped viruses sense pH is largely undefined. Here, we report both high- and low-pH structures of bluetongue virus (BTV), which enters cells via a two-stage endosomal process. The receptor-binding protein VP2 possesses a zinc finger that may function to maintain VP2 in a metastable state and a conserved His866, which senses early-endosomal pH. The membrane-penetration protein VP5 has three domains: dagger, unfurling and anchoring. Notably, the β-meander motif of the anchoring domain contains a histidine cluster that can sense late-endosomal pH and also possesses four putative membrane-interaction elements. Exposing BTV to low pH detaches VP2 and dramatically refolds the dagger and unfurling domains of VP5. Our biochemical and structure-guided-mutagenesis studies support these coordinated pH-sensing mechanisms. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6239.map.gz emd_6239.map.gz | 477.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6239-v30.xml emd-6239-v30.xml emd-6239.xml emd-6239.xml | 9.2 KB 9.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6239.gif 400_6239.gif 80_6239.gif 80_6239.gif | 35.7 KB 3.3 KB | ||
| その他 |  emd_6239_additional_1.map.gz emd_6239_additional_1.map.gz | 55 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6239 http://ftp.pdbj.org/pub/emdb/structures/EMD-6239 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6239 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6239 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6239_validation.pdf.gz emd_6239_validation.pdf.gz | 287.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6239_full_validation.pdf.gz emd_6239_full_validation.pdf.gz | 286.6 KB | 表示 | |
| XML形式データ |  emd_6239_validation.xml.gz emd_6239_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6239 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6239 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6239 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6239 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6239.map.gz / 形式: CCP4 / 大きさ: 11.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6239.map.gz / 形式: CCP4 / 大きさ: 11.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of BTV virion | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.01 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 6239 additional 1.map
| ファイル | emd_6239_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
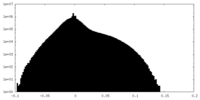
| 投影像・断面図 |
| ||||||||||||
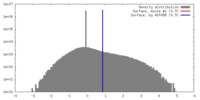
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VP2 of Bluetongue Virus
| 全体 | 名称: VP2 of Bluetongue Virus |
|---|---|
| 要素 |
|
-超分子 #1000: VP2 of Bluetongue Virus
| 超分子 | 名称: VP2 of Bluetongue Virus / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Bluetongue virus 1
| 超分子 | 名称: Bluetongue virus 1 / タイプ: virus / ID: 1 / NCBI-ID: 35327 / 生物種: Bluetongue virus 1 / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 800 Å / T番号(三角分割数): 13 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.8 |
|---|---|
| グリッド | 詳細: 400 mesh grid with thin carbon support |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 90 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 平均: 82 K |
| 日付 | 2013年11月29日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / デジタル化 - サンプリング間隔: 5 µm / 実像数: 1630 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 32 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 24140 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.3 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 14000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: each particle |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Frealign / 使用した粒子像数: 5008 |
| 最終 2次元分類 | クラス数: 1 |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)