+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5odq | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Heterodisulfide reductase / [NiFe]-hydrogenase complex from Methanothermococcus thermolithotrophicus soaked with bromoethanesulfonate. | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | OXIDOREDUCTASE / Heterodisulfide reductase / [NiFe]-hydrogenase / FeS cluster / ferredoxin / CCG motif / methanogenesis / flavoprotein / flavin-based electron bifurcation / [2Fe-2S] cluster / macromolecular complex / anaerobic / thioredoxin / bromoethanesulfonate / metabolism | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dihydromethanophenazine:CoB-CoM heterodisulfide reductase / hydrogen dehydrogenase / hydrogen dehydrogenase activity / CoB--CoM heterodisulfide reductase activity / 酸化還元酵素; 含硫化合物に対し酸化酵素として働く / hydrogenase (acceptor) / methanogenesis / hydrogenase (acceptor) activity / ferredoxin hydrogenase activity / nickel cation binding ...dihydromethanophenazine:CoB-CoM heterodisulfide reductase / hydrogen dehydrogenase / hydrogen dehydrogenase activity / CoB--CoM heterodisulfide reductase activity / 酸化還元酵素; 含硫化合物に対し酸化酵素として働く / hydrogenase (acceptor) / methanogenesis / hydrogenase (acceptor) activity / ferredoxin hydrogenase activity / nickel cation binding / 2 iron, 2 sulfur cluster binding / 4 iron, 4 sulfur cluster binding / oxidoreductase activity / nucleotide binding / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.15 Å 分子置換 / 解像度: 2.15 Å | ||||||
 データ登録者 データ登録者 | Wagner, T. / Koch, J. / Ermler, U. / Shima, S. | ||||||
 引用 引用 |  ジャーナル: Science / 年: 2017 ジャーナル: Science / 年: 2017タイトル: Methanogenic heterodisulfide reductase (HdrABC-MvhAGD) uses two noncubane [4Fe-4S] clusters for reduction. 著者: Wagner, T. / Koch, J. / Ermler, U. / Shima, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5odq.cif.gz 5odq.cif.gz | 1.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5odq.ent.gz pdb5odq.ent.gz | 1.3 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5odq.json.gz 5odq.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5odq_validation.pdf.gz 5odq_validation.pdf.gz | 3.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5odq_full_validation.pdf.gz 5odq_full_validation.pdf.gz | 3.7 MB | 表示 | |
| XML形式データ |  5odq_validation.xml.gz 5odq_validation.xml.gz | 139.7 KB | 表示 | |
| CIF形式データ |  5odq_validation.cif.gz 5odq_validation.cif.gz | 194.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/od/5odq https://data.pdbj.org/pub/pdb/validation_reports/od/5odq ftp://data.pdbj.org/pub/pdb/validation_reports/od/5odq ftp://data.pdbj.org/pub/pdb/validation_reports/od/5odq | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Heterodisulfide reductase, subunit ... , 3種, 6分子 AGBHCI
| #1: タンパク質 | 分子量: 71591.672 Da / 分子数: 2 / 変異: Wild-type / 由来タイプ: 天然 由来: (天然)  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌)細胞株: / / 器官: / / Variant: / / 組織: / 参照: UniProt: A0A2D0TCB9*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase #2: タンパク質 | 分子量: 32075.318 Da / 分子数: 2 / 変異: Wild-type / 由来タイプ: 天然 詳細: This subunit contains two pentacoordinated non cubane [4Fe-4S] cluster 由来: (天然)  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌)細胞株: / / 器官: / / Variant: / / 組織: / 参照: UniProt: A0A2D0TCB4*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase #3: タンパク質 | 分子量: 20483.650 Da / 分子数: 2 / 変異: Wild-type / 由来タイプ: 天然 由来: (天然)  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌)細胞株: / / 器官: / / Variant: / / 組織: / 参照: UniProt: A0A2D0TC97*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase |
|---|
-Methyl-viologen reducing hydrogenase, subunit ... , 3種, 6分子 DJEKFL
| #4: タンパク質 | 分子量: 16057.495 Da / 分子数: 2 / 変異: Wild-type / 由来タイプ: 天然 由来: (天然)  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌)細胞株: / / 器官: / / Variant: / / 組織: / 参照: UniProt: A0A2D0TC98*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase #5: タンパク質 | 分子量: 32511.432 Da / 分子数: 2 / 変異: Wild-type / 由来タイプ: 天然 由来: (天然)  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌)細胞株: / / 器官: / / Variant: / / 組織: / / 参照: UniProt: A0A2D0TC99*PLUS, hydrogenase (acceptor) #6: タンパク質 | 分子量: 53129.602 Da / 分子数: 2 / 変異: Wild-type / 由来タイプ: 天然 詳細: The C-terminus has been post-translationally cleaved 由来: (天然)  Methanothermococcus thermolithotrophicus DSM 2095 (古細菌) Methanothermococcus thermolithotrophicus DSM 2095 (古細菌)細胞株: / / 器官: / / Variant: / / 組織: / / 参照: UniProt: A0A2D0TCA6*PLUS, hydrogenase (acceptor) |
|---|
-非ポリマー , 12種, 970分子 



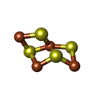



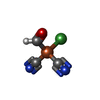










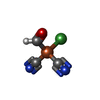



| #7: 化合物 | | #8: 化合物 | ChemComp-ACT / #9: 化合物 | ChemComp-SF4 / #10: 化合物 | ChemComp-GOL / #11: 化合物 | ChemComp-9S8 / #12: 化合物 | ChemComp-9SB / #13: 化合物 | #14: 化合物 | ChemComp-TRS / | #15: 化合物 | #16: 化合物 | #17: 化合物 | #18: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.17 Å3/Da / 溶媒含有率: 43.36 % 解説: Black thick plate square shape of about 0.2-0.4 mm length. |
|---|---|
| 結晶化 | 温度: 294 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: All crystallization was performed in an anaerobic chamber (95% N2/5% H2) with anoxic solution using the sitting drop method (96-well 2-drop MRC Crystallization Plates in polystyrene, ...詳細: All crystallization was performed in an anaerobic chamber (95% N2/5% H2) with anoxic solution using the sitting drop method (96-well 2-drop MRC Crystallization Plates in polystyrene, Molecular Dimensions, Suffolk, UK). The crystallization reservoir contained 100 mM Tris/HCl, pH 8.5, 30% (w/v) polyethylene glycol 4000, and 200 mM Na acetate trihydrate. Crystallization drop contained 1 ul HdrABC-MvhAGD at 25 mg/ml premixed with 2 mM FAD and 1 ul of precipitant. The soaking experiment with the bromoethanesulfonate was done by soaking the crystals in the same crystallization solution supplemented with 66 mM of bromoethanesulfonate for 7 min. The crystal has been cryo-protected using the the mother liquor solution supplemented with 30% glycerol and 10 mM bromoethanesulfonate. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 0.91941 Å / ビームライン: X10SA / 波長: 0.91941 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2016年6月23日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.91941 Å / 相対比: 1 |
| 反射 | 解像度: 2.15→48.85 Å / Num. obs: 241487 / % possible obs: 100 % / 冗長度: 6.9 % / Biso Wilson estimate: 17.13 Å2 / CC1/2: 0.991 / Rmerge(I) obs: 0.21 / Rpim(I) all: 0.086 / Net I/σ(I): 8 |
| 反射 シェル | 解像度: 2.15→2.27 Å / 冗長度: 6.8 % / Rmerge(I) obs: 0.606 / Mean I/σ(I) obs: 3.3 / Num. unique obs: 35106 / CC1/2: 0.327 / Rpim(I) all: 0.249 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 5ODC 解像度: 2.15→48.68 Å / Cor.coef. Fo:Fc: 0.9051 / Cor.coef. Fo:Fc free: 0.8848 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.217 / SU Rfree Blow DPI: 0.167
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 33.25 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.291 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.15→48.68 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.15→2.21 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj























