[English] 日本語
 Yorodumi
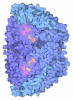
Yorodumi- PDB-5odr: Heterodisulfide reductase / [NiFe]-hydrogenase complex from Metha... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5odr | ||||||
|---|---|---|---|---|---|---|---|
| Title | Heterodisulfide reductase / [NiFe]-hydrogenase complex from Methanothermococcus thermolithotrophicus soaked with heterodisulfide for 2 minutes. | ||||||
 Components Components |
| ||||||
 Keywords Keywords | OXIDOREDUCTASE / Heterodisulfide reductase / [NiFe]-hydrogenase / FeS cluster / ferredoxin / CCG motif / methanogenesis / flavoprotein / flavin-based electron bifurcation / [2Fe-2S] cluster / macromolecular complex / anaerobic / thioredoxin / metabolism | ||||||
| Function / homology |  Function and homology information Function and homology informationdihydromethanophenazine:CoB-CoM heterodisulfide reductase / hydrogen dehydrogenase / hydrogen dehydrogenase activity / CoB--CoM heterodisulfide reductase activity / hydrogenase (acceptor) / methanogenesis / hydrogenase (acceptor) activity / Oxidoreductases; Acting on a sulfur group of donors / ferredoxin hydrogenase activity / nickel cation binding ...dihydromethanophenazine:CoB-CoM heterodisulfide reductase / hydrogen dehydrogenase / hydrogen dehydrogenase activity / CoB--CoM heterodisulfide reductase activity / hydrogenase (acceptor) / methanogenesis / hydrogenase (acceptor) activity / Oxidoreductases; Acting on a sulfur group of donors / ferredoxin hydrogenase activity / nickel cation binding / 2 iron, 2 sulfur cluster binding / 4 iron, 4 sulfur cluster binding / oxidoreductase activity / nucleotide binding / metal ion binding / plasma membrane Similarity search - Function | ||||||
| Biological species |  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.2 Å MOLECULAR REPLACEMENT / Resolution: 2.2 Å | ||||||
 Authors Authors | Wagner, T. / Koch, J. / Ermler, U. / Shima, S. | ||||||
 Citation Citation |  Journal: Science / Year: 2017 Journal: Science / Year: 2017Title: Methanogenic heterodisulfide reductase (HdrABC-MvhAGD) uses two noncubane [4Fe-4S] clusters for reduction. Authors: Wagner, T. / Koch, J. / Ermler, U. / Shima, S. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5odr.cif.gz 5odr.cif.gz | 1.6 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5odr.ent.gz pdb5odr.ent.gz | 1.3 MB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5odr.json.gz 5odr.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/od/5odr https://data.pdbj.org/pub/pdb/validation_reports/od/5odr ftp://data.pdbj.org/pub/pdb/validation_reports/od/5odr ftp://data.pdbj.org/pub/pdb/validation_reports/od/5odr | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5odcSC  5odhC  5odiC  5odqC S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Heterodisulfide reductase, subunit ... , 3 types, 6 molecules AGBHCI
| #1: Protein | Mass: 71591.672 Da / Num. of mol.: 2 / Mutation: Wild-type / Source method: isolated from a natural source Source: (natural)  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea)References: UniProt: A0A2D0TCB9*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase #2: Protein | Mass: 32075.318 Da / Num. of mol.: 2 / Mutation: Wild-type / Source method: isolated from a natural source Source: (natural)  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea)References: UniProt: A0A2D0TCB4*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase #3: Protein | Mass: 20483.650 Da / Num. of mol.: 2 / Mutation: Wild-type / Source method: isolated from a natural source Source: (natural)  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea)References: UniProt: A0A2D0TC97*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase |
|---|
-Methyl-viologen reducing hydrogenase, subunit ... , 3 types, 6 molecules DJEKFL
| #4: Protein | Mass: 16057.495 Da / Num. of mol.: 2 / Mutation: Wild-type / Source method: isolated from a natural source Source: (natural)  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea)References: UniProt: A0A2D0TC98*PLUS, dihydromethanophenazine:CoB-CoM heterodisulfide reductase #5: Protein | Mass: 32511.432 Da / Num. of mol.: 2 / Mutation: Wild-type / Source method: isolated from a natural source Source: (natural)  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea)References: UniProt: A0A2D0TC99*PLUS, hydrogenase (acceptor) #6: Protein | Mass: 53129.602 Da / Num. of mol.: 2 / Mutation: Wild-type / Source method: isolated from a natural source Details: The C-terminus has been post-translationally cleaved. Source: (natural)  Methanothermococcus thermolithotrophicus DSM 2095 (archaea) Methanothermococcus thermolithotrophicus DSM 2095 (archaea)References: UniProt: A0A2D0TCA6*PLUS, hydrogenase (acceptor) |
|---|
-Non-polymers , 13 types, 1133 molecules 


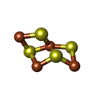
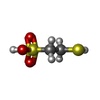



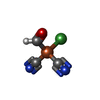















| #7: Chemical | | #8: Chemical | ChemComp-SF4 / #9: Chemical | ChemComp-GOL / #10: Chemical | ChemComp-9S8 / #11: Chemical | #12: Chemical | ChemComp-SO4 / | #13: Chemical | #14: Chemical | ChemComp-ACT / #15: Chemical | #16: Chemical | ChemComp-PE3 / #17: Chemical | #18: Chemical | ChemComp-TP7 / | #19: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.18 Å3/Da / Density % sol: 43.62 % Description: Black thick plate square shape of about 0.2 - 0.4 mm length. |
|---|---|
| Crystal grow | Temperature: 294 K / Method: vapor diffusion, sitting drop / pH: 8.5 Details: All crystallization was performed in an anaerobic chamber (95% N2/5% H2) with anoxic solution using the sitting drop method (96-well 2-drop MRC Crystallization Plates in polystyrene, ...Details: All crystallization was performed in an anaerobic chamber (95% N2/5% H2) with anoxic solution using the sitting drop method (96-well 2-drop MRC Crystallization Plates in polystyrene, Molecular Dimensions, Suffolk, UK). The crystallization reservoir contained 100 mM Tris/HCl, pH 8.5, 30% (w/v) polyethylene glycol 4000, and 200 mM Na acetate trihydrate. Crystallization drop contained 1 ul HdrABC-MvhAGD at 25 mg/ml premixed with 2 mM FAD and 1 ul of precipitant. Crystal was soaked for 2 minutes in the crystallization solution supplemented with 66 mM of heterodisulfide (CoM-S-S-CoB). Then, the crystal was transferred in a solution containing the mother liquor with 30% glycerol and 10 mM CoM-S-S-CoB prior freezing. |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SLS SLS  / Beamline: X10SA / Wavelength: 0.97916 Å / Beamline: X10SA / Wavelength: 0.97916 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Jun 23, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.97916 Å / Relative weight: 1 |
| Reflection | Resolution: 2.2→93.36 Å / Num. obs: 224984 / % possible obs: 99.4 % / Redundancy: 4.3 % / Biso Wilson estimate: 35.26 Å2 / CC1/2: 0.983 / Rmerge(I) obs: 0.181 / Rpim(I) all: 0.096 / Net I/σ(I): 4.6 |
| Reflection shell | Resolution: 2.2→2.32 Å / Redundancy: 4 % / Rmerge(I) obs: 0.897 / Mean I/σ(I) obs: 1.3 / Num. unique obs: 32472 / CC1/2: 0.563 / Rpim(I) all: 0.496 / % possible all: 98.7 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5ODC Resolution: 2.2→90.26 Å / Cor.coef. Fo:Fc: 0.9242 / Cor.coef. Fo:Fc free: 0.9148 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.252 / SU Rfree Blow DPI: 0.183
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 52.38 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.335 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: 1 / Resolution: 2.2→90.26 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.2→2.26 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller








 PDBj
PDBj