+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5mg9 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Putative Ancestral Mamba toxin 1 (AncTx1-W28R/I38S) | ||||||
 要素 要素 | AncTx1-W28R/I38S | ||||||
 キーワード キーワード | TOXIN / Ancestral mamba snake toxin / three-finger fold / Ancestral toxin resurrection and engineering / aminergic toxin | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Dendroaspis angusticeps (コブラ) Dendroaspis angusticeps (コブラ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.801 Å 分子置換 / 解像度: 1.801 Å | ||||||
 データ登録者 データ登録者 | Stura, E.A. / Tepshi, L. / Blanchet, G. / Mourier, G. / Servent, D. | ||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2017 ジャーナル: Sci Rep / 年: 2017タイトル: Ancestral protein resurrection and engineering opportunities of the mamba aminergic toxins. 著者: Blanchet, G. / Alili, D. / Protte, A. / Upert, G. / Gilles, N. / Tepshi, L. / Stura, E.A. / Mourier, G. / Servent, D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5mg9.cif.gz 5mg9.cif.gz | 45.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5mg9.ent.gz pdb5mg9.ent.gz | 31.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5mg9.json.gz 5mg9.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mg/5mg9 https://data.pdbj.org/pub/pdb/validation_reports/mg/5mg9 ftp://data.pdbj.org/pub/pdb/validation_reports/mg/5mg9 ftp://data.pdbj.org/pub/pdb/validation_reports/mg/5mg9 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  4iyeS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 7255.345 Da / 分子数: 1 / 由来タイプ: 合成 詳細: Putative ancetral mamba toxin, Residues 1 to 6 (LEU THR CYS VAL LYS SER ) match to UNP Q8QGR0 NXM11_DENAN 22 to 28, Residues 7 to 11 (LYS SER ILE PHE GLY ) match to UNP P85092 TXAD1_DENAN 7 ...詳細: Putative ancetral mamba toxin, Residues 1 to 6 (LEU THR CYS VAL LYS SER ) match to UNP Q8QGR0 NXM11_DENAN 22 to 28, Residues 7 to 11 (LYS SER ILE PHE GLY ) match to UNP P85092 TXAD1_DENAN 7 to 11, Residues 12 to 17 (VAL THR THR GLU ASP CYS ) match to UNP P18328 TXM2_DENAN 12 to 17, Residues 18 to 26 (PRO ASP GLY GLN ASN LEU CYS PHE LYS ) match to UNP P81030 TXM1_DENAN 18 to 26, Residues 27 to 31 (ARG ARG HIS TYR ILE ) match to UNP P86419 3SI1B_DENAN 27 to 31, Residues 32 to 34 (VAL PRO LYS ) match to UNP P85092 TXAD1_DENAN 32 to 34, Residues 35 to 37 (MET TYR ASP ) match to UNP Q8QGR0 NXM11_DENAN 56 to 58, Residues 38 to 48 (SER THR ARG GLY CYS ALA ALA THR CYS PRO ILE ) match to UNP P85092 TXAD1_DENAN 38 to 48, Residues 49 to 50 (ALA GLU ) match to UNP Q8QGR0 NXM11_DENAN 61 to 62, Residues 51 to 54 (ASN ARG ASP VAL ) match to UNP P82462 3SUC1_NAJKA 51 to 54, Residues 55 to 58 (ILE HIS CYS CYS ) match to UNP P85092 TXAD1_DENAN 55 to 58, Residues 59 to 62 (GLY THR ASP LYS ) match to UNP P18328 TXM2_DENAN 59 to 62, Residues 63 to 65 (CYS ASN GLU ) match to UNP P85092 TXAD1_DENAN 63 to 65, 由来: (合成)  Dendroaspis angusticeps (コブラ) / 参照: UniProt: P85092*PLUS Dendroaspis angusticeps (コブラ) / 参照: UniProt: P85092*PLUS |
|---|
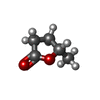
-非ポリマー , 5種, 94分子 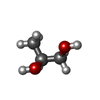







| #2: 化合物 | ChemComp-PGO / | ||||
|---|---|---|---|---|---|
| #3: 化合物 | ChemComp-EDO / | ||||
| #4: 化合物 | ChemComp-SO4 / #5: 化合物 | ChemComp-YVR / | #6: 水 | ChemComp-HOH / | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.25 Å3/Da / 溶媒含有率: 45.44 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5.5 詳細: Protein: lyophilized synthetic toxin at 5 mg/mL in 1 M Na Acetate pH 5.5. Precipitant: 1.9 M ammonium sulfate, 4 % MPD, 2 % 1,4-dioxane, 2 % gamma-valerolactone, 0.132 M sodium citrate, pH 5. ...詳細: Protein: lyophilized synthetic toxin at 5 mg/mL in 1 M Na Acetate pH 5.5. Precipitant: 1.9 M ammonium sulfate, 4 % MPD, 2 % 1,4-dioxane, 2 % gamma-valerolactone, 0.132 M sodium citrate, pH 5.5. Cryoprotectant: 80% saturated lithium sulfate, 10% Dioxane, 10% gamma-valerolactone PH範囲: 5.5 / Temp details: Cooled incubator |
-データ収集
| 回折 | 平均測定温度: 100 K / Ambient temp details: cryostream |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: MASSIF-1 / 波長: 0.965 Å / ビームライン: MASSIF-1 / 波長: 0.965 Å |
| 検出器 | タイプ: DECTRIS PILATUS 2M / 検出器: PIXEL / 日付: 2015年1月29日 詳細: Compound Refractive Lens Fully automatic data collection |
| 放射 | モノクロメーター: diamond beam splitter / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.965 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→35.556 Å / Num. obs: 11687 / % possible obs: 100 % / Observed criterion σ(F): 0 / Observed criterion σ(I): -3 / 冗長度: 8.3 % / CC1/2: 0.999 / Rmerge(I) obs: 0.111 / Rsym value: 0.104 / Net I/σ(I): 12.24 |
| 反射 シェル | 解像度: 1.8→1.91 Å / 冗長度: 8.2 % / Rmerge(I) obs: 1.53 / Mean I/σ(I) obs: 1.16 / CC1/2: 0.719 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 4IYE 解像度: 1.801→35.556 Å / SU ML: 0.29 / 交差検証法: THROUGHOUT / σ(F): 1.34 / 位相誤差: 30.62 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.801→35.556 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: 20.7852 Å / Origin y: -8.891 Å / Origin z: -8.7359 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





