+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5cgc | ||||||
|---|---|---|---|---|---|---|---|
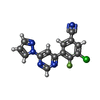
| タイトル | Structure of the human class C GPCR metabotropic glutamate receptor 5 transmembrane domain in complex with the negative allosteric modulator 3-chloro-4-fluoro-5-[6-(1H-pyrazol-1-yl)pyrimidin-4-yl]benzonitrile | ||||||
 要素 要素 | Metabotropic glutamate receptor 5,Endolysin,Metabotropic glutamate receptor 5 | ||||||
 キーワード キーワード | SIGNALING PROTEIN / 7TM / Receptor / GPCR / Membrane-Protein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報A2A adenosine receptor binding / : / G protein-coupled receptor activity involved in regulation of postsynaptic membrane potential / adenylate cyclase inhibiting G protein-coupled glutamate receptor activity / phospholipase C-activating G protein-coupled glutamate receptor signaling pathway / positive regulation of long-term neuronal synaptic plasticity / desensitization of G protein-coupled receptor signaling pathway / G protein-coupled glutamate receptor signaling pathway / Class C/3 (Metabotropic glutamate/pheromone receptors) / glutamate receptor activity ...A2A adenosine receptor binding / : / G protein-coupled receptor activity involved in regulation of postsynaptic membrane potential / adenylate cyclase inhibiting G protein-coupled glutamate receptor activity / phospholipase C-activating G protein-coupled glutamate receptor signaling pathway / positive regulation of long-term neuronal synaptic plasticity / desensitization of G protein-coupled receptor signaling pathway / G protein-coupled glutamate receptor signaling pathway / Class C/3 (Metabotropic glutamate/pheromone receptors) / glutamate receptor activity / Neurexins and neuroligins / astrocyte projection / protein tyrosine kinase activator activity / regulation of synaptic transmission, glutamatergic / viral release from host cell by cytolysis / peptidoglycan catabolic process / positive regulation of calcium-mediated signaling / protein tyrosine kinase binding / dendritic shaft / learning / locomotory behavior / synapse organization / postsynaptic density membrane / G protein-coupled receptor activity / Schaffer collateral - CA1 synapse / cognition / cellular response to amyloid-beta / cell wall macromolecule catabolic process / lysozyme / lysozyme activity / dendritic spine / G alpha (q) signalling events / chemical synaptic transmission / host cell cytoplasm / learning or memory / positive regulation of MAPK cascade / defense response to bacterium / neuronal cell body / dendrite / regulation of DNA-templated transcription / glutamatergic synapse / identical protein binding / plasma membrane / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.101 Å 分子置換 / 解像度: 3.101 Å | ||||||
 データ登録者 データ登録者 | Christopher, J.A. / Aves, S.J. / Bennett, K.A. / Dore, A.S. / Errey, J.C. / Jazayeri, A. / Marshall, F.H. / Okrasa, K. / Serrano-Vega, M.J. / Tehan, B.G. ...Christopher, J.A. / Aves, S.J. / Bennett, K.A. / Dore, A.S. / Errey, J.C. / Jazayeri, A. / Marshall, F.H. / Okrasa, K. / Serrano-Vega, M.J. / Tehan, B.G. / Wiggin, G.R. / Congreve, M. | ||||||
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2015 ジャーナル: J.Med.Chem. / 年: 2015タイトル: Fragment and Structure-Based Drug Discovery for a Class C GPCR: Discovery of the mGlu5 Negative Allosteric Modulator HTL14242 (3-Chloro-5-[6-(5-fluoropyridin-2-yl)pyrimidin-4-yl]benzonitrile). 著者: Christopher, J.A. / Aves, S.J. / Bennett, K.A. / Dore, A.S. / Errey, J.C. / Jazayeri, A. / Marshall, F.H. / Okrasa, K. / Serrano-Vega, M.J. / Tehan, B.G. / Wiggin, G.R. / Congreve, M. #1:  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Structure of class C GPCR metabotropic glutamate receptor 5 transmembrane domain. 著者: Dore, A.S. / Okrasa, K. / Patel, J.C. / Serrano-Vega, M. / Bennett, K. / Cooke, R.M. / Errey, J.C. / Jazayeri, A. / Khan, S. / Tehan, B. / Weir, M. / Wiggin, G.R. / Marshall, F.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5cgc.cif.gz 5cgc.cif.gz | 100.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5cgc.ent.gz pdb5cgc.ent.gz | 73.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5cgc.json.gz 5cgc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cg/5cgc https://data.pdbj.org/pub/pdb/validation_reports/cg/5cgc ftp://data.pdbj.org/pub/pdb/validation_reports/cg/5cgc ftp://data.pdbj.org/pub/pdb/validation_reports/cg/5cgc | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 49802.531 Da / 分子数: 1 変異: E579A N667Y I669A G675M C1054T C1097A T742A S753A,E579A N667Y I669A G675M C1054T C1097A T742A S753A,E579A N667Y I669A G675M C1054T C1097A T742A S753A 由来タイプ: 組換発現 詳細: A chimera of the human mGluR5 gene and T4-Lysozyme from enterobacteria phage T4 in complex with a new chemical entity (NCE): 3-chloro-4-fluoro-5-[6-(1H-pyrazol-1-yl)pyrimidin-4-yl]benzonitrile 由来: (組換発現)  Homo sapiens (ヒト), (組換発現) Homo sapiens (ヒト), (組換発現)  Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ)遺伝子: GRM5, GPRC1E, MGLUR5 / 細胞株 (発現宿主): Sf21 発現宿主:  参照: UniProt: P41594, UniProt: P00720, lysozyme | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| #2: 化合物 | ChemComp-OLA / #3: 化合物 | ChemComp-MES / | #4: 化合物 | ChemComp-51D / | #5: 水 | ChemComp-HOH / | Has protein modification | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.53 Å3/Da / 溶媒含有率: 51.37 % |
|---|---|
| 結晶化 | 温度: 293.1 K / 手法: 脂質キュービック相法 / pH: 6.8 詳細: 24-34% V/V PEG400, 0.2 M AMMONIUM PHOSPHATE DIBASIC, 0.1 M MES, PH 6.8, Temp details: Constant |
-データ収集
| 回折 | 平均測定温度: 200 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I24 / 波長: 0.96861 Å / ビームライン: I24 / 波長: 0.96861 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2014年5月22日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.96861 Å / 相対比: 1 |
| 反射 | 解像度: 3.1→32.8 Å / Num. obs: 8350 / % possible obs: 91.4 % / 冗長度: 3 % / Biso Wilson estimate: 49.91 Å2 / Rmerge(I) obs: 0.194 / Net I/σ(I): 7 |
| 反射 シェル | 解像度: 3.1→3.31 Å / 冗長度: 3 % / Rmerge(I) obs: 0.522 / Mean I/σ(I) obs: 2.4 / % possible all: 93.4 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 4OO9 解像度: 3.101→29.735 Å / SU ML: 0.37 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 29.86 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 48.3 Å2 | ||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.101→29.735 Å
| ||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj