+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5185 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Helical Reconstruction of TMV | |||||||||
 マップデータ マップデータ | Helically symmetrized volume of Tobacco Mosaic Virus. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TMV / helical / RNA | |||||||||
| 機能・相同性 | Tobacco mosaic virus-like, coat protein / Tobacco mosaic virus-like, coat protein / Tobacco mosaic virus-like, coat protein superfamily / Virus coat protein (TMV like) / helical viral capsid / structural molecule activity / identical protein binding / Capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Ge P / Zhou ZH | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2011 ジャーナル: Proc Natl Acad Sci U S A / 年: 2011タイトル: Hydrogen-bonding networks and RNA bases revealed by cryo electron microscopy suggest a triggering mechanism for calcium switches. 著者: Peng Ge / Z Hong Zhou /  要旨: Helical assemblies such as filamentous viruses, flagella, and F-actin represent an important category of structures in biology. As the first discovered virus, tobacco mosaic virus (TMV) was at the ...Helical assemblies such as filamentous viruses, flagella, and F-actin represent an important category of structures in biology. As the first discovered virus, tobacco mosaic virus (TMV) was at the center of virus research. Previously, the structure of TMV was solved at atomic detail by X-ray fiber diffraction but only for its dormant or high-calcium-concentration state, not its low-calcium-concentration state, which is relevant to viral assembly and disassembly inside host cells. Here we report a helical reconstruction of TMV in its calcium-free, metastable assembling state at 3.3 Å resolution by cryo electron microscopy, revealing both protein side chains and RNA bases. An atomic model was built de novo showing marked differences from the high-calcium, dormant-state structure. Although it could be argued that there might be inaccuracies in the latter structure derived from X-ray fiber diffraction, these differences can be interpreted as conformational changes effected by calcium-driven switches, a common regulatory mechanism in plant viruses. Our comparisons of the structures of the low- and high-calcium states indicate that hydrogen bonds formed by Asp116 and Arg92 in the place of the calcium ion of the dormant (high-calcium) state might trigger allosteric changes in the RNA base-binding pockets of the coat protein. In turn, the coat protein-RNA interactions in our structure favor an adenine-X-guanine (A*G) motif over the G*A motif of the dormant state, thus offering an explanation underlying viral assembly initiation by an AAG motif. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5185.map.gz emd_5185.map.gz | 483.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5185-v30.xml emd-5185-v30.xml emd-5185.xml emd-5185.xml | 9.8 KB 9.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5185_1.png emd_5185_1.png | 224.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5185 http://ftp.pdbj.org/pub/emdb/structures/EMD-5185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5185 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5185_validation.pdf.gz emd_5185_validation.pdf.gz | 378.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5185_full_validation.pdf.gz emd_5185_full_validation.pdf.gz | 378.5 KB | 表示 | |
| XML形式データ |  emd_5185_validation.xml.gz emd_5185_validation.xml.gz | 8.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5185 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5185 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5185.map.gz / 形式: CCP4 / 大きさ: 500 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5185.map.gz / 形式: CCP4 / 大きさ: 500 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helically symmetrized volume of Tobacco Mosaic Virus. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.869 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
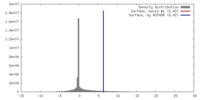
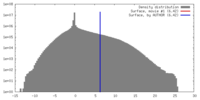
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tobacco Mosaic Virus (Vulgare)
| 全体 | 名称:  Tobacco Mosaic Virus (Vulgare) (ウイルス) Tobacco Mosaic Virus (Vulgare) (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: Tobacco Mosaic Virus (Vulgare)
| 超分子 | 名称: Tobacco Mosaic Virus (Vulgare) / タイプ: sample / ID: 1000 / 集合状態: helix / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 37.8 MDa |
-分子 #1: Coat Protein
| 分子 | 名称: Coat Protein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Coat Protein / コピー数: 2160 / 集合状態: helix / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) / 株: vulgare |
| 分子量 | 理論値: 17.3 MDa |
| 配列 | GO: helical viral capsid / InterPro: Tobacco mosaic virus-like, coat protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 10mM Tris, 130mM NaCl, pH 7.4 |
|---|---|
| グリッド | 詳細: Quantifoil 1.3/1.2 400 mesh |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 93 K / 装置: FEI VITROBOT MARK IV 詳細: Vitrification instrument: FEI Vitrobot Mark IV. 2 uL sample 手法: 10 second wait time, 1-2 second blot time, 1-2 second drain time, blot force 1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 平均: 90 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 250,000 times magnification Legacy - Electron beam tilt params: 0 |
| 日付 | 2009年6月15日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 0.869 µm / 実像数: 386 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 73000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.86 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IHRSR, EMAN |
|---|---|
| CTF補正 | 詳細: EMAN |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)