+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 BA-2.87.1 Spike ectodomain | |||||||||
 マップデータ マップデータ | BA2-87.1 cryoEM map C3 symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Viral protein / immune system / SARS-CoV-2 / RBD / Spike / glycoprotein / BA.2.87.1 / receptor / coronavirus-2 / N-terminal domain / Supersite | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virion component / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ...virion component / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / membrane fusion / Attachment and Entry / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / symbiont-mediated suppression of host innate immune response / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Ren J / Stuart DI / Duyvesteyn HME | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Concerted deletions eliminate a neutralising supersite in SARS-CoV-2 BA.2.87.1 spike 著者: Stuart DI / Ren J #1:  ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr. ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr.年: 2019 タイトル: Macromolecular structure determination using X-rays, neutrons and electrons: recent developments in Phenix 著者: Liebschner D / Afonine PV #2:  ジャーナル: Nature Methods / 年: 2017 ジャーナル: Nature Methods / 年: 2017タイトル: cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination 著者: Punjani A / Rubinstein JL | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50263.map.gz emd_50263.map.gz | 151.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50263-v30.xml emd-50263-v30.xml emd-50263.xml emd-50263.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_50263.png emd_50263.png | 85.5 KB | ||
| Filedesc metadata |  emd-50263.cif.gz emd-50263.cif.gz | 7.4 KB | ||
| その他 |  emd_50263_half_map_1.map.gz emd_50263_half_map_1.map.gz emd_50263_half_map_2.map.gz emd_50263_half_map_2.map.gz | 147.6 MB 151 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50263 http://ftp.pdbj.org/pub/emdb/structures/EMD-50263 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50263 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50263 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9f9yMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50263.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50263.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BA2-87.1 cryoEM map C3 symmetry | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.7303 Å | ||||||||||||||||||||||||||||||||||||
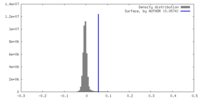
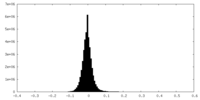
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: BA2-87.1 cryoEM half map C3 symmetry
| ファイル | emd_50263_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BA2-87.1 cryoEM half map C3 symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
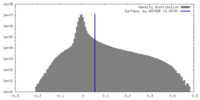
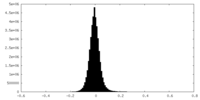
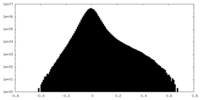
| 密度ヒストグラム |
-ハーフマップ: BA2-87.1 cryoEM half map C3 symmetry
| ファイル | emd_50263_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BA2-87.1 cryoEM half map C3 symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
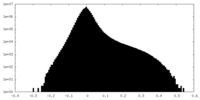
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BA-2.87.1 variant SARS-CoV2 S protein
| 全体 | 名称: BA-2.87.1 variant SARS-CoV2 S protein |
|---|---|
| 要素 |
|
-超分子 #1: BA-2.87.1 variant SARS-CoV2 S protein
| 超分子 | 名称: BA-2.87.1 variant SARS-CoV2 S protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Spike protein recombinantly expressed using sequence of human-derived BA-2.87.1 variant of SARS-CoV2. |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Spike glycoprotein,Fibritin
| 分子 | 名称: Spike glycoprotein,Fibritin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 139.502484 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQSYTNSF TRGVYYPDKV FRSSVLHSTQ DLFLPFFSNV TWFHAISGTN DTKRFDNPVL PFNDGVYFAS TEKFNIIRG WIFGTTLDSK TQSLLIVNNA TNAVIKVCEF QFKNNKSLME SEFRVYSSAN NCTFEYVSQP FLMDLEGKQG N FKNLSEFV ...文字列: MFVFLVLLPL VSSQSYTNSF TRGVYYPDKV FRSSVLHSTQ DLFLPFFSNV TWFHAISGTN DTKRFDNPVL PFNDGVYFAS TEKFNIIRG WIFGTTLDSK TQSLLIVNNA TNAVIKVCEF QFKNNKSLME SEFRVYSSAN NCTFEYVSQP FLMDLEGKQG N FKNLSEFV FKNIDGYFKI YSKHTPINLG RGLPQGFSAL EPLVDLPIGI NITRFQTLLA LHRSYLTPGD SSSGWTAGAA AY YVGYLQP RTFLLKYNEN GTITDAVDCA LDPLSETKCT LKSFTVEKGI YQTSNFRVQP TESIVRFPNI TNLCPFDEVF NAT RFASVY AWNRKRISNC VADYSVLYNF APFFAFKCYG VSPTKLNDLC FTNVYADSFV IRGNEVSQIA PGQTGTIADY NYKL PDDFT GCVIAWNSNK LDSNGGGNYN YMYRLFRKSK LKPFERDIST EIYQAGNTPC KGVAGFNCYF PLQSYGFRPT YGVGH QPYR VVVLSFELLH APATVCGPKK STNLVKNKCV NFNFNGLTGT GVLTESNKKF LPFQQFGRDI ADTTDAVRDP QTLEIL DIT PCSFGGVSVI TPGTNTSNQV AVLYQGVNCT EVSVAIHADQ LTPTWRVYST GSNGFQTRAG CLIGAEYVNN SYECDIP IG AGICASYQTQ TRSHGSASSV ASQPIIAYTM SLGAENSVAY SNNSIAIPTN FTISVTTEIL PVSMTKTSVD CTMYICGD S TECSNLLLQY GSFCTQLKRA LTGIAVEQDK NTQEVFAQVK QIYKIPPIKH FGGFNFSQIL PDPSKPSKRS FIEDLLFNK VTLADAGFIK QYGDCLGDIA ARDLICAQKF NGLTVLPPLL TDEMIAQYTS ALLAGTITSG WTFGAGAALQ IPFAMQMAYR FNGIGVTQN VLYENQKLIA NQFNSAIGKI QGSLSSTASA LGKLQDVVNH NAQALNTLVK QLSSKFGAIS SVLNDILSRL D PPEAEVQI DRLITGRLQS LQTYVTQQLI RAAEIRASAN LAATKMSECV LGQSKRVDFC GKGYHLMSFP QSAPHGVVFL HV TYVPAQE KNFTTAPAIC HDGKAHFPRE GVFVSNGTHW FVTQRNFYEP QIITTDNTFV SGNCDVVIGI VNNTVYDPLQ PEL DSFKEE LDKYFKNHTS PDVDLGDISG INASVVNIQK EIDRLNEVAK NLNESLIDLQ ELGKYEQGSG YIPEAPRDGQ AYVR KDGEW VLLSTFLGRS LEVLFQGPGH HHHHHHHGSA WSHPQFEKGG GSGGGSGGSA WSHPQFEK UniProtKB: Spike glycoprotein, Fibritin |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 39 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | BA.2.87.1 Spike |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 9818 / 平均電子線量: 40.0 e/Å2 / 詳細: Images were collected in EER format. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)