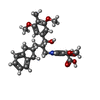
| 登録情報 | データベース: PDB / ID: 4z34
|
|---|
| タイトル | Crystal Structure of Human Lysophosphatidic Acid Receptor 1 in complex with ONO9780307 |
|---|
 要素 要素 | Lysophosphatidic acid receptor 1, Soluble cytochrome b562 |
|---|
 キーワード キーワード | TRANSPORT PROTEIN/inhibitor / human lysophosphatidic acid receptor 1 (LPA1) / G-protein coupled receptor (GPCR) / membrane protein / antagonist / endogenous ligand / PSI-biology / structural genomics / GPCR network / lipidic cubic phase (LCP) / Compound design / polypharmacology / lipid receptor / TRANSPORT PROTEIN-inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cellular response to 1-oleoyl-sn-glycerol 3-phosphate / lysophosphatidic acid receptor activity / positive regulation of smooth muscle cell chemotaxis / Lysosphingolipid and LPA receptors / : / lysophosphatidic acid binding / negative regulation of cilium assembly / regulation of synaptic vesicle cycle / corpus callosum development / bleb assembly ...cellular response to 1-oleoyl-sn-glycerol 3-phosphate / lysophosphatidic acid receptor activity / positive regulation of smooth muscle cell chemotaxis / Lysosphingolipid and LPA receptors / : / lysophosphatidic acid binding / negative regulation of cilium assembly / regulation of synaptic vesicle cycle / corpus callosum development / bleb assembly / oligodendrocyte development / negative regulation of cAMP-mediated signaling / cellular response to oxygen levels / regulation of metabolic process / regulation of postsynaptic neurotransmitter receptor internalization / optic nerve development / positive regulation of Rho protein signal transduction / positive regulation of dendritic spine development / : / G-protein alpha-subunit binding / GABA-ergic synapse / positive regulation of stress fiber assembly / myelination / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / neurogenesis / cerebellum development / cell chemotaxis / dendritic shaft / PDZ domain binding / G protein-coupled receptor activity / electron transport chain / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of neuron projection development / presynaptic membrane / regulation of cell shape / positive regulation of cytosolic calcium ion concentration / G alpha (i) signalling events / G alpha (q) signalling events / postsynaptic membrane / positive regulation of canonical NF-kappaB signal transduction / positive regulation of MAPK cascade / dendritic spine / periplasmic space / electron transfer activity / endosome / iron ion binding / positive regulation of apoptotic process / G protein-coupled receptor signaling pathway / neuronal cell body / glutamatergic synapse / heme binding / cell surface / plasma membrane / cytoplasm類似検索 - 分子機能 Lysophosphatidic acid receptor EDG-2 / Lysophosphatidic acid receptor / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like / GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family)類似検索 - ドメイン・相同性 (2S)-2,3-dihydroxypropyl (7Z)-tetradec-7-enoate / Chem-ON7 / Soluble cytochrome b562 / Lysophosphatidic acid receptor 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
  Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3 Å 分子置換 / 解像度: 3 Å |
|---|
 データ登録者 データ登録者 | Chrencik, J.E. / Roth, C.B. / Terakado, M. / Kurata, H. / Omi, R. / Kihara, Y. / Warshaviak, D. / Nakade, S. / Asmar-Rovira, G. / Mileni, M. ...Chrencik, J.E. / Roth, C.B. / Terakado, M. / Kurata, H. / Omi, R. / Kihara, Y. / Warshaviak, D. / Nakade, S. / Asmar-Rovira, G. / Mileni, M. / Mizuno, H. / Griffith, M.T. / Rodgers, C. / Han, G.W. / Velasquez, J. / Chun, J. / Stevens, R.C. / Hanson, M.A. / GPCR Network (GPCR) |
|---|
 引用 引用 |  ジャーナル: Cell / 年: 2015 ジャーナル: Cell / 年: 2015
タイトル: Crystal Structure of Antagonist Bound Human Lysophosphatidic Acid Receptor 1.
著者: Chrencik, J.E. / Roth, C.B. / Terakado, M. / Kurata, H. / Omi, R. / Kihara, Y. / Warshaviak, D. / Nakade, S. / Asmar-Rovira, G. / Mileni, M. / Mizuno, H. / Griffith, M.T. / Rodgers, C. / Han, ...著者: Chrencik, J.E. / Roth, C.B. / Terakado, M. / Kurata, H. / Omi, R. / Kihara, Y. / Warshaviak, D. / Nakade, S. / Asmar-Rovira, G. / Mileni, M. / Mizuno, H. / Griffith, M.T. / Rodgers, C. / Han, G.W. / Velasquez, J. / Chun, J. / Stevens, R.C. / Hanson, M.A. |
|---|
| 履歴 | | 登録 | 2015年3月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年6月3日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年7月1日 | Group: Database references |
|---|
| 改定 1.2 | 2023年9月27日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description / Source and taxonomy / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / diffrn_radiation_wavelength / entity / entity_src_gen / pdbx_entity_nonpoly / pdbx_initial_refinement_model / pdbx_prerelease_seq / pdbx_struct_assembly / pdbx_struct_oper_list / struct_keywords
Item: _chem_comp.name / _database_2.pdbx_DOI ..._chem_comp.name / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _diffrn_radiation_wavelength.wavelength / _entity.pdbx_description / _entity_src_gen.pdbx_alt_source_flag / _pdbx_entity_nonpoly.name / _pdbx_struct_assembly.oligomeric_details / _pdbx_struct_oper_list.symmetry_operation / _struct_keywords.text |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3 Å
分子置換 / 解像度: 3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell / 年: 2015
ジャーナル: Cell / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4z34.cif.gz
4z34.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4z34.ent.gz
pdb4z34.ent.gz PDB形式
PDB形式 4z34.json.gz
4z34.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/z3/4z34
https://data.pdbj.org/pub/pdb/validation_reports/z3/4z34 ftp://data.pdbj.org/pub/pdb/validation_reports/z3/4z34
ftp://data.pdbj.org/pub/pdb/validation_reports/z3/4z34 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト), (組換発現)
Homo sapiens (ヒト), (組換発現) 

 X線回折
X線回折 試料調製
試料調製 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj