登録情報 データベース : EMDB / ID : EMD-4890タイトル Structure of Vaccinia Virus DNA-dependent RNA polymerase co-transcriptional capping complex Post-processed cryo-EM map. 複合体 : Vaccinia Virus DNA-dependent RNA polymerase co-transcriptional capping complex複合体 : DNA-dependent RNA polymerase subunit複合体 : RNA複合体 : syntheticリガンド : x 3種機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 手法 / / 解像度 : 3.1 Å Hillen HS / Bartuli J / Grimm C / Dienemann C / Bedenk K / Szalar A / Fischer U / Cramer P 資金援助 Organization Grant number 国 German Research Foundation SFB860 German Federal Ministry for Education and Research EXC 2067/1- 390729940 German Research Foundation SPP1935 Volkswagen Foundation European Research Council TRANSREGULON German Research Foundation Fi 573 18-1 German Research Foundation Fi 573 7-2
ジャーナル : Cell / 年 : 2019タイトル : Structural Basis of Poxvirus Transcription: Transcribing and Capping Vaccinia Complexes.著者 : Hauke S Hillen / Julia Bartuli / Clemens Grimm / Christian Dienemann / Kristina Bedenk / Aladar A Szalay / Utz Fischer / Patrick Cramer / 要旨 : Poxviruses use virus-encoded multisubunit RNA polymerases (vRNAPs) and RNA-processing factors to generate mG-capped mRNAs in the host cytoplasm. In the accompanying paper, we report structures of ... Poxviruses use virus-encoded multisubunit RNA polymerases (vRNAPs) and RNA-processing factors to generate mG-capped mRNAs in the host cytoplasm. In the accompanying paper, we report structures of core and complete vRNAP complexes of the prototypic Vaccinia poxvirus (Grimm et al., 2019; in this issue of Cell). Here, we present the cryo-electron microscopy (cryo-EM) structures of Vaccinia vRNAP in the form of a transcribing elongation complex and in the form of a co-transcriptional capping complex that contains the viral capping enzyme (CE). The trifunctional CE forms two mobile modules that bind the polymerase surface around the RNA exit tunnel. RNA extends from the vRNAP active site through this tunnel and into the active site of the CE triphosphatase. Structural comparisons suggest that growing RNA triggers large-scale rearrangements on the surface of the transcription machinery during the transition from transcription initiation to RNA capping and elongation. Our structures unravel the basis for synthesis and co-transcriptional modification of poxvirus RNA. 履歴 登録 2019年4月23日 - ヘッダ(付随情報) 公開 2019年12月18日 - マップ公開 2019年12月18日 - 更新 2020年12月2日 - 現状 2020年12月2日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 ポリヌクレオチド-5'-ホスファターゼ / DNA-templated viral transcription / inorganic triphosphate phosphatase activity /
ポリヌクレオチド-5'-ホスファターゼ / DNA-templated viral transcription / inorganic triphosphate phosphatase activity /  virion component => GO:0044423 / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /
virion component => GO:0044423 / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity / viral transcription /
polynucleotide 5'-phosphatase activity / viral transcription /  RNA polymerase I activity /
RNA polymerase I activity /  RNA polymerase II activity ...
RNA polymerase II activity ... ポリヌクレオチド-5'-ホスファターゼ / DNA-templated viral transcription / inorganic triphosphate phosphatase activity /
ポリヌクレオチド-5'-ホスファターゼ / DNA-templated viral transcription / inorganic triphosphate phosphatase activity /  virion component => GO:0044423 / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /
virion component => GO:0044423 / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity / viral transcription /
polynucleotide 5'-phosphatase activity / viral transcription /  RNA polymerase I activity /
RNA polymerase I activity /  RNA polymerase II activity /
RNA polymerase II activity /  DNA-directed RNA polymerase complex /
DNA-directed RNA polymerase complex /  virion component / DNA-templated transcription termination /
virion component / DNA-templated transcription termination /  ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /
ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /  ポリメラーゼ /
ポリメラーゼ /  mRNA guanylyltransferase activity /
mRNA guanylyltransferase activity /  mRNA guanylyltransferase / mRNA (guanine-N7)-methyltransferase / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / DNA-templated transcription / GTP binding /
mRNA guanylyltransferase / mRNA (guanine-N7)-methyltransferase / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / DNA-templated transcription / GTP binding /  DNA binding /
DNA binding /  RNA binding / zinc ion binding /
RNA binding / zinc ion binding /  metal ion binding
metal ion binding
 Vaccinia virus (ワクチニアウイルス)
Vaccinia virus (ワクチニアウイルス) 単粒子再構成法 /
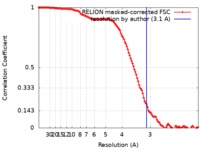
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å
クライオ電子顕微鏡法 / 解像度: 3.1 Å  データ登録者
データ登録者 ドイツ, 7件
ドイツ, 7件  引用
引用 ジャーナル: Cell / 年: 2019
ジャーナル: Cell / 年: 2019

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_4890.map.gz
emd_4890.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-4890-v30.xml
emd-4890-v30.xml emd-4890.xml
emd-4890.xml EMDBヘッダ
EMDBヘッダ emd_4890_fsc.xml
emd_4890_fsc.xml FSCデータファイル
FSCデータファイル emd_4890.png
emd_4890.png emd_4890_msk_1.map
emd_4890_msk_1.map マスクマップ
マスクマップ emd_4890_additional_1.map.gz
emd_4890_additional_1.map.gz emd_4890_additional_2.map.gz
emd_4890_additional_2.map.gz emd_4890_additional_3.map.gz
emd_4890_additional_3.map.gz emd_4890_additional_4.map.gz
emd_4890_additional_4.map.gz emd_4890_half_map_1.map.gz
emd_4890_half_map_1.map.gz emd_4890_half_map_2.map.gz
emd_4890_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-4890
http://ftp.pdbj.org/pub/emdb/structures/EMD-4890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4890
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4890 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_4890.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_4890.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_4890_msk_1.map
emd_4890_msk_1.map 試料の構成要素
試料の構成要素 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000
Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X