+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4567 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Viral pore protein | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | portal / translocase / motor / pore / bacteriophage / thermophage / caudovirales / siphoviridae / virus / VIRAL PROTEIN | |||||||||
| 機能・相同性 | : / Phage P23-45 portal protein / viral capsid / Portal protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Thermus phage P2345 (ファージ) / Thermus phage P2345 (ファージ) /  Thermus virus P23-45 (ウイルス) Thermus virus P23-45 (ウイルス) | |||||||||
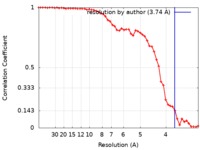
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.74 Å | |||||||||
 データ登録者 データ登録者 | Bayfield OW / Antson AA / Winkler DC / Hesketh EL / Chechik M / Cheng N / Dykeman EC / Minakhin L / Ranson NA / Severinov K / Steven AC | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Cryo-EM structure in situ reveals a molecular switch that safeguards virus against genome loss. 著者: Oliver W Bayfield / Alasdair C Steven / Alfred A Antson /   要旨: The portal protein is a key component of many double-stranded DNA viruses, governing capsid assembly and genome packaging. Twelve subunits of the portal protein define a tunnel, through which DNA is ...The portal protein is a key component of many double-stranded DNA viruses, governing capsid assembly and genome packaging. Twelve subunits of the portal protein define a tunnel, through which DNA is translocated into the capsid. It is unknown how the portal protein functions as a gatekeeper, preventing DNA slippage, whilst allowing its passage into the capsid, and how these processes are controlled. A cryo-EM structure of the portal protein of thermostable virus P23-45, determined in situ in its procapsid-bound state, indicates a mechanism that naturally safeguards the virus against genome loss. This occurs via an inversion of the conformation of the loops that define the constriction in the central tunnel, accompanied by a hydrophilic-hydrophobic switch. The structure also shows how translocation of DNA into the capsid could be modulated by a changing mode of protein-protein interactions between portal and capsid, across a symmetry-mismatched interface. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4567.map.gz emd_4567.map.gz | 9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4567-v30.xml emd-4567-v30.xml emd-4567.xml emd-4567.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4567_fsc.xml emd_4567_fsc.xml | 5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4567.png emd_4567.png | 183.6 KB | ||
| マスクデータ |  emd_4567_msk_1.map emd_4567_msk_1.map | 10 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4567.cif.gz emd-4567.cif.gz | 5.8 KB | ||
| その他 |  emd_4567_half_map_1.map.gz emd_4567_half_map_1.map.gz emd_4567_half_map_2.map.gz emd_4567_half_map_2.map.gz | 5.8 MB 5.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4567 http://ftp.pdbj.org/pub/emdb/structures/EMD-4567 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4567 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4567 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4567.map.gz / 形式: CCP4 / 大きさ: 10 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4567.map.gz / 形式: CCP4 / 大きさ: 10 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5975 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
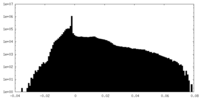
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4567_msk_1.map emd_4567_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
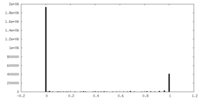
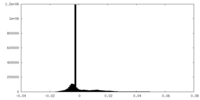
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_4567_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
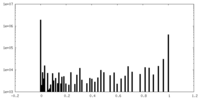
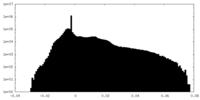
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_4567_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
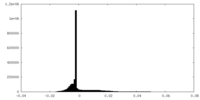
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Thermus phage P2345
| 全体 | 名称:   Thermus phage P2345 (ファージ) Thermus phage P2345 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Thermus phage P2345
| 超分子 | 名称: Thermus phage P2345 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Thermus phage P2345 (ファージ) Thermus phage P2345 (ファージ) |
| 分子量 | 理論値: 596 KDa |
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermus virus P23-45 (ウイルス) Thermus virus P23-45 (ウイルス) |
| 分子量 | 理論値: 49.714141 KDa |
| 配列 | 文字列: MAKRGRKPKE LVPGPGSIDP SDVPKLEGAS VPVMSTSYDV VVDREFDELL QGKDGLLVYH KMLSDGTVKN ALNYIFGRIR SAKWYVEPA STDPEDIAIA AFIHAQLGID DASVGKYPFG RLFAIYENAY IYGMAAGEIV LTLGADGKLI LDKIVPIHPF N IDEVLYDE ...文字列: MAKRGRKPKE LVPGPGSIDP SDVPKLEGAS VPVMSTSYDV VVDREFDELL QGKDGLLVYH KMLSDGTVKN ALNYIFGRIR SAKWYVEPA STDPEDIAIA AFIHAQLGID DASVGKYPFG RLFAIYENAY IYGMAAGEIV LTLGADGKLI LDKIVPIHPF N IDEVLYDE EGGPKALKLS GEVKGGSQFV NGLEIPIWKT VVFLHNDDGS FTGQSALRAA VPHWLAKRAL ILLINHGLER FM IGVPTLT IPKSVRQGTK QWEAAKEIVK NFVQKPRHGI ILPDDWKFDT VDLKSAMPDA IPYLTYHDAG IARALGIDFN TVQ LNMGVQ AVNIGEFVSL TQQTIISLQR EFASAVNLYL IPKLVLPNWP GATRFPRLTF EMEERNDFSA AANLMGMLIN AVKD SEDIP TELKALIDAL PSKMRRALGV VDEVREAVRQ PADSRYLYTR RRR UniProtKB: Portal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均露光時間: 2.0 sec. / 平均電子線量: 99.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 75000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6qjt: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)