+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3jav | ||||||
|---|---|---|---|---|---|---|---|
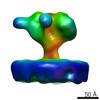
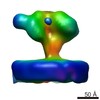
| タイトル | Structure of full-length IP3R1 channel in the apo-state determined by single particle cryo-EM | ||||||
 要素 要素 | Inositol 1,4,5-trisphosphate receptor type 1 | ||||||
 キーワード キーワード | TRANSPORT PROTEIN / inositol 1 / 4 / 5-trisphosphate receptor / calcium release channel / calcium signaling | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / release of sequestered calcium ion into cytosol by endoplasmic reticulum / smooth endoplasmic reticulum membrane / cGMP effects / Elevation of cytosolic Ca2+ levels / platelet dense tubular network / calcineurin complex / platelet dense granule membrane ...Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / release of sequestered calcium ion into cytosol by endoplasmic reticulum / smooth endoplasmic reticulum membrane / cGMP effects / Elevation of cytosolic Ca2+ levels / platelet dense tubular network / calcineurin complex / platelet dense granule membrane / epithelial fluid transport / inositol 1,4,5-trisphosphate-gated calcium channel activity / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / calcium import into the mitochondrion / voluntary musculoskeletal movement / positive regulation of hepatocyte proliferation / inositol 1,4,5 trisphosphate binding / negative regulation of calcium-mediated signaling / positive regulation of calcium ion transport / endoplasmic reticulum calcium ion homeostasis / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / nuclear inner membrane / Ion homeostasis / transport vesicle membrane / dendrite development / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / regulation of cytosolic calcium ion concentration / single fertilization / calcium channel inhibitor activity / release of sequestered calcium ion into cytosol / phosphatidylinositol binding / liver regeneration / secretory granule membrane / synaptic membrane / cellular response to cAMP / sarcoplasmic reticulum / post-embryonic development / positive regulation of neuron projection development / positive regulation of insulin secretion / GABA-ergic synapse / Schaffer collateral - CA1 synapse / cell morphogenesis / calcium ion transport / nuclear envelope / presynapse / positive regulation of cytosolic calcium ion concentration / protein phosphatase binding / protein homotetramerization / cellular response to hypoxia / phospholipase C-activating G protein-coupled receptor signaling pathway / transmembrane transporter binding / response to hypoxia / postsynapse / postsynaptic density / positive regulation of apoptotic process / protein domain specific binding / neuronal cell body / calcium ion binding / dendrite / synapse / endoplasmic reticulum membrane / negative regulation of apoptotic process / protein-containing complex binding / nucleolus / perinuclear region of cytoplasm / endoplasmic reticulum / protein homodimerization activity / protein-containing complex / ATP binding / identical protein binding / membrane / plasma membrane / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | ||||||
 データ登録者 データ登録者 | Fan, G. / Baker, M.L. / Wang, Z. / Baker, M.R. / Sinyagovskiy, P.A. / Chiu, W. / Ludtke, S.J. / Serysheva, I.I. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2015 ジャーナル: Nature / 年: 2015タイトル: Gating machinery of InsP3R channels revealed by electron cryomicroscopy. 著者: Guizhen Fan / Matthew L Baker / Zhao Wang / Mariah R Baker / Pavel A Sinyagovskiy / Wah Chiu / Steven J Ludtke / Irina I Serysheva /  要旨: Inositol-1,4,5-trisphosphate receptors (InsP3Rs) are ubiquitous ion channels responsible for cytosolic Ca(2+) signalling and essential for a broad array of cellular processes ranging from contraction ...Inositol-1,4,5-trisphosphate receptors (InsP3Rs) are ubiquitous ion channels responsible for cytosolic Ca(2+) signalling and essential for a broad array of cellular processes ranging from contraction to secretion, and from proliferation to cell death. Despite decades of research on InsP3Rs, a mechanistic understanding of their structure-function relationship is lacking. Here we present the first, to our knowledge, near-atomic (4.7 Å) resolution electron cryomicroscopy structure of the tetrameric mammalian type 1 InsP3R channel in its apo-state. At this resolution, we are able to trace unambiguously ∼85% of the protein backbone, allowing us to identify the structural elements involved in gating and modulation of this 1.3-megadalton channel. Although the central Ca(2+)-conduction pathway is similar to other ion channels, including the closely related ryanodine receptor, the cytosolic carboxy termini are uniquely arranged in a left-handed α-helical bundle, directly interacting with the amino-terminal domains of adjacent subunits. This configuration suggests a molecular mechanism for allosteric regulation of channel gating by intracellular signals. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3jav.cif.gz 3jav.cif.gz | 1.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3jav.ent.gz pdb3jav.ent.gz | 580 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3jav.json.gz 3jav.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ja/3jav https://data.pdbj.org/pub/pdb/validation_reports/ja/3jav ftp://data.pdbj.org/pub/pdb/validation_reports/ja/3jav ftp://data.pdbj.org/pub/pdb/validation_reports/ja/3jav | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 313657.406 Da / 分子数: 4 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Inositol 1,4,5-trisphosphate receptor, type 1 / タイプ: COMPLEX / 詳細: homotetramer |
|---|---|
| 分子量 | 値: 1.3 MDa / 実験値: NO |
| 緩衝液 | 名称: 50 mM Tris-HCl, 0.4% CHAPS, 150 mM NaCl, 1 mM DTT, 1 mM EGTA, 1 mM EDTA pH: 7.4 詳細: 50 mM Tris-HCl, 0.4% CHAPS, 150 mM NaCl, 1 mM DTT, 1 mM EGTA, 1 mM EDTA |
| 試料 | 濃度: 0.4 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: 400 mesh copper grids with thin carbon support |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / Temp: 120 K / 湿度: 100 % 詳細: Blot once for 3 seconds before plunging into liquid ethane (FEI VITROBOT MARK IV). 手法: blot once for 3 seconds |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI POLARA 300 / 日付: 2014年1月1日 |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 23000 X / 倍率(補正後): 30886 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 600 nm / Cs: 2 mm 非点収差: Objective lens astigmatism was corrected at 100,000 times magnification. |
| 試料ホルダ | 試料ホルダーモデル: GATAN LIQUID NITROGEN / 温度: 100 K / 最高温度: 102 K / 最低温度: 95 K |
| 撮影 | 電子線照射量: 22 e/Å2 / フィルム・検出器のモデル: GATAN K2 (4k x 4k) |
| 電子光学装置 | エネルギーフィルター名称: FEI |
| 画像スキャン | デジタル画像の数: 4160 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: CTFFIND3 | ||||||||||||
| 対称性 | 点対称性: C4 (4回回転対称) | ||||||||||||
| 3次元再構成 | 手法: K-means clustering / 解像度: 4.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 96106 / ピクセルサイズ(公称値): 1.62 Å / ピクセルサイズ(実測値): 1.62 Å / 詳細: Applied symmetry: C4 / 対称性のタイプ: POINT | ||||||||||||
| 精密化ステップ | サイクル: LAST
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


