+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 DMV nsp3-4 pore complex (extended-pore) | |||||||||
 マップデータ マップデータ | local resolution map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Double membrane vesicle / pore complex / nsp3 / nsp4 / RNA transport / VIRAL PROTEIN | |||||||||
| 生物種 |  | |||||||||
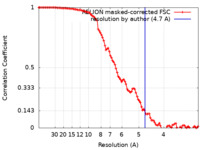
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Huang YX / Zhong LJ / Zhang WX / Ni T | |||||||||
| 資金援助 |  香港, 1件 香港, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Molecular architecture of coronavirus double-membrane vesicle pore complex. 著者: Yixin Huang / Tongyun Wang / Lijie Zhong / Wenxin Zhang / Yu Zhang / Xiulian Yu / Shuofeng Yuan / Tao Ni /  要旨: Coronaviruses remodel the intracellular host membranes during replication, forming double-membrane vesicles (DMVs) to accommodate viral RNA synthesis and modifications. SARS-CoV-2 non-structural ...Coronaviruses remodel the intracellular host membranes during replication, forming double-membrane vesicles (DMVs) to accommodate viral RNA synthesis and modifications. SARS-CoV-2 non-structural protein 3 (nsp3) and nsp4 are the minimal viral components required to induce DMV formation and to form a double-membrane-spanning pore, essential for the transport of newly synthesized viral RNAs. The mechanism of DMV pore complex formation remains unknown. Here we describe the molecular architecture of the SARS-CoV-2 nsp3-nsp4 pore complex, as resolved by cryogenic electron tomography and subtomogram averaging in isolated DMVs. The structures uncover an unexpected stoichiometry and topology of the nsp3-nsp4 pore complex comprising 12 copies each of nsp3 and nsp4, organized in 4 concentric stacking hexamer rings, mimicking a miniature nuclear pore complex. The transmembrane domains are interdigitated to create a high local curvature at the double-membrane junction, coupling double-membrane reorganization with pore formation. The ectodomains form extensive contacts in a pseudo-12-fold symmetry, belting the pore complex from the intermembrane space. A central positively charged ring of arginine residues coordinates the putative RNA translocation, essential for virus replication. Our work establishes a framework for understanding DMV pore formation and RNA translocation, providing a structural basis for the development of new antiviral strategies to combat coronavirus infection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39111.map.gz emd_39111.map.gz | 51.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39111-v30.xml emd-39111-v30.xml emd-39111.xml emd-39111.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39111_fsc.xml emd_39111_fsc.xml | 10.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39111.png emd_39111.png | 104.4 KB | ||
| マスクデータ |  emd_39111_msk_1.map emd_39111_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-39111.cif.gz emd-39111.cif.gz | 4.6 KB | ||
| その他 |  emd_39111_half_map_1.map.gz emd_39111_half_map_1.map.gz emd_39111_half_map_2.map.gz emd_39111_half_map_2.map.gz | 43.6 MB 43.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39111 http://ftp.pdbj.org/pub/emdb/structures/EMD-39111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39111 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39111_validation.pdf.gz emd_39111_validation.pdf.gz | 908.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39111_full_validation.pdf.gz emd_39111_full_validation.pdf.gz | 908.1 KB | 表示 | |
| XML形式データ |  emd_39111_validation.xml.gz emd_39111_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_39111_validation.cif.gz emd_39111_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39111 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39111 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39111.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39111.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ |
| ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_39111_msk_1.map emd_39111_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_39111_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 nsp3-4 pore complex
| 全体 | 名称: SARS-CoV-2 nsp3-4 pore complex |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 nsp3-4 pore complex
| 超分子 | 名称: SARS-CoV-2 nsp3-4 pore complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 150mM NaCl, 10mM Tris-HCl, 1mM EDTA | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: EMS Lacey Carbon / 支持フィルム - 材質: CARBON | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 平均電子線量: 3.0 e/Å2 詳細: The tilt-series were acquired using Thermofisher Krios equipped with a Falcon 4i camera and Selectris energy filter. A dose-symmetric scheme (group of 2) was used, with a tilt range of -51 to ...詳細: The tilt-series were acquired using Thermofisher Krios equipped with a Falcon 4i camera and Selectris energy filter. A dose-symmetric scheme (group of 2) was used, with a tilt range of -51 to 51 (or -60 to 60) at 3 increments and an exposure dose of 3 e-/A2 per image. The total dose was 105 (or 123) e-/A2. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)