+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human erythrocyte catalase with SLS as additive during cryo-EM grid preparation | |||||||||
 マップデータ マップデータ | Relion sharpened full map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Catalase / Oxidoreductase / heme / NADP | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to phenylpropanoid / aminoacylase activity / catalase complex / hemoglobin metabolic process / response to inactivity / cellular detoxification of hydrogen peroxide / oxidoreductase activity, acting on peroxide as acceptor / response to L-ascorbic acid / response to ozone / catalase ...response to phenylpropanoid / aminoacylase activity / catalase complex / hemoglobin metabolic process / response to inactivity / cellular detoxification of hydrogen peroxide / oxidoreductase activity, acting on peroxide as acceptor / response to L-ascorbic acid / response to ozone / catalase / UV protection / response to fatty acid / response to light intensity / response to vitamin A / catalase activity / peroxisomal membrane / ureteric bud development / triglyceride metabolic process / Detoxification of Reactive Oxygen Species / antioxidant activity / peroxisomal matrix / positive regulation of cell division / response to vitamin E / response to hyperoxia / FOXO-mediated transcription of oxidative stress, metabolic and neuronal genes / response to cadmium ion / aerobic respiration / cholesterol metabolic process / response to reactive oxygen species / response to activity / hydrogen peroxide catabolic process / Peroxisomal protein import / response to lead ion / response to insulin / response to hydrogen peroxide / cellular response to growth factor stimulus / osteoblast differentiation / peroxisome / response to estradiol / NADP binding / secretory granule lumen / response to ethanol / ficolin-1-rich granule lumen / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / response to hypoxia / response to xenobiotic stimulus / intracellular membrane-bounded organelle / focal adhesion / heme binding / Neutrophil degranulation / negative regulation of apoptotic process / enzyme binding / protein homodimerization activity / protein-containing complex / mitochondrion / extracellular exosome / extracellular region / identical protein binding / membrane / metal ion binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
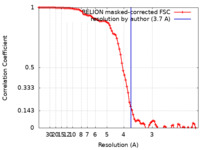
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Yadav S / Vinothkumar KR | |||||||||
| 資金援助 |  インド, 2件 インド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2024 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2024タイトル: Factors affecting macromolecule orientations in thin films formed in cryo-EM. 著者: Swati Yadav / Kutti R Vinothkumar /  要旨: The formation of a vitrified thin film embedded with randomly oriented macromolecules is an essential prerequisite for cryogenic sample electron microscopy. Most commonly, this is achieved using the ...The formation of a vitrified thin film embedded with randomly oriented macromolecules is an essential prerequisite for cryogenic sample electron microscopy. Most commonly, this is achieved using the plunge-freeze method first described nearly 40 years ago. Although this is a robust method, the behaviour of different macromolecules shows great variation upon freezing and often needs to be optimized to obtain an isotropic, high-resolution reconstruction. For a macromolecule in such a film, the probability of encountering the air-water interface in the time between blotting and freezing and adopting preferred orientations is very high. 3D reconstruction using preferentially oriented particles often leads to anisotropic and uninterpretable maps. Currently, there are no general solutions to this prevalent issue, but several approaches largely focusing on sample preparation with the use of additives and novel grid modifications have been attempted. In this study, the effect of physical and chemical factors on the orientations of macromolecules was investigated through an analysis of selected well studied macromolecules, and important parameters that determine the behaviour of proteins on cryo-EM grids were revealed. These insights highlight the nature of the interactions that cause preferred orientations and can be utilized to systematically address orientation bias for any given macromolecule and to provide a framework to design small-molecule additives to enhance sample stability and behaviour. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37952.map.gz emd_37952.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37952-v30.xml emd-37952-v30.xml emd-37952.xml emd-37952.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37952_fsc.xml emd_37952_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37952.png emd_37952.png | 47.2 KB | ||
| Filedesc metadata |  emd-37952.cif.gz emd-37952.cif.gz | 6.3 KB | ||
| その他 |  emd_37952_half_map_1.map.gz emd_37952_half_map_1.map.gz emd_37952_half_map_2.map.gz emd_37952_half_map_2.map.gz | 48.4 MB 48.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37952 http://ftp.pdbj.org/pub/emdb/structures/EMD-37952 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37952 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37952 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37952_validation.pdf.gz emd_37952_validation.pdf.gz | 951 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37952_full_validation.pdf.gz emd_37952_full_validation.pdf.gz | 950.5 KB | 表示 | |
| XML形式データ |  emd_37952_validation.xml.gz emd_37952_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_37952_validation.cif.gz emd_37952_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37952 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37952 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37952 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37952 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8wzhMC  8wv4C  8wv5C  8wv6C  8wziC  8wzjC  8wzkC  8wzmC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37952.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37952.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion sharpened full map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Relion unsharpened half map
| ファイル | emd_37952_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion unsharpened half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Relion unsharpened half map
| ファイル | emd_37952_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion unsharpened half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Catalase purified from human erythrocytes
| 全体 | 名称: Catalase purified from human erythrocytes |
|---|---|
| 要素 |
|
-超分子 #1: Catalase purified from human erythrocytes
| 超分子 | 名称: Catalase purified from human erythrocytes / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 238 KDa |
-分子 #1: Catalase
| 分子 | 名称: Catalase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: catalase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 59.836996 KDa |
| 配列 | 文字列: MADSRDPASD QMQHWKEQRA AQKADVLTTG AGNPVGDKLN VITVGPRGPL LVQDVVFTDE MAHFDRERIP ERVVHAKGAG AFGYFEVTH DITKYSKAKV FEHIGKKTPI AVRFSTVAGE SGSADTVRDP RGFAVKFYTE DGNWDLVGNN TPIFFIRDPI L FPSFIHSQ ...文字列: MADSRDPASD QMQHWKEQRA AQKADVLTTG AGNPVGDKLN VITVGPRGPL LVQDVVFTDE MAHFDRERIP ERVVHAKGAG AFGYFEVTH DITKYSKAKV FEHIGKKTPI AVRFSTVAGE SGSADTVRDP RGFAVKFYTE DGNWDLVGNN TPIFFIRDPI L FPSFIHSQ KRNPQTHLKD PDMVWDFWSL RPESLHQVSF LFSDRGIPDG HRHMNGYGSH TFKLVNANGE AVYCKFHYKT DQ GIKNLSV EDAARLSQED PDYGIRDLFN AIATGKYPSW TFYIQVMTFN QAETFPFNPF DLTKVWPHKD YPLIPVGKLV LNR NPVNYF AEVEQIAFDP SNMPPGIEAS PDKMLQGRLF AYPDTHRHRL GPNYLHIPVN CPYRARVANY QRDGPMCMQD NQGG APNYY PNSFGAPEQQ PSALEHSIQY SGEVRRFNTA NDDNVTQVRA FYVNVLNEEQ RKRLCENIAG HLKDAQIFIQ KKAVK NFTE VHPDYGSHIQ ALLDKYNAEK PKNAIHTFVQ SGSHLAAREK ANL UniProtKB: Catalase |
-分子 #2: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #3: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
| 分子 | 名称: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE タイプ: ligand / ID: 3 / コピー数: 4 / 式: NDP |
|---|---|
| 分子量 | 理論値: 745.421 Da |
| Chemical component information |  ChemComp-NDP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.4 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot force, 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 平均露光時間: 60.0 sec. / 平均電子線量: 33.0 e/Å2 詳細: Images were collected in movie mode at 40 frames per second. Total of 25 frames were saved for each movie. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 130841 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)