+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3701 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Rubisco from Rhodobacter sphaeroides in complex with CABP and RcaCC | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Rhodobacter sphaeroides (バクテリア) Rhodobacter sphaeroides (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.56 Å | |||||||||
 データ登録者 データ登録者 | Bracher A / Milicic G / Ciniawsky S / Wendler P / Hayer-Hartl M / Hartl FU | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2017 ジャーナル: Mol Cell / 年: 2017タイトル: Mechanism of Enzyme Repair by the AAA Chaperone Rubisco Activase. 著者: Javaid Y Bhat / Goran Miličić / Gabriel Thieulin-Pardo / Andreas Bracher / Andrew Maxwell / Susanne Ciniawsky / Oliver Mueller-Cajar / John R Engen / F Ulrich Hartl / Petra Wendler / Manajit Hayer-Hartl /   要旨: How AAA+ chaperones conformationally remodel specific target proteins in an ATP-dependent manner is not well understood. Here, we investigated the mechanism of the AAA+ protein Rubisco activase (Rca) ...How AAA+ chaperones conformationally remodel specific target proteins in an ATP-dependent manner is not well understood. Here, we investigated the mechanism of the AAA+ protein Rubisco activase (Rca) in metabolic repair of the photosynthetic enzyme Rubisco, a complex of eight large (RbcL) and eight small (RbcS) subunits containing eight catalytic sites. Rubisco is prone to inhibition by tight-binding sugar phosphates, whose removal is catalyzed by Rca. We engineered a stable Rca hexamer ring and analyzed its functional interaction with Rubisco. Hydrogen/deuterium exchange and chemical crosslinking showed that Rca structurally destabilizes elements of the Rubisco active site with remarkable selectivity. Cryo-electron microscopy revealed that Rca docks onto Rubisco over one active site at a time, positioning the C-terminal strand of RbcL, which stabilizes the catalytic center, for access to the Rca hexamer pore. The pulling force of Rca is fine-tuned to avoid global destabilization and allow for precise enzyme repair. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3701.map.gz emd_3701.map.gz | 20.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3701-v30.xml emd-3701-v30.xml emd-3701.xml emd-3701.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3701.png emd_3701.png | 70.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3701 http://ftp.pdbj.org/pub/emdb/structures/EMD-3701 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3701 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3701 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3701_validation.pdf.gz emd_3701_validation.pdf.gz | 211.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3701_full_validation.pdf.gz emd_3701_full_validation.pdf.gz | 211 KB | 表示 | |
| XML形式データ |  emd_3701_validation.xml.gz emd_3701_validation.xml.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3701 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3701 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3701 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3701 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3701.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3701.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
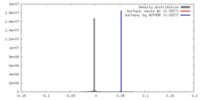
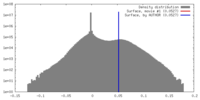
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rubisco-RcaCC complex
| 全体 | 名称: Rubisco-RcaCC complex |
|---|---|
| 要素 |
|
-超分子 #1: Rubisco-RcaCC complex
| 超分子 | 名称: Rubisco-RcaCC complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Rubisco was treated with the inhibitor CABP. RcaCC, ATP, ATP-gammaS and RuBP were added. The sample was treated with glutaraldehyde crosslinker. Rubisco-RcaCC complexes were purified by SEC. |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter sphaeroides (バクテリア) Rhodobacter sphaeroides (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 768 KDa |
-分子 #1: CbbX
| 分子 | 名称: CbbX / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter sphaeroides (バクテリア) Rhodobacter sphaeroides (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDAATAPTS IDLRAEYEGS GAKEVLEELD RELIGLKPVK DRIRETAAcL LVERARQKLG LAHETPTLH MSFTGNPGTG KTTVALKMAG LLHRLGYVRK GHLVSVTRDD LVGQYIGHTA P KTKEVLKR AMGGVLFIDE AYYLYRPDNE RDYGQEAIEI LLQVMENNRD ...文字列: MTDAATAPTS IDLRAEYEGS GAKEVLEELD RELIGLKPVK DRIRETAAcL LVERARQKLG LAHETPTLH MSFTGNPGTG KTTVALKMAG LLHRLGYVRK GHLVSVTRDD LVGQYIGHTA P KTKEVLKR AMGGVLFIDE AYYLYRPDNE RDYGQEAIEI LLQVMENNRD DLVVILAGYA DR MENFFQS NPGFRSRIAH HIEFPDYSDE ELFEIAGHML DDQNYQMTPE AETALRAYIG LRR NQPHFA NARSIRNALD RARLRQcNRL FTASSGPLDA RALSTMAEED IRASRVFKGG LDSE RRAAE ALAR |
-分子 #2: RbcL
| 分子 | 名称: RbcL / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter sphaeroides (バクテリア) Rhodobacter sphaeroides (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDTKTTEIKG KERYKAGVLK YAQMGYWDGD YVPKDTDVLA LFRITPQEGV DPVEAAAAVA GESSTATWT VVWTDRLTAC DSYRAKAYRV EPVPGTPGQY FCYVAYDLIL FEEGSIANLT A SIIGNVFS FKPLKAARLE DMRFPVAYVK TYKGPPTGIV GERERLDKFG ...文字列: MDTKTTEIKG KERYKAGVLK YAQMGYWDGD YVPKDTDVLA LFRITPQEGV DPVEAAAAVA GESSTATWT VVWTDRLTAC DSYRAKAYRV EPVPGTPGQY FCYVAYDLIL FEEGSIANLT A SIIGNVFS FKPLKAARLE DMRFPVAYVK TYKGPPTGIV GERERLDKFG KPLLGATTKP KL GLSGKNY GRVVYEGLKG GLDFMKDDEN INSQPFMHWR DRFLYVMEAV NLASAQTGEV KGH YLNITA GTMEEMYRRA EFAKSLGSVI VMVDLIIGYT AIQSISEWCR QNDMILHMHR AGHG TYTRQ KNHGISFRVI AKWLRLAGVD HLHCGTAVGK LEGDPLTVQG YYNVCREPFN TVDLP RGIF FEQDWADLRK VMPVASGGIH AGQMHQLLSL FGDDVVLQFG GGTIGHPMGI QAGATA NRV ALEAMVLARN EGRNIDVEGP EILRAAAKWC KPLEAALDTW GNITFNYTST DTSDFVP TA SVAM |
-分子 #3: RbcS
| 分子 | 名称: RbcS / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter sphaeroides (バクテリア) Rhodobacter sphaeroides (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRITQGCFSF LPDLTDEQIS AQVDYCLGRG WAVSLEHTDD PHPRNTYWEM WGMPMFDLRD PKGVMIELDE CRKAWPGRYI RINAFDSTRG FETVTMSFIV NRPEVEPSLR MERTEVDGRS IRYTHSIVR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - Film thickness: 2.0 nm / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | RcaCC hexamers (20 micromolar monomer) were mixed with E.C.M-CABP octamers (10 micromolar monomer) in a reaction containing 20 mM HEPES pH 7.5, 50 mM NaCl, 10 mM MgCl2, 10 mM ATP and 1mM RuBP, for 1 min at 25oC prior to addition of 0.125 % of glutaraldehyde (GA). After 10 min the reaction was quenched by addition of 0.1M Tris HCl pH 8 followed by gel filtration on a Superdex 200 PC 3.2/30 column (GE Healthcare).The fractions were eluted in buffer A and analyzed on a 6 % native gel. Fraction 13 containing HMW complexes with the least amount of free Rubisco were chosen for cryo-EM. The crosslinked E.C.M.-CABP-RcaCC complexes were diluted to 0.0030-0.0035 g ml-1 in 20 mM Tris-HCl pH 8.0, 50 mM NaCl, 1 mM ATP, 1 mM ATP-gammaS and 1 mM RuBP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Cs corrected Krios 1 at NeCEN (June 2016) |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均露光時間: 1.25 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 7.56 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 333711 |
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: OTHER / ソフトウェア - 名称: RELION |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)