+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of arginine oxidase from Pseudomonas sp. TRU 7192 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | OXIDOREDUCTASE | |||||||||
| 生物種 |  Pseudomonas sp. (バクテリア) Pseudomonas sp. (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.34 Å | |||||||||
 データ登録者 データ登録者 | Yamaguchi H / Numoto N / Suzuki H / Nishikawa K / Kamegawa A / Takahashi K / Sugiki M / Fujiyoshi Y | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural basis of arginine oxidase from Pseudomonas sp. TRU 7192 著者: Yamaguchi H / Numoto N / Suzuki H / Nishikawa K / Kamegawa A / Takahashi K / Matsui D / Asano Y / Sugiki M / Fujiyoshi Y | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36635.map.gz emd_36635.map.gz | 33.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36635-v30.xml emd-36635-v30.xml emd-36635.xml emd-36635.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36635_fsc.xml emd_36635_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36635.png emd_36635.png | 117.7 KB | ||
| マスクデータ |  emd_36635_msk_1.map emd_36635_msk_1.map | 36.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-36635.cif.gz emd-36635.cif.gz | 6.5 KB | ||
| その他 |  emd_36635_half_map_1.map.gz emd_36635_half_map_1.map.gz emd_36635_half_map_2.map.gz emd_36635_half_map_2.map.gz | 28.9 MB 28.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36635 http://ftp.pdbj.org/pub/emdb/structures/EMD-36635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36635 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36635.map.gz / 形式: CCP4 / 大きさ: 36.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36635.map.gz / 形式: CCP4 / 大きさ: 36.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.96 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_36635_msk_1.map emd_36635_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
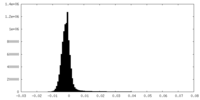
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36635_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
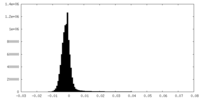
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36635_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
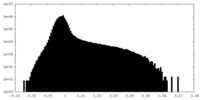
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : homooctamer of arginine oxidase
| 全体 | 名称: homooctamer of arginine oxidase |
|---|---|
| 要素 |
|
-超分子 #1: homooctamer of arginine oxidase
| 超分子 | 名称: homooctamer of arginine oxidase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas sp. (バクテリア) / 株: TPU 7192 Pseudomonas sp. (バクテリア) / 株: TPU 7192 |
| 分子量 | 理論値: 540 KDa |
-分子 #1: Amine oxidoreductase
| 分子 | 名称: Amine oxidoreductase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas sp. (バクテリア) / 株: TPU 7192 Pseudomonas sp. (バクテリア) / 株: TPU 7192 |
| 分子量 | 理論値: 67.393164 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHMSQ TQPLDVAIIG GGVSGTYSAW RLQEAQGDHQ RIQLFEYSDR IGGRLFSINL PGLPNVVAEV GGMRWMPATK DNTGGHVMV DKLVGELKLE SKNFPMGSNL PDKDPVGAKD NLFYLRGERF RLRDFTEAPD KIPYKLAWSE RGYGPEDLQV K VMHNIYPG ...文字列: MHHHHHHMSQ TQPLDVAIIG GGVSGTYSAW RLQEAQGDHQ RIQLFEYSDR IGGRLFSINL PGLPNVVAEV GGMRWMPATK DNTGGHVMV DKLVGELKLE SKNFPMGSNL PDKDPVGAKD NLFYLRGERF RLRDFTEAPD KIPYKLAWSE RGYGPEDLQV K VMHNIYPG FDKLSLAEQM QVKVFGKEIW RYGFWDLLYR VLSNEGYQFM KDAGGYEANV ANASAVTQLP ATEYSDKTVF LA LKKGFQA LPLTLAKRFA EVPGGLIAGE QRIRMNRRLA SVQFSDDTEY PYRLHFQATR TVDGKTSDVP GAEEIIHARQ VIL ALPRRS LELIQSPLFD DPWLKENIDS VLVQSAFKLF LAYEQPWWRS QGLVAGRSVT DLPIRQCYYM GTECEQDGGE KTLN SLLMA SYNDIGTVPF WKGLEDGAPF EGYQPKSLQG RIDANEVVPK MQYQISEEMV RIAQRQVTSL HDQIELPAPY SAVYH AWDA DPFGGGWHEW KANYRLDLII QRMRHPVQEQ EVYIVGEAYS YGQGWVEGAL TTAESTLQDF FGLPRPAWLP EAYQLL PAP APVDIDNPPA LACTDCKKTL TEVTEFAYTG IKA |
-分子 #2: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 29 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: MOLYBDENUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / 装置: LEICA KF80 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 5035 / 平均露光時間: 8.0 sec. / 平均電子線量: 69.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 3.4 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8jt7: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X