+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Fiber I and fiber-tail-adaptor of phage GP4 | |||||||||||||||
 マップデータ マップデータ | portal-vertex of phage GP4 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Complex / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 | Virion-associated phage protein / Virion-associated phage protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Ralstonia phage GP4 (ファージ) Ralstonia phage GP4 (ファージ) | |||||||||||||||
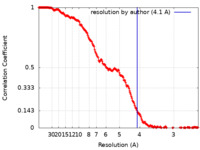
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Liu H / Chen W | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2023 ジャーナル: J Mol Biol / 年: 2023タイトル: Asymmetric Structure of Podophage GP4 Reveals a Novel Architecture of Three Types of Tail Fibers. 著者: Jing Zheng / Wenyuan Chen / Hao Xiao / Fan Yang / Jingdong Song / Lingpeng Cheng / Hongrong Liu /  要旨: Bacteriophage tail fibers (or called tail spikes) play a critical role in the early stage of infection by binding to the bacterial surface. Podophages with known structures usually possess one or two ...Bacteriophage tail fibers (or called tail spikes) play a critical role in the early stage of infection by binding to the bacterial surface. Podophages with known structures usually possess one or two types of fibers. Here, we resolved an asymmetric structure of the podophage GP4 to near-atomic resolution by cryo-EM. Our structure revealed a symmetry-mismatch relationship between the components of the GP4 tail with previously unseen topologies. In detail, two dodecameric adaptors (adaptors I and II), a hexameric nozzle, and a tail needle form a conserved tail body connected to a dodecameric portal occupying a unique vertex of the icosahedral head. However, five chain-like extended fibers (fiber I) and five tulip-like short fibers (fiber II) are anchored to a 15-fold symmetric fiber-tail adaptor, encircling the adaptor I, and six bamboo-like trimeric fibers (fiber III) are connected to the nozzle. Five fibers I, each composed of five dimers of the protein gp80 linked by an elongated rope protein, are attached to the five edges of the tail vertex of the icosahedral head. In this study, we identified a new structure of the podophage with three types of tail fibers, and such phages with different types of fibers may have a broad host range and/or infect host cells with considerably high efficiency, providing evolutionary advantages in harsh environments. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36462.map.gz emd_36462.map.gz | 580.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36462-v30.xml emd-36462-v30.xml emd-36462.xml emd-36462.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36462_fsc.xml emd_36462_fsc.xml | 21.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36462.png emd_36462.png | 174.6 KB | ||
| Filedesc metadata |  emd-36462.cif.gz emd-36462.cif.gz | 5.1 KB | ||
| その他 |  emd_36462_half_map_1.map.gz emd_36462_half_map_1.map.gz emd_36462_half_map_2.map.gz emd_36462_half_map_2.map.gz | 582.1 MB 582.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36462 http://ftp.pdbj.org/pub/emdb/structures/EMD-36462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36462 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36462_validation.pdf.gz emd_36462_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36462_full_validation.pdf.gz emd_36462_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_36462_validation.xml.gz emd_36462_validation.xml.gz | 27.6 KB | 表示 | |
| CIF形式データ |  emd_36462_validation.cif.gz emd_36462_validation.cif.gz | 37.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36462 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36462 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36462.map.gz / 形式: CCP4 / 大きさ: 641.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36462.map.gz / 形式: CCP4 / 大きさ: 641.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | portal-vertex of phage GP4 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.27 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map2
| ファイル | emd_36462_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map1
| ファイル | emd_36462_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ralstonia phage GP4
| 全体 | 名称:  Ralstonia phage GP4 (ファージ) Ralstonia phage GP4 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Ralstonia phage GP4
| 超分子 | 名称: Ralstonia phage GP4 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2282904 / 生物種: Ralstonia phage GP4 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Virion-associated phage protein
| 分子 | 名称: Virion-associated phage protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Ralstonia phage GP4 (ファージ) Ralstonia phage GP4 (ファージ) |
| 分子量 | 理論値: 14.360909 KDa |
| 配列 | 文字列: MAAASTYTEN NILNALLRGV AFPLPAKTYV SLHTGDPGVG AGANEVSLSN WPAYVRREAE QGGAIGSGWT PAASGQTSNV NQLTYPANN GVAAVTVTHY AVFDAPTGGN LLFKAPLTVA RTLQVGDVFV FDVGSLTAQA S UniProtKB: Virion-associated phage protein |
-分子 #2: rope protein of phage GP4
| 分子 | 名称: rope protein of phage GP4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Ralstonia phage GP4 (ファージ) Ralstonia phage GP4 (ファージ) |
| 分子量 | 理論値: 10.230603 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #3: Virion-associated phage protein
| 分子 | 名称: Virion-associated phage protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Ralstonia phage GP4 (ファージ) Ralstonia phage GP4 (ファージ) |
| 分子量 | 理論値: 9.908155 KDa |
| 配列 | 文字列: MLGIVQKTAT EQLDYDIDFA RWMPDGDVLQ SAGVVITPDD GTLTSPAYEI DGTVVKVWLA GGTAGASYNV DVTVATAAGR IKETCFKTR VRSC UniProtKB: Virion-associated phage protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.8000000000000003 µm 最小 デフォーカス(公称値): 0.1 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X