+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SV2A in complex with BoNT/A2 Hc | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Synaptic vesicle / epilepsy / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of gamma-aminobutyric acid secretion / Toxicity of botulinum toxin type F (botF) / Toxicity of botulinum toxin type D (botD) / Toxicity of botulinum toxin type E (botE) / Toxicity of botulinum toxin type A (botA) / synaptic vesicle priming / presynaptic active zone / transmembrane transporter activity / protein transmembrane transporter activity / GABA-ergic synapse ...regulation of gamma-aminobutyric acid secretion / Toxicity of botulinum toxin type F (botF) / Toxicity of botulinum toxin type D (botD) / Toxicity of botulinum toxin type E (botE) / Toxicity of botulinum toxin type A (botA) / synaptic vesicle priming / presynaptic active zone / transmembrane transporter activity / protein transmembrane transporter activity / GABA-ergic synapse / neuromuscular junction / metalloendopeptidase activity / synaptic vesicle membrane / intracellular calcium ion homeostasis / cell-cell junction / synaptic vesicle / toxin activity / neuron projection / neuronal cell body / glutamatergic synapse / dendrite / protein kinase binding / endoplasmic reticulum / proteolysis / zinc ion binding / extracellular region / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
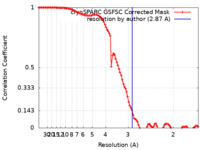
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.87 Å | |||||||||
 データ登録者 データ登録者 | Yamagata A | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for antiepileptic drugs and botulinum neurotoxin recognition of SV2A. 著者: Atsushi Yamagata / Kaori Ito / Takehiro Suzuki / Naoshi Dohmae / Tohru Terada / Mikako Shirouzu /  要旨: More than one percent of people have epilepsy worldwide. Levetiracetam (LEV) is a successful new-generation antiepileptic drug (AED), and its derivative, brivaracetam (BRV), shows improved efficacy. ...More than one percent of people have epilepsy worldwide. Levetiracetam (LEV) is a successful new-generation antiepileptic drug (AED), and its derivative, brivaracetam (BRV), shows improved efficacy. Synaptic vesicle glycoprotein 2a (SV2A), a putative membrane transporter in the synaptic vesicles (SVs), has been identified as a target of LEV and BRV. SV2A also serves as a receptor for botulinum neurotoxin (BoNT), which is the most toxic protein and has paradoxically emerged as a potent reagent for therapeutic and cosmetic applications. Nevertheless, no structural analysis on AEDs and BoNT recognition by full-length SV2A has been available. Here we describe the cryo-electron microscopy structures of the full-length SV2A in complex with the BoNT receptor-binding domain, BoNT/A2 H and either LEV or BRV. The large fourth luminal domain of SV2A binds to BoNT/A2 H through protein-protein and protein-glycan interactions. LEV and BRV occupy the putative substrate-binding site in an outward-open conformation. A propyl group in BRV creates additional contacts with SV2A, explaining its higher binding affinity than that of LEV, which was further supported by label-free spectral shift assay. Numerous LEV derivatives have been developed as AEDs and positron emission tomography (PET) tracers for neuroimaging. Our work provides a structural framework for AEDs and BoNT recognition of SV2A and a blueprint for the rational design of additional AEDs and PET tracers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36396.map.gz emd_36396.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36396-v30.xml emd-36396-v30.xml emd-36396.xml emd-36396.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36396_fsc.xml emd_36396_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36396.png emd_36396.png | 33.8 KB | ||
| マスクデータ |  emd_36396_msk_1.map emd_36396_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-36396.cif.gz emd-36396.cif.gz | 6 KB | ||
| その他 |  emd_36396_half_map_1.map.gz emd_36396_half_map_1.map.gz emd_36396_half_map_2.map.gz emd_36396_half_map_2.map.gz | 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36396 http://ftp.pdbj.org/pub/emdb/structures/EMD-36396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36396 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36396_validation.pdf.gz emd_36396_validation.pdf.gz | 971 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36396_full_validation.pdf.gz emd_36396_full_validation.pdf.gz | 970.6 KB | 表示 | |
| XML形式データ |  emd_36396_validation.xml.gz emd_36396_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_36396_validation.cif.gz emd_36396_validation.cif.gz | 22.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36396 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36396 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36396 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36396 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jlgMC  8jlcC  8jleC  8jlfC  8jlhC  8jliC  8js8C  8js9C  8k77C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36396.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36396.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
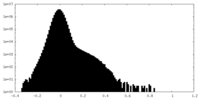
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_36396_msk_1.map emd_36396_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
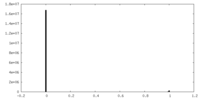
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36396_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
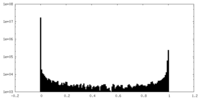
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36396_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
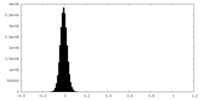
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SV2A LD4 in complex with BoNT/A2 Hc domain from the SV2A-BoNT/A2 ...
| 全体 | 名称: SV2A LD4 in complex with BoNT/A2 Hc domain from the SV2A-BoNT/A2 Hc complex |
|---|---|
| 要素 |
|
-超分子 #1: SV2A LD4 in complex with BoNT/A2 Hc domain from the SV2A-BoNT/A2 ...
| 超分子 | 名称: SV2A LD4 in complex with BoNT/A2 Hc domain from the SV2A-BoNT/A2 Hc complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Synaptic vesicle glycoprotein 2A
| 分子 | 名称: Synaptic vesicle glycoprotein 2A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.056498 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: YASRTKVFPG ERVEHVTFNF TLENQIHRGG QYFNDKFIGL RLKSVSFEDS LFEECYFEDV TSSNTFFRNC TFINTVFYNT DLFEYKFVN SRLINSTFLH NKEGCPLDVT GT UniProtKB: Synaptic vesicle glycoprotein 2A |
-分子 #2: Botulinum neurotoxin
| 分子 | 名称: Botulinum neurotoxin / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.456191 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KNIVNTSILS IVYKKDDLID LSRYGAKINI GDRVYYDSID KNQIKLINLE SSTIEVILKN AIVYNSMYEN FSTSFWIKIP KYFSKINLN NEYTIINCIE NNSGWKVSLN YGEIIWTLQD NKQNIQRVVF KYSQMVNISD YINRWIFVTI TNNRLTKSKI Y INGRLIDQ ...文字列: KNIVNTSILS IVYKKDDLID LSRYGAKINI GDRVYYDSID KNQIKLINLE SSTIEVILKN AIVYNSMYEN FSTSFWIKIP KYFSKINLN NEYTIINCIE NNSGWKVSLN YGEIIWTLQD NKQNIQRVVF KYSQMVNISD YINRWIFVTI TNNRLTKSKI Y INGRLIDQ KPISNLGNIH ASNKIMFKLD GCRDPRRYIM IKYFNLFDKE LNEKEIKDLY DSQSNSGILK DFWGNYLQYD KP YYMLNLF DPNKYVDVNN IGIRGYMYLK GPRGSVVTTN IYLNSTLYEG TKFIIKKYAS GNEDNIVRNN DRVYINVVVK NKE YRLATN ASQAGVEKIL SALEIPDVGN LSQVVVMKSK DDQGIRNKCK MNLQDNNGND IGFIGFHLYD NIAKLVASNW YNRQ VGKAS RTFGCSWEFI PVDDGWGESS L UniProtKB: Botulinum neurotoxin |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: 20 mM Hepes (pH7.5), 150 mM NaCl, 0.001% LMNG, 0.0002% cholesterol hemisuccinate |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 297 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4890 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X