+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3627 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RNA polymerase II-Paf1C-TFIIS-A | |||||||||||||||
 マップデータ マップデータ | Non-B factor sharpened, locally filtered map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |  | |||||||||||||||
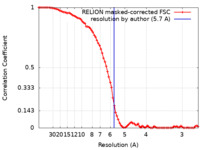
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Xu Y / Cramer P | |||||||||||||||
| 資金援助 |  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Architecture of the RNA polymerase II-Paf1C-TFIIS transcription elongation complex. 著者: Youwei Xu / Carrie Bernecky / Chung-Tien Lee / Kerstin C Maier / Björn Schwalb / Dimitry Tegunov / Jürgen M Plitzko / Henning Urlaub / Patrick Cramer /  要旨: The conserved polymerase-associated factor 1 complex (Paf1C) plays multiple roles in chromatin transcription and genomic regulation. Paf1C comprises the five subunits Paf1, Leo1, Ctr9, Cdc73 and ...The conserved polymerase-associated factor 1 complex (Paf1C) plays multiple roles in chromatin transcription and genomic regulation. Paf1C comprises the five subunits Paf1, Leo1, Ctr9, Cdc73 and Rtf1, and binds to the RNA polymerase II (Pol II) transcription elongation complex (EC). Here we report the reconstitution of Paf1C from Saccharomyces cerevisiae, and a structural analysis of Paf1C bound to a Pol II EC containing the elongation factor TFIIS. Cryo-electron microscopy and crosslinking data reveal that Paf1C is highly mobile and extends over the outer Pol II surface from the Rpb2 to the Rpb3 subunit. The Paf1-Leo1 heterodimer and Cdc73 form opposite ends of Paf1C, whereas Ctr9 bridges between them. Consistent with the structural observations, the initiation factor TFIIF impairs Paf1C binding to Pol II, whereas the elongation factor TFIIS enhances it. We further show that Paf1C is globally required for normal mRNA transcription in yeast. These results provide a three-dimensional framework for further analysis of Paf1C function in transcription through chromatin. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3627.map.gz emd_3627.map.gz | 48 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3627-v30.xml emd-3627-v30.xml emd-3627.xml emd-3627.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3627_fsc.xml emd_3627_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3627.png emd_3627.png | 106.4 KB | ||
| マスクデータ |  emd_3627_msk_1.map emd_3627_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_3627_half_map_1.map.gz emd_3627_half_map_1.map.gz emd_3627_half_map_2.map.gz emd_3627_half_map_2.map.gz | 40.7 MB 40.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3627 http://ftp.pdbj.org/pub/emdb/structures/EMD-3627 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3627 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3627 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3627_validation.pdf.gz emd_3627_validation.pdf.gz | 383 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3627_full_validation.pdf.gz emd_3627_full_validation.pdf.gz | 382.1 KB | 表示 | |
| XML形式データ |  emd_3627_validation.xml.gz emd_3627_validation.xml.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3627 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3627 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3627 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3627 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3627.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3627.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-B factor sharpened, locally filtered map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_3627_msk_1.map emd_3627_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
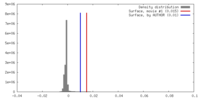
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_3627_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
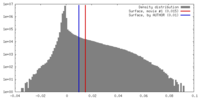
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_3627_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA polymerase II-Paf1C-TFIIS-A
| 全体 | 名称: RNA polymerase II-Paf1C-TFIIS-A |
|---|---|
| 要素 |
|
-超分子 #1: RNA polymerase II-Paf1C-TFIIS-A
| 超分子 | 名称: RNA polymerase II-Paf1C-TFIIS-A / タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Elongation complex |
|---|---|
| 分子量 | 理論値: 900 KDa |
-超分子 #2: Paf1C
| 超分子 | 名称: Paf1C / タイプ: complex / ID: 2 / 親要素: 1 / 詳細: Ctr9 C-terminal truncation |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 340 KDa |
-超分子 #3: RNA polymerase II
| 超分子 | 名称: RNA polymerase II / タイプ: complex / ID: 3 / 親要素: 1 / 詳細: 12 subunits |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 500 KDa |
-超分子 #4: TFIIS
| 超分子 | 名称: TFIIS / タイプ: complex / ID: 4 / 親要素: 1 / 詳細: TFIIS (D290A, E291A) inactive mutant |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 35 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil Cu R3.5/1 and Cu R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 4 micro-L of sample was placed onto Quantifoil Cu R3.5/1 and Cu R2/1 glow-discharged 200 mesh holey carbon grids, which were then blotted for 8.5 s with blot force 13 to remove the excess ...詳細: 4 micro-L of sample was placed onto Quantifoil Cu R3.5/1 and Cu R2/1 glow-discharged 200 mesh holey carbon grids, which were then blotted for 8.5 s with blot force 13 to remove the excess solution before they were flash-frozen in liquid ethane.. | ||||||||||||||||||
| 詳細 | Gradient fixation |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: SUPER-RESOLUTION / #0 - 撮影したグリッド数: 1 / #0 - 実像数: 595 / #0 - 平均露光時間: 0.4 sec. / #0 - 平均電子線量: 1.22 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 検出モード: SUPER-RESOLUTION / #1 - 撮影したグリッド数: 1 / #1 - 実像数: 2146 / #1 - 平均露光時間: 0.4 sec. / #1 - 平均電子線量: 0.93 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 37000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)