+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Helicobacter pylori UreFD/urease complex | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Urease / activation complex / UreA / UreB / UreC / UreF / UreD / UreH / Helicobacter pylori / HYDROLASE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報urease complex / urease / urease activity / urea catabolic process / metabolic process / nickel cation binding / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) | |||||||||||||||
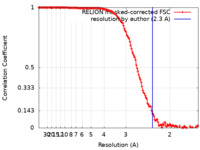
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.3 Å | |||||||||||||||
 データ登録者 データ登録者 | Nim YS / Fong IYH / Deme J / Tsang KL / Caesar J / Johnson S / Wong KB / Lea SM | |||||||||||||||
| 資金援助 |  香港, 香港,  米国, 米国,  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Delivering a toxic metal to the active site of urease. 著者: Yap Shing Nim / Ivan Yu Hang Fong / Justin Deme / Ka Lung Tsang / Joseph Caesar / Steven Johnson / Longson Tsz Hin Pang / Nicholas Man Hon Yuen / Tin Long Chris Ng / Tung Choi / Yakie Yat Hei ...著者: Yap Shing Nim / Ivan Yu Hang Fong / Justin Deme / Ka Lung Tsang / Joseph Caesar / Steven Johnson / Longson Tsz Hin Pang / Nicholas Man Hon Yuen / Tin Long Chris Ng / Tung Choi / Yakie Yat Hei Wong / Susan M Lea / Kam-Bo Wong /    要旨: Urease is a nickel (Ni) enzyme that is essential for the colonization of in the human stomach. To solve the problem of delivering the toxic Ni ion to the active site without diffusing into the ...Urease is a nickel (Ni) enzyme that is essential for the colonization of in the human stomach. To solve the problem of delivering the toxic Ni ion to the active site without diffusing into the cytoplasm, cells have evolved metal carrier proteins, or metallochaperones, to deliver the toxic ions to specific protein complexes. Ni delivery requires urease to form an activation complex with the urease accessory proteins UreFD and UreG. Here, we determined the cryo-electron microscopy structures of UreFD/urease and UreD/urease complexes at 2.3- and 2.7-angstrom resolutions, respectively. Combining structural, mutagenesis, and biochemical studies, we show that the formation of the activation complex opens a 100-angstrom-long tunnel, where the Ni ion is delivered through UreFD to the active site of urease. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34648.map.gz emd_34648.map.gz | 189.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34648-v30.xml emd-34648-v30.xml emd-34648.xml emd-34648.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34648_fsc.xml emd_34648_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34648.png emd_34648.png | 164.7 KB | ||
| Filedesc metadata |  emd-34648.cif.gz emd-34648.cif.gz | 6.6 KB | ||
| その他 |  emd_34648_half_map_1.map.gz emd_34648_half_map_1.map.gz emd_34648_half_map_2.map.gz emd_34648_half_map_2.map.gz | 190.6 MB 190.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34648 http://ftp.pdbj.org/pub/emdb/structures/EMD-34648 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34648 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34648 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hc1MC  8hcnC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34648.map.gz / 形式: CCP4 / 大きさ: 240.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34648.map.gz / 形式: CCP4 / 大きさ: 240.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_34648_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
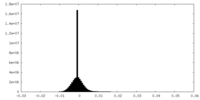
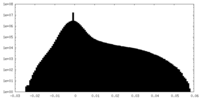
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34648_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
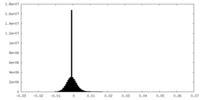
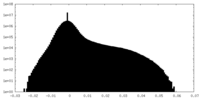
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helicobacter pylori UreFD/urease complex
| 全体 | 名称: Helicobacter pylori UreFD/urease complex |
|---|---|
| 要素 |
|
-超分子 #1: Helicobacter pylori UreFD/urease complex
| 超分子 | 名称: Helicobacter pylori UreFD/urease complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) |
| 分子量 | 理論値: 1.76 MDa |
-分子 #1: Urease subunit alpha
| 分子 | 名称: Urease subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 |
| 分子量 | 理論値: 26.587662 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKLTPKELDK LMLHYAGELA KKRKEKGIKL NYVEAVALIS AHIMEEARAG KKTAAELMQE GRTLLKPDDV MDGVASMIHE VGIEAMFPD GTKLVTVHTP IEANGKLVPG ELFLKNEDIT INEGKKAVSV KVKNVGDRPV QIGSHFHFFE VNRCLDFDRE K TFGKRLDI ...文字列: MKLTPKELDK LMLHYAGELA KKRKEKGIKL NYVEAVALIS AHIMEEARAG KKTAAELMQE GRTLLKPDDV MDGVASMIHE VGIEAMFPD GTKLVTVHTP IEANGKLVPG ELFLKNEDIT INEGKKAVSV KVKNVGDRPV QIGSHFHFFE VNRCLDFDRE K TFGKRLDI ASGTAVRFEP GEEKSVELID IGGNRRIFGF NALVDRQADN ESKKIALHRA KERGFHGAKS DDNYVKTIKE UniProtKB: Urease subunit alpha |
-分子 #2: Urease subunit beta
| 分子 | 名称: Urease subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 |
| 分子量 | 理論値: 61.759508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKKISRKEYV SMYGPTTGDK VRLGDTDLIA EVEHDYTIYG EELKFGGGKT LREGMSQSNN PSKEELDLII TNALIVDYTG IYKADIGIK DGKIAGIGKG GNKDMQDGVK NNLSVGPATE ALAGEGLIVT AGGIDTHIHF ISPQQIPTAF ASGVTTMIGG G TGPADGTN ...文字列: MKKISRKEYV SMYGPTTGDK VRLGDTDLIA EVEHDYTIYG EELKFGGGKT LREGMSQSNN PSKEELDLII TNALIVDYTG IYKADIGIK DGKIAGIGKG GNKDMQDGVK NNLSVGPATE ALAGEGLIVT AGGIDTHIHF ISPQQIPTAF ASGVTTMIGG G TGPADGTN ATTITPGRRN LKWMLRAAEE YSMNLGFLAK GNASNDASLA DQIEAGAIGF KIHEDWGTTP SAINHALDVA DK YDVQVAI HTDTLNEAGC VEDTMAAIAG RTMHTFHTEG AGGGHAPDII KVAGEHNILP ASTNPTIPFT VNTEAEHMDM LMV CHHLDK SIKEDVQFAD SRIRPQTIAA EDTLHDMGIF SITSSDSQAM GRVGEVITRT WQTADKNKKE FGRLKEEKGD NDNF RIKRY LSKYTINPAI AHGISEYVGS VEVGKVADLV LWSPAFFGVK PNMIIKGGFI ALSQMGDANA SIPTPQPVYY REMFA HHGK AKYDANITFV SQAAYDKGIK EELGLERQVL PVKNCRNITK KDMQFNDTTA HIEVNPETYH VFVDGKEVTS KPANKV SLA QLFSIF UniProtKB: Urease subunit beta |
-分子 #3: Urease accessory protein UreH
| 分子 | 名称: Urease accessory protein UreH / タイプ: protein_or_peptide / ID: 3 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 |
| 分子量 | 理論値: 30.741244 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNTYAQESKL RLKTKIGADG RCVIEDNFFT PPFKLMAPFY PKDDLAEIML LAVSPGMMRG DAQDVQLNIG PNCKLRITSQ SFEKIHNTE DGFASRDMHI VVGENAFLDF APFPLIPFEN AHFKGNTTIS LRSSSQLLYS AIIVAGRVAR NELFKFNRLH T KISILQDE ...文字列: MNTYAQESKL RLKTKIGADG RCVIEDNFFT PPFKLMAPFY PKDDLAEIML LAVSPGMMRG DAQDVQLNIG PNCKLRITSQ SFEKIHNTE DGFASRDMHI VVGENAFLDF APFPLIPFEN AHFKGNTTIS LRSSSQLLYS AIIVAGRVAR NELFKFNRLH T KISILQDE KPIYYDNTIL DPKTTDLNNM CMFDGYTHYL NLVLVNCPIE LSGVRECIEE SEGVDGAVSE TASSHLCVKA LA KGSEPLL HLREKIARLV TQTTTQKVWS HPQFEK UniProtKB: Urease accessory protein UreH |
-分子 #4: Urease accessory protein UreF
| 分子 | 名称: Urease accessory protein UreF / タイプ: protein_or_peptide / ID: 4 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 Helicobacter pylori 26695 (ピロリ菌) / 株: ATCC 700392 / 26695 |
| 分子量 | 理論値: 28.517635 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDKGKSVKST EKSVGMPPKT PKTDNNAHVD NEFLILQVND AVFPIGSYTH SFGLETYIQQ KKVTNKESAL EYLKANLSSQ FLYTEMLSL KLTYESALQQ DLKKILGVEE VIMLSTSPME LRLANQKLGN RFIKTLQAMN ELDMGEFFNA YAQKTKDPTH A TSYGVFAA ...文字列: MDKGKSVKST EKSVGMPPKT PKTDNNAHVD NEFLILQVND AVFPIGSYTH SFGLETYIQQ KKVTNKESAL EYLKANLSSQ FLYTEMLSL KLTYESALQQ DLKKILGVEE VIMLSTSPME LRLANQKLGN RFIKTLQAMN ELDMGEFFNA YAQKTKDPTH A TSYGVFAA SLGIELKKAL AHYLDAQTSN MVINCVKSVP LSQNDGQKIL LSLQSPFNQL IEKTLELDES HLCTASVQND IK AMQHESL YSRLYMS UniProtKB: Urease accessory protein UreF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -1.5 µm / 最小 デフォーカス(公称値): -0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X