+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-32377 | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 2.02 angstrom cryo-EM structure of the pump-like channelrhodopsin ChRmine | |||||||||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
| 生物種 |  Rhodomonas lens (真核生物) Rhodomonas lens (真核生物) | |||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.02 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Kishi KE / Kim Y / Fukuda M / Yamashita K / Deisseroth K / Kato HE | |||||||||||||||||||||||||||
| 資金援助 |  日本, 8件 日本, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: Structural basis for channel conduction in the pump-like channelrhodopsin ChRmine. 著者: Koichiro E Kishi / Yoon Seok Kim / Masahiro Fukuda / Masatoshi Inoue / Tsukasa Kusakizako / Peter Y Wang / Charu Ramakrishnan / Eamon F X Byrne / Elina Thadhani / Joseph M Paggi / Toshiki E ...著者: Koichiro E Kishi / Yoon Seok Kim / Masahiro Fukuda / Masatoshi Inoue / Tsukasa Kusakizako / Peter Y Wang / Charu Ramakrishnan / Eamon F X Byrne / Elina Thadhani / Joseph M Paggi / Toshiki E Matsui / Keitaro Yamashita / Takashi Nagata / Masae Konno / Sean Quirin / Maisie Lo / Tyler Benster / Tomoko Uemura / Kehong Liu / Mikihiro Shibata / Norimichi Nomura / So Iwata / Osamu Nureki / Ron O Dror / Keiichi Inoue / Karl Deisseroth / Hideaki E Kato /    要旨: ChRmine, a recently discovered pump-like cation-conducting channelrhodopsin, exhibits puzzling properties (large photocurrents, red-shifted spectrum, and extreme light sensitivity) that have created ...ChRmine, a recently discovered pump-like cation-conducting channelrhodopsin, exhibits puzzling properties (large photocurrents, red-shifted spectrum, and extreme light sensitivity) that have created new opportunities in optogenetics. ChRmine and its homologs function as ion channels but, by primary sequence, more closely resemble ion pump rhodopsins; mechanisms for passive channel conduction in this family have remained mysterious. Here, we present the 2.0 Å resolution cryo-EM structure of ChRmine, revealing architectural features atypical for channelrhodopsins: trimeric assembly, a short transmembrane-helix 3, a twisting extracellular-loop 1, large vestibules within the monomer, and an opening at the trimer interface. We applied this structure to design three proteins (rsChRmine and hsChRmine, conferring further red-shifted and high-speed properties, respectively, and frChRmine, combining faster and more red-shifted performance) suitable for fundamental neuroscience opportunities. These results illuminate the conduction and gating of pump-like channelrhodopsins and point the way toward further structure-guided creation of channelrhodopsins for applications across biology. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32377.map.gz emd_32377.map.gz | 2.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32377-v30.xml emd-32377-v30.xml emd-32377.xml emd-32377.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32377_fsc.xml emd_32377_fsc.xml | 15.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32377.png emd_32377.png | 165.2 KB | ||
| マスクデータ |  emd_32377_msk_1.map emd_32377_msk_1.map | 16.2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_32377_half_map_1.map.gz emd_32377_half_map_1.map.gz emd_32377_half_map_2.map.gz emd_32377_half_map_2.map.gz | 14.9 MB 14.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32377 http://ftp.pdbj.org/pub/emdb/structures/EMD-32377 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32377 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32377 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32377_validation.pdf.gz emd_32377_validation.pdf.gz | 653.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32377_full_validation.pdf.gz emd_32377_full_validation.pdf.gz | 653.2 KB | 表示 | |
| XML形式データ |  emd_32377_validation.xml.gz emd_32377_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_32377_validation.cif.gz emd_32377_validation.cif.gz | 22.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32377 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32377 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32377 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32377 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7w9wMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10926 (タイトル: The pump-like chanelrhodopsin ChRmine / Data size: 850.8 EMPIAR-10926 (タイトル: The pump-like chanelrhodopsin ChRmine / Data size: 850.8 Data #1: Multiframe micrographs for Structural basis for channel conduction in the pump-like channelrhodopsin ChRmine [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32377.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32377.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.94318 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
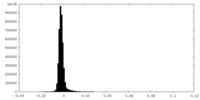
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
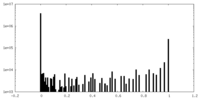
| ファイル |  emd_32377_msk_1.map emd_32377_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
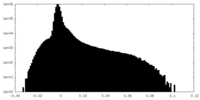
| ファイル | emd_32377_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
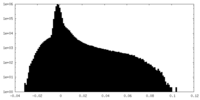
| ファイル | emd_32377_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ChRmine
| 全体 | 名称: ChRmine |
|---|---|
| 要素 |
|
-超分子 #1: ChRmine
| 超分子 | 名称: ChRmine / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Rhodomonas lens (真核生物) Rhodomonas lens (真核生物) |
| 組換発現 | 生物種:  |
-分子 #1: ChRmine
| 分子 | 名称: ChRmine / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodomonas lens (真核生物) Rhodomonas lens (真核生物) |
| 分子量 | 理論値: 35.772965 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPMAHAPGTD QMFYVGTMDG WYLDTKLNSV AIGAHWSCFI VLTITTFYLG YESWTSRGPS KRTSFYAGYQ EEQNLALFVN FFAMLSYFG KIVADTLGHN FGDVGPFIIG FGNYRYADYM LTCPMLVYDL LYQLRAPYRV SCSAIIFAIL MSGVLAEFYA E GDPRLRNG ...文字列: GPMAHAPGTD QMFYVGTMDG WYLDTKLNSV AIGAHWSCFI VLTITTFYLG YESWTSRGPS KRTSFYAGYQ EEQNLALFVN FFAMLSYFG KIVADTLGHN FGDVGPFIIG FGNYRYADYM LTCPMLVYDL LYQLRAPYRV SCSAIIFAIL MSGVLAEFYA E GDPRLRNG AYAWYGFGCF WFIFAYSIVM SIVAKQYSRL AQLAQDTGAE HSLHVLKFAV FTFSMLWILF PLVWAICPRG FG WIDDNWT EVAHCVCDIV AKSCYGFALA RFRKTYDEEL FRLLEQLGHD EDEFQKLELD MRLSSNGERL EVLFQ |
-分子 #2: RETINAL
| 分子 | 名称: RETINAL / タイプ: ligand / ID: 2 / コピー数: 1 / 式: RET |
|---|---|
| 分子量 | 理論値: 284.436 Da |
| Chemical component information |  ChemComp-RET: |
-分子 #3: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 3 / コピー数: 2 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #4: PALMITIC ACID
| 分子 | 名称: PALMITIC ACID / タイプ: ligand / ID: 4 / コピー数: 5 / 式: PLM |
|---|---|
| 分子量 | 理論値: 256.424 Da |
| Chemical component information |  ChemComp-PLM: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 48 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 51.533 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X