+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-32068 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human ATP13A2 (SPM-bound E2P state) | ||||||||||||
 マップデータ マップデータ | postprocess_masked.mrc from Relion3.1 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | ATP13A2 / PARK9 / P-type ATPase / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報polyamine transmembrane transporter activity / polyamine transmembrane transport / spermine transmembrane transport / peptidyl-aspartic acid autophosphorylation / regulation of ubiquitin-specific protease activity / ABC-type polyamine transporter activity / regulation of autophagosome size / extracellular exosome biogenesis / P-type ion transporter activity / regulation of chaperone-mediated autophagy ...polyamine transmembrane transporter activity / polyamine transmembrane transport / spermine transmembrane transport / peptidyl-aspartic acid autophosphorylation / regulation of ubiquitin-specific protease activity / ABC-type polyamine transporter activity / regulation of autophagosome size / extracellular exosome biogenesis / P-type ion transporter activity / regulation of chaperone-mediated autophagy / negative regulation of lysosomal protein catabolic process / regulation of autophagy of mitochondrion / regulation of lysosomal protein catabolic process / intracellular monoatomic cation homeostasis / autophagosome-lysosome fusion / autophagosome organization / protein localization to lysosome / phosphatidic acid binding / positive regulation of exosomal secretion / multivesicular body membrane / ATPase-coupled monoatomic cation transmembrane transporter activity / intracellular zinc ion homeostasis / regulation of protein localization to nucleus / トランスロカーゼ; 他の化合物の輸送を触媒; ヌクレオシド三リン酸の加水分解に伴う / cupric ion binding / regulation of mitochondrion organization / regulation of endopeptidase activity / lysosomal transport / phosphatidylinositol-3,5-bisphosphate binding / cellular response to zinc ion / regulation of intracellular protein transport / lipid homeostasis / Ion transport by P-type ATPases / autophagosome membrane / cellular response to manganese ion / regulation of macroautophagy / regulation of neuron apoptotic process / transport vesicle / autophagosome / multivesicular body / lysosomal lumen / positive regulation of protein secretion / transmembrane transport / autophagy / intracellular calcium ion homeostasis / late endosome / late endosome membrane / manganese ion binding / cellular response to oxidative stress / monoatomic ion transmembrane transport / intracellular iron ion homeostasis / vesicle / protein autophosphorylation / lysosome / neuron projection / lysosomal membrane / neuronal cell body / positive regulation of gene expression / ATP hydrolysis activity / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
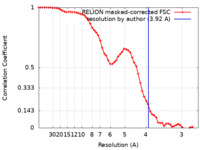
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.92 Å | ||||||||||||
 データ登録者 データ登録者 | Tomita A / Yamashita K / Nishizawa T / Nureki O | ||||||||||||
| 資金援助 |  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Cryo-EM reveals mechanistic insights into lipid-facilitated polyamine export by human ATP13A2. 著者: Atsuhiro Tomita / Takashi Daiho / Tsukasa Kusakizako / Keitaro Yamashita / Satoshi Ogasawara / Takeshi Murata / Tomohiro Nishizawa / Osamu Nureki /   要旨: The cytoplasmic polyamine maintains cellular homeostasis by chelating toxic metal cations, regulating transcriptional activity, and protecting DNA. ATP13A2 was identified as a lysosomal polyamine ...The cytoplasmic polyamine maintains cellular homeostasis by chelating toxic metal cations, regulating transcriptional activity, and protecting DNA. ATP13A2 was identified as a lysosomal polyamine exporter responsible for polyamine release into the cytosol, and its dysfunction is associated with Alzheimer's disease and other neural degradation diseases. ATP13A2 belongs to the P5 subfamily of the P-type ATPase family, but its mechanisms remain unknown. Here, we report the cryoelectron microscopy (cryo-EM) structures of human ATP13A2 under four different conditions, revealing the structural coupling between the polyamine binding and the dephosphorylation. Polyamine is bound at the luminal tunnel and recognized through numerous electrostatic and π-cation interactions, explaining its broad specificity. The unique N-terminal domain is anchored to the lipid membrane to stabilize the E2P conformation, thereby accelerating the E1P-to-E2P transition. These findings reveal the distinct mechanism of P5B ATPases, thereby paving the way for neuroprotective therapy by activating ATP13A2. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32068.map.gz emd_32068.map.gz | 2.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32068-v30.xml emd-32068-v30.xml emd-32068.xml emd-32068.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32068_fsc.xml emd_32068_fsc.xml | 5.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32068.png emd_32068.png | 129.1 KB | ||
| マスクデータ |  emd_32068_msk_1.map emd_32068_msk_1.map | 14.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-32068.cif.gz emd-32068.cif.gz | 6.6 KB | ||
| その他 |  emd_32068_half_map_1.map.gz emd_32068_half_map_1.map.gz emd_32068_half_map_2.map.gz emd_32068_half_map_2.map.gz | 11.3 MB 11.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32068 http://ftp.pdbj.org/pub/emdb/structures/EMD-32068 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32068 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32068 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32068_validation.pdf.gz emd_32068_validation.pdf.gz | 801.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32068_full_validation.pdf.gz emd_32068_full_validation.pdf.gz | 801.2 KB | 表示 | |
| XML形式データ |  emd_32068_validation.xml.gz emd_32068_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_32068_validation.cif.gz emd_32068_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32068 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32068 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32068 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32068 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7vpkMC  7vpiC  7vpjC  7vplC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10975 (タイトル: Cryo-EM structure of the human ATP13A2 / Data size: 3.8 TB EMPIAR-10975 (タイトル: Cryo-EM structure of the human ATP13A2 / Data size: 3.8 TBData #1: Unaligned movies for E1-ATP state [micrographs - multiframe] Data #2: Unaligned movies for E1P-ADP state [micrographs - multiframe] Data #3: Unaligned movies for SPM-bound E2P state [micrographs - multiframe] Data #4: Unaligned movies for SPM-bound E2Pi state [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32068.map.gz / 形式: CCP4 / 大きさ: 14.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32068.map.gz / 形式: CCP4 / 大きさ: 14.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocess_masked.mrc from Relion3.1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34875 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_32068_msk_1.map emd_32068_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: run half1 class001.mrc from Relion3.1
| ファイル | emd_32068_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | run_half1_class001.mrc from Relion3.1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: run half1 class002.mrc from Relion3.1
| ファイル | emd_32068_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | run_half1_class002.mrc from Relion3.1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ATP13A2
| 全体 | 名称: ATP13A2 |
|---|---|
| 要素 |
|
-超分子 #1: ATP13A2
| 超分子 | 名称: ATP13A2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: Human ATP13A2 in complex with SPM and BeF |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Polyamine-transporting ATPase 13A2
| 分子 | 名称: Polyamine-transporting ATPase 13A2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; 他の化合物の輸送を触媒; ヌクレオシド三リン酸の加水分解に伴う |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 129.313391 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPSRMSADSS PLVGSTPTGY GTLTIGTSID PLSSSVSSVR LSGYCGSPWR VIGYHVVVWM MAGIPLLLFR WKPLWGVRLR LRPCNLAHA ETLVIEIRDK EDSSWQLFTV QVQTEAIGEG SLEPSPQSQA EDGRSQAAVG AVPEGAWKDT AQLHKSEEAV S VGQKRVLR ...文字列: GPSRMSADSS PLVGSTPTGY GTLTIGTSID PLSSSVSSVR LSGYCGSPWR VIGYHVVVWM MAGIPLLLFR WKPLWGVRLR LRPCNLAHA ETLVIEIRDK EDSSWQLFTV QVQTEAIGEG SLEPSPQSQA EDGRSQAAVG AVPEGAWKDT AQLHKSEEAV S VGQKRVLR YYLFQGQRYI WIETQQAFYQ VSLLDHGRSC DDVHRSRHGL SLQDQMVRKA IYGPNVISIP VKSYPQLLVD EA LNPYYGF QAFSIALWLA DHYYWYALCI FLISSISICL SLYKTRKQSQ TLRDMVKLSM RVCVCRPGGE EEWVDSSELV PGD CLVLPQ EGGLMPCDAA LVAGECMVNE SSLTGESIPV LKTALPEGLG PYCAETHRRH TLFCGTLILQ ARAYVGPHVL AVVT RTGFC TAKGGLVSSI LHPRPINFKF YKHSMKFVAA LSVLALLGTI YSIFILYRNR VPLNEIVIRA LDLVTVVVPP ALPAA MTVC TLYAQSRLRR QGIFCIHPLR INLGGKLQLV CFDKTGTLTE DGLDVMGVVP LKGQAFLPLV PEPRRLPVGP LLRALA TCH ALSRLQDTPV GDPMDLKMVE STGWVLEEEP AADSAFGTQV LAVMRPPLWE PQLQAMEEPP VPVSVLHRFP FSSALQR MS VVVAWPGATQ PEAYVKGSPE LVAGLCNPET VPTDFAQMLQ SYTAAGYRVV ALASKPLPTV PSLEAAQQLT RDTVEGDL S LLGLLVMRNL LKPQTTPVIQ ALRRTRIRAV MVTGDNLQTA VTVARGCGMV APQEHLIIVH ATHPERGQPA SLEFLPMES PTAVNGVKDP DQAASYTVEP DPRSRHLALS GPTFGIIVKH FPKLLPKVLV QGTVFARMAP EQKTELVCEL QKLQYCVGMC GDGANDCGA LKAADVGISL SQAEASVVSP FTSSMASIEC VPMVIREGRC SLDTSFSVFK YMALYSLTQF ISVLILYTIN T NLGDLQFL AIDLVITTTV AVLMSRTGPA LVLGRVRPPG ALLSVPVLSS LLLQMVLVTG VQLGGYFLTL AQPWFVPLNR TV AAPDNLP NYENTVVFSL SSFQYLILAA AVSKGAPFRR PLYTNVPFLV ALALLSSVLV GLVLVPGLLQ GPLALRNITD TGF KLLLLG LVTLNFVGAF MLESVLDQCL PACLRRLRPK RASKKRFKQL ERELAEQPWP PLPAGPLR UniProtKB: Polyamine-transporting ATPase 13A2 |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: BERYLLIUM TRIFLUORIDE ION
| 分子 | 名称: BERYLLIUM TRIFLUORIDE ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: BEF |
|---|---|
| 分子量 | 理論値: 66.007 Da |
| Chemical component information |  ChemComp-BEF: |
-分子 #5: SPERMINE
| 分子 | 名称: SPERMINE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SPM |
|---|---|
| 分子量 | 理論値: 202.34 Da |
| Chemical component information |  ChemComp-SPM: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7vpk: |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)