+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30299 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Cas12f1-sgRNA-target DNA complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cas12f / Cas14 / sgRNA / target DNA / CRISPR / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 機能・相同性 | Transposase IS605, OrfB, C-terminal / Putative transposase DNA-binding domain / defense response to virus / endonuclease activity / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding / CRISPR-associated endodeoxyribonuclease Cas12f1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  uncultured archaeon (環境試料) / synthetic construct (人工物) uncultured archaeon (環境試料) / synthetic construct (人工物) | |||||||||
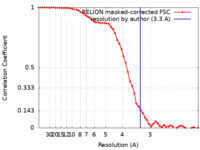
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Takeda NS / Nakagawa R | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Structure of the miniature type V-F CRISPR-Cas effector enzyme. 著者: Satoru N Takeda / Ryoya Nakagawa / Sae Okazaki / Hisato Hirano / Kan Kobayashi / Tsukasa Kusakizako / Tomohiro Nishizawa / Keitaro Yamashita / Hiroshi Nishimasu / Osamu Nureki /  要旨: RNA-guided DNA endonucleases derived from CRISPR-Cas adaptive immune systems are widely used as powerful genome-engineering tools. Among the diverse CRISPR-Cas nucleases, the type V-F Cas12f (also ...RNA-guided DNA endonucleases derived from CRISPR-Cas adaptive immune systems are widely used as powerful genome-engineering tools. Among the diverse CRISPR-Cas nucleases, the type V-F Cas12f (also known as Cas14) proteins are exceptionally compact and associate with a guide RNA to cleave single- and double-stranded DNA targets. Here, we report the cryo-electron microscopy structure of Cas12f1 (also known as Cas14a) in complex with a guide RNA and its target DNA. Unexpectedly, the structure revealed that two Cas12f1 molecules assemble with the single guide RNA to recognize the double-stranded DNA target. Each Cas12f1 protomer adopts a different conformation and plays distinct roles in nucleic acid recognition and DNA cleavage, thereby explaining how the miniature Cas12f1 enzyme achieves RNA-guided DNA cleavage as an "asymmetric homodimer." Our findings augment the mechanistic understanding of diverse CRISPR-Cas nucleases and provide a framework for the development of compact genome-engineering tools critical for therapeutic genome editing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30299.map.gz emd_30299.map.gz | 2.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30299-v30.xml emd-30299-v30.xml emd-30299.xml emd-30299.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30299_fsc.xml emd_30299_fsc.xml | 5.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30299.png emd_30299.png | 183.3 KB | ||
| マスクデータ |  emd_30299_msk_1.map emd_30299_msk_1.map | 12.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-30299.cif.gz emd-30299.cif.gz | 6.2 KB | ||
| その他 |  emd_30299_half_map_1.map.gz emd_30299_half_map_1.map.gz emd_30299_half_map_2.map.gz emd_30299_half_map_2.map.gz | 9.8 MB 9.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30299 http://ftp.pdbj.org/pub/emdb/structures/EMD-30299 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30299 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30299 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30299_validation.pdf.gz emd_30299_validation.pdf.gz | 802.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30299_full_validation.pdf.gz emd_30299_full_validation.pdf.gz | 802.2 KB | 表示 | |
| XML形式データ |  emd_30299_validation.xml.gz emd_30299_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_30299_validation.cif.gz emd_30299_validation.cif.gz | 14.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30299 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30299 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30299 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30299 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7c7lMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10585 (タイトル: Cryo-EM structure of the Cas12f1-sgRNA-target DNA complex EMPIAR-10585 (タイトル: Cryo-EM structure of the Cas12f1-sgRNA-target DNA complexData size: 649.5 Data #1: Unaligned movies for Cas12f1-sgRNA-target DNA complex [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30299.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30299.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05133 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_30299_msk_1.map emd_30299_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_30299_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_30299_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the Cas12f1-sgRNA-target DNA complex
| 全体 | 名称: Cryo-EM structure of the Cas12f1-sgRNA-target DNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the Cas12f1-sgRNA-target DNA complex
| 超分子 | 名称: Cryo-EM structure of the Cas12f1-sgRNA-target DNA complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
-超分子 #2: Cas12f1
| 超分子 | 名称: Cas12f1 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|
-超分子 #4: target DNA
| 超分子 | 名称: target DNA / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|
-超分子 #3: sgRNA
| 超分子 | 名称: sgRNA / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: CRISPR-associated protein Cas14a.1
| 分子 | 名称: CRISPR-associated protein Cas14a.1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
| 分子量 | 理論値: 62.748598 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHGS MAKNTITKTL KLRIVRPYNS AEVEKIVADE KNNREKIALE KNKDKVKEAC SKHLKVAAYC TTQVERNACL FCKARKLDD KFYQKLRGQF PDAVFWQEIS EIFRQLQKQA AEIYNQSLIE LYYEIFIKGK GIANASSVEH YLSDVCYTRA A ELFKNAAI ...文字列: MGHHHHHHGS MAKNTITKTL KLRIVRPYNS AEVEKIVADE KNNREKIALE KNKDKVKEAC SKHLKVAAYC TTQVERNACL FCKARKLDD KFYQKLRGQF PDAVFWQEIS EIFRQLQKQA AEIYNQSLIE LYYEIFIKGK GIANASSVEH YLSDVCYTRA A ELFKNAAI ASGLRSKIKS NFRLKELKNM KSGLPTTKSD NFPIPLVKQK GGQYTGFEIS NHNSDFIIKI PFGRWQVKKE ID KYRPWEK FDFEQVQKSP KPISLLLSTQ RRKRNKGWSK DEGTEAEIKK VMNGDYQTSY IEVKRGSKIG EKSAWMLNLS IDV PKIDKG VDPSIIGGIA VGVKSPLVCA INNAFSRYSI SDNDLFHFNK KMFARRRILL KKNRHKRAGH GAKNKLKPIT ILTE KSERF RKKLIERWAC EIADFFIKNK VGTVQMENLE SMKRKEDSYF NIRLRGFWPY AEMQNKIEFK LKQYGIEIRK VAPNN TSKT CSKCGHLNNY FNFEYRKKNK FPHFKCEKCN FKENADYNAA LNISNPKLKS TKEEP UniProtKB: CRISPR-associated endodeoxyribonuclease Cas12f1 |
-分子 #2: sgRNA
| 分子 | 名称: sgRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
| 分子量 | 理論値: 58.133543 KDa |
| 配列 | 文字列: UUCACUGAUA AAGUGGAGAA CCGCUUCACC AAAAGCUGUC CCUUAGGGGA UUAGAACUUG AGUGAAGGUG GGCUGCUUGC AUCAGCCUA AUGUCGAGAA GUGCUUUCUU CGGAAAGUAA CCCUCGAAAC AAAUUCAUUU GAAAGAAUGA AGGAAUGCAA C GGAAAUUA GGUGCGCUUG GC |
-分子 #3: DNA (40-mer)
| 分子 | 名称: DNA (40-mer) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.297954 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DT)(DG)(DG)(DT)(DT)(DG)(DC) (DC)(DA)(DA)(DG)(DC)(DG)(DC)(DA)(DC)(DC) (DT)(DA)(DA)(DT)(DT)(DT)(DC)(DC)(DC) (DA)(DA)(DA)(DT)(DT)(DA)(DG)(DA)(DA)(DA) (DA) |
-分子 #4: DNA (40-mer)
| 分子 | 名称: DNA (40-mer) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.323922 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DC)(DT)(DA)(DA)(DT)(DT) (DT)(DG)(DG)(DG)(DA)(DA)(DA)(DT)(DT)(DA) (DG)(DG)(DT)(DG)(DC)(DG)(DC)(DT)(DT) (DG)(DG)(DC)(DA)(DA)(DC)(DC)(DA)(DT)(DT) (DC) |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 48.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)