+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30017 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
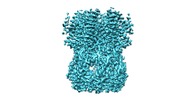
| タイトル | AtMSL1 wild type | |||||||||
 マップデータ マップデータ | EM map at 3.3 angstrom | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mechanosensitive / Ion channel / Plant / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mechanosensitive monoatomic ion channel activity / chloroplast envelope / chloroplast / cellular response to oxidative stress / mitochondrial inner membrane / mitochondrion 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Sun L | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2020 ジャーナル: Cell Rep / 年: 2020タイトル: Structural Insights into a Plant Mechanosensitive Ion Channel MSL1. 著者: Yawen Li / Yufei Hu / Jiawei Wang / Xin Liu / Wei Zhang / Linfeng Sun /  要旨: The small conductance mechanosensitive ion channel (MscS)-like (MSL) proteins in plants are evolutionarily conserved homologs of the bacterial small conductance mechanosensitive ion channels. As the ...The small conductance mechanosensitive ion channel (MscS)-like (MSL) proteins in plants are evolutionarily conserved homologs of the bacterial small conductance mechanosensitive ion channels. As the sole member of the Arabidopsis MSL family localized in the mitochondrial inner membrane, MSL1 is essential to maintain the normal membrane potential of mitochondria. Here, we report a cryoelectron microscopy (cryo-EM) structure of Arabidopsis thaliana MSL1 (AtMSL1) at 3.3 Å. The overall architecture of AtMSL1 is similar to MscS. However, the transmembrane domain of AtMSL1 is larger. Structural differences are observed in both the transmembrane and the matrix domain of AtMSL1. The carboxyl-terminus of AtMSL1 is more flexible and the β-barrel structure observed in MscS is absent. The side portals in AtMSL1 are significantly smaller, and enlarging the size of the portal by mutagenesis can increase the channel conductance. Our study provides a framework for eukaryotic MscS-like mechanosensitive ion channels and the gating mechanism of the MscS family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30017.map.gz emd_30017.map.gz | 49.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30017-v30.xml emd-30017-v30.xml emd-30017.xml emd-30017.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_30017.png emd_30017.png | 238.7 KB | ||
| Filedesc metadata |  emd-30017.cif.gz emd-30017.cif.gz | 5.3 KB | ||
| その他 |  emd_30017_half_map_1.map.gz emd_30017_half_map_1.map.gz emd_30017_half_map_2.map.gz emd_30017_half_map_2.map.gz | 40.5 MB 40.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30017 http://ftp.pdbj.org/pub/emdb/structures/EMD-30017 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30017 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30017 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30017_validation.pdf.gz emd_30017_validation.pdf.gz | 761.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30017_full_validation.pdf.gz emd_30017_full_validation.pdf.gz | 760.7 KB | 表示 | |
| XML形式データ |  emd_30017_validation.xml.gz emd_30017_validation.xml.gz | 11.6 KB | 表示 | |
| CIF形式データ |  emd_30017_validation.cif.gz emd_30017_validation.cif.gz | 13.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30017 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30017 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30017 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30017 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30017.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30017.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map at 3.3 angstrom | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half 1 map
| ファイル | emd_30017_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half 1 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half 2 map
| ファイル | emd_30017_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half 2 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Wild type of AtMSL1
| 全体 | 名称: Wild type of AtMSL1 |
|---|---|
| 要素 |
|
-超分子 #1: Wild type of AtMSL1
| 超分子 | 名称: Wild type of AtMSL1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 350 KDa |
-分子 #1: Mechanosensitive ion channel protein 1, mitochondrial
| 分子 | 名称: Mechanosensitive ion channel protein 1, mitochondrial タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.942 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAGVRLSLLK SIQRSIKPHA TAKSCSGLLN SHARAFTCGN LLDGPKASPS MISFSSNIRL HNDAKPFNYL GHSSYARAFS SKSDDFGSI VASGVTGSGD GNGNGNDWVE KAKDVLQTSV DAVTETAKKT KDVSDEMIPH VQQFLDSNPY LKDVIVPVSL T MTGTLFAW ...文字列: MAGVRLSLLK SIQRSIKPHA TAKSCSGLLN SHARAFTCGN LLDGPKASPS MISFSSNIRL HNDAKPFNYL GHSSYARAFS SKSDDFGSI VASGVTGSGD GNGNGNDWVE KAKDVLQTSV DAVTETAKKT KDVSDEMIPH VQQFLDSNPY LKDVIVPVSL T MTGTLFAW VVMPRILRRF HTYAMQSSAK LLPVGFSNED VPYEKSFWGA LEDPARYLVT FIAFAQIAAM VAPTTAIAQY FS PTVKGAV ILSLVWFLYR WKTNVITRML SAKSFGGLDR EKVLTLDKVS SVGLFAIGLM ASAEACGVAV QSILTVGGVG GVA TAFAAR DILGNVLSGL SMQFSRPFSM GDTIKAGSVE GQVIEMGLTT TSLLNAEKFP VLVPNSLFSS QVIVNKSRAQ WRAI ASKIP LQIDDLDMIP QISNEIKEML RSNTKVFLGK EAPHCYLSRV EKSFAELTIG CNLIRMGKEE LYNTQQEVLL EAVKI IKKH GVSLGTTWDN STL UniProtKB: Mechanosensitive ion channel protein 1, mitochondrial |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C7 (7回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 251528 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)