登録構造単位
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細
分子量 (理論値) 分子数 合計 (水以外) 101,111 29 ポリマ- 97,019 6 非ポリマー 4,092 23 水 2,774 154
1
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,055 12 ポリマ- 48,510 3 非ポリマー 1,546 9 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_655 -y+1,x-y,z 1 crystal symmetry operation 3_665 -x+y+1,-x+1,z 1
Buried area 4800 Å2 ΔGint -31.7 kcal/mol Surface area 23700 Å2 手法
2
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 51,412 18 ポリマ- 48,510 3 非ポリマー 2,903 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_665 -y+1,x-y+1,z 1 crystal symmetry operation 3_565 -x+y,-x+1,z 1
Buried area 4810 Å2 ΔGint -32.5 kcal/mol Surface area 24160 Å2 手法
3
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,055 12 ポリマ- 48,510 3 非ポリマー 1,546 9 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_555 -y,x-y,z 1 crystal symmetry operation 3_555 -x+y,-x,z 1
Buried area 4690 Å2 ΔGint -33.8 kcal/mol Surface area 23800 Å2 手法
4
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 51,412 18 ポリマ- 48,510 3 非ポリマー 2,903 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 3_665 -x+y+1,-x+1,z 1 crystal symmetry operation 2_655 -y+1,x-y,z 1
Buried area 4850 Å2 ΔGint -31.8 kcal/mol Surface area 24400 Å2 手法
5
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,055 12 ポリマ- 48,510 3 非ポリマー 1,546 9 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 3_765 -x+y+2,-x+1,z 1 crystal symmetry operation 2_645 -y+1,x-y-1,z 1
Buried area 4750 Å2 ΔGint -34.6 kcal/mol Surface area 23540 Å2 手法
6
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,343 15 ポリマ- 48,510 3 非ポリマー 1,834 12 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_755 -y+2,x-y,z 1 crystal symmetry operation 3_775 -x+y+2,-x+2,z 1
Buried area 4710 Å2 ΔGint -27.6 kcal/mol Surface area 24390 Å2 手法
単位格子 Length a, b, c (Å) 119.600, 119.600, 57.700 Angle α, β, γ (deg.) 90.00, 90.00, 120.00 Int Tables number 143 Space group name H-M P3
非結晶学的対称性 (NCS) NCS oper ID Code Matrix ベクター 1 given(-0.48979, -0.87184, 0.00166), (0.87182, -0.48976, 0.00878), (-0.00684, 0.00575, 0.99996)59.33665, 33.84259, 3.200312 given(-0.40844, 0.91266, -0.01534), (-0.91262, -0.40863, -0.01246), (-0.01764, 0.00891, 0.9998)-7.14714, 68.60696, 1.573663 given(-0.999, 0.054), (0.054, 0.999, 0.005), (0.005, -1)117.56602, -3.03136, 30.087574 given(0.5561, -0.83111, 0.00285), (-0.83111, -0.5561, -0.00266), (0.0038, -0.00089, -0.99999)114.95987, 68.82652, 31.151175 given(0.54413, 0.83899, 0.00501), (0.83896, -0.54404, -0.01259), (-0.00784, 0.01105, -0.99991)58.06283, 37.49632, 33.26504
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  分子置換 / 解像度: 2.3 Å
分子置換 / 解像度: 2.3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2009
ジャーナル: Acta Crystallogr.,Sect.D / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2jkj.cif.gz
2jkj.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2jkj.ent.gz
pdb2jkj.ent.gz PDB形式
PDB形式 2jkj.json.gz
2jkj.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2jkj_validation.pdf.gz
2jkj_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2jkj_full_validation.pdf.gz
2jkj_full_validation.pdf.gz 2jkj_validation.xml.gz
2jkj_validation.xml.gz 2jkj_validation.cif.gz
2jkj_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/jk/2jkj
https://data.pdbj.org/pub/pdb/validation_reports/jk/2jkj ftp://data.pdbj.org/pub/pdb/validation_reports/jk/2jkj
ftp://data.pdbj.org/pub/pdb/validation_reports/jk/2jkj リンク
リンク 集合体
集合体






 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: BRUKER AXS MICROSTAR / 波長: 1.5418
回転陽極 / タイプ: BRUKER AXS MICROSTAR / 波長: 1.5418  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













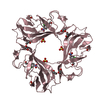


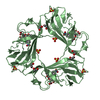



 PDBj
PDBj