+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of E.coli transcription elongation complex | ||||||||||||||||||
 マップデータ マップデータ | W6_refine122 | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Transcription elongation / TRANSCRIPTION-DNA-RNA complex | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / bacterial-type RNA polymerase core enzyme binding / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex ...RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / bacterial-type RNA polymerase core enzyme binding / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation / DNA-templated transcription initiation / transcription antitermination / cell motility / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis / protein dimerization activity / response to antibiotic / magnesium ion binding / DNA binding / zinc ion binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.42 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Duan W / Serganov A | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Control of transcription elongation and DNA repair by alarmone ppGpp. 著者: Jacob W Weaver / Sergey Proshkin / Wenqian Duan / Vitaly Epshtein / Manjunath Gowder / Binod K Bharati / Elena Afanaseva / Alexander Mironov / Alexander Serganov / Evgeny Nudler /   要旨: Second messenger (p)ppGpp (collectively guanosine tetraphosphate and guanosine pentaphosphate) mediates bacterial adaptation to nutritional stress by modulating transcription initiation. More ...Second messenger (p)ppGpp (collectively guanosine tetraphosphate and guanosine pentaphosphate) mediates bacterial adaptation to nutritional stress by modulating transcription initiation. More recently, ppGpp has been implicated in coupling transcription and DNA repair; however, the mechanism of ppGpp engagement remained elusive. Here we present structural, biochemical and genetic evidence that ppGpp controls Escherichia coli RNA polymerase (RNAP) during elongation via a specific site that is nonfunctional during initiation. Structure-guided mutagenesis renders the elongation (but not initiation) complex unresponsive to ppGpp and increases bacterial sensitivity to genotoxic agents and ultraviolet radiation. Thus, ppGpp binds RNAP at sites with distinct functions in initiation and elongation, with the latter being important for promoting DNA repair. Our data provide insights on the molecular mechanism of ppGpp-mediated adaptation during stress, and further highlight the intricate relationships between genome stability, stress responses and transcription. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29491.map.gz emd_29491.map.gz | 230.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29491-v30.xml emd-29491-v30.xml emd-29491.xml emd-29491.xml | 28.4 KB 28.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29491.png emd_29491.png | 51.7 KB | ||
| Filedesc metadata |  emd-29491.cif.gz emd-29491.cif.gz | 9 KB | ||
| その他 |  emd_29491_half_map_1.map.gz emd_29491_half_map_1.map.gz emd_29491_half_map_2.map.gz emd_29491_half_map_2.map.gz | 225.9 MB 225.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29491 http://ftp.pdbj.org/pub/emdb/structures/EMD-29491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29491 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29491_validation.pdf.gz emd_29491_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29491_full_validation.pdf.gz emd_29491_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_29491_validation.xml.gz emd_29491_validation.xml.gz | 15.4 KB | 表示 | |
| CIF形式データ |  emd_29491_validation.cif.gz emd_29491_validation.cif.gz | 18.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29491 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29491 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29491 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29491 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fvrMC  8fvwC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29491.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29491.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | W6_refine122 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_29491_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
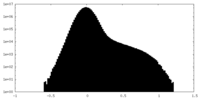
| 投影像・断面図 |
| ||||||||||||
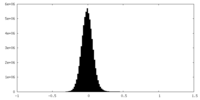
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29491_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
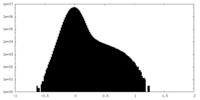
| 投影像・断面図 |
| ||||||||||||
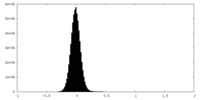
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : E. coli transcription elongation complex (EC)
+超分子 #1: E. coli transcription elongation complex (EC)
+分子 #1: DNA-directed RNA polymerase subunit alpha
+分子 #2: DNA-directed RNA polymerase subunit beta
+分子 #3: DNA-directed RNA polymerase subunit beta'
+分子 #4: DNA-directed RNA polymerase subunit omega
+分子 #5: DNA (53-MER)
+分子 #7: DNA (53-MER)
+分子 #6: RNA (16-MER)
+分子 #8: ZINC ION
+分子 #9: MAGNESIUM ION
+分子 #10: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES, pH 8.0, 150 mM NaCl, 10 mM MgCl2, 10 uM ZnCl2, 5 mM DTT |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 260.0 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 79.95 K / 最高: 79.95 K |
| アライメント法 | Coma free - Residual tilt: 0.05 mrad |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 3298 / 平均露光時間: 2.0 sec. / 平均電子線量: 59.07 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8fvr: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)